使用适配器
简介
您可以定义适配器来在优化过程中实现动态变异和交叉概率,而非固定值。其理念是将这些概率设为世代数的函数;这种定义可以实现不同的训练策略,例如:
开始时采用较高的变异概率以探索更多样化的解决方案,然后逐步降低变异概率,专注于保留更有潜力的解。
初始使用较低交叉概率,最终采用较高概率
为每个参数组合不同的策略
所有方法都使用三个参数:
initial_value: 这是在第0代时使用的初始值
end_value: 这是参数从initial_value开始可以取到的极限值
adaptive_rate: 控制数值接近end_value的速度;数值越大收敛速度越快
在接下来的章节中,理解以下符号表示非常重要:
名称 |
符号 |
|---|---|
初始值 |
\(p_0\) |
终止值 |
\(p_f\) |
当前世代 |
\(t\) |
自适应率 |
\(\alpha\) |
第t代的值 |
\(p(t; \alpha)\) |
请注意 \(p_0\) 不需要大于 \(p_f\)。
如果 \(p_0 > p_f\),则表示您正在向 \(p_f\) 进行衰减。
如果 \(p_0 < p_f\),则表示您正在向 \(p_f\) 进行上升操作。
所有非常数适配器 \(p(t; \alpha)\),当 \(\alpha \in (0,1)\) 时, 具有以下特性:
以下适配器可用:
常量适配器
指数适配器
反向适配器
潜在适配器
ConstantAdapter
该适配器旨在供包内部使用;当用户未创建适配器,而是将交叉或变异概率定义为实数时,包会将其转换为ConstantAdapter,以便库能在两种情况下使用相同方法调用内部API。因此,其定义为:
ExponentialAdapter
指数适配器使用以下形式来改变初始值
使用示例:
from sklearn_genetic.schedules import ExponentialAdapter
# Decay over initial_value
adapter = ExponentialAdapter(initial_value=0.8, end_value=0.2, adaptive_rate=0.1)
# Run a few iterations
for _ in range(3):
adapter.step() # 0.8, 0.74, 0.69
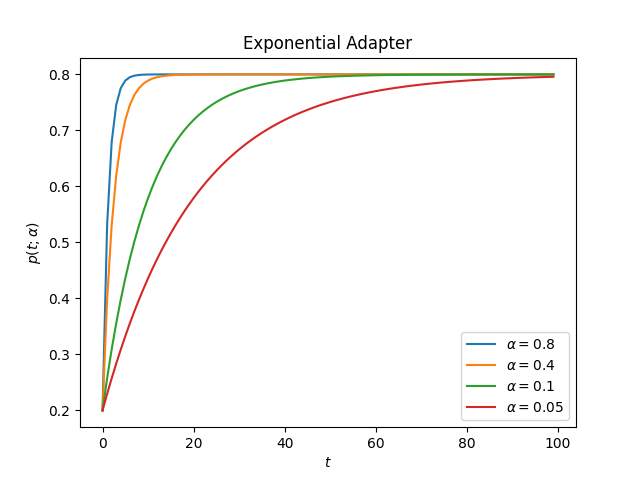
这是适配器在不同alpha值下的表现
衰减:
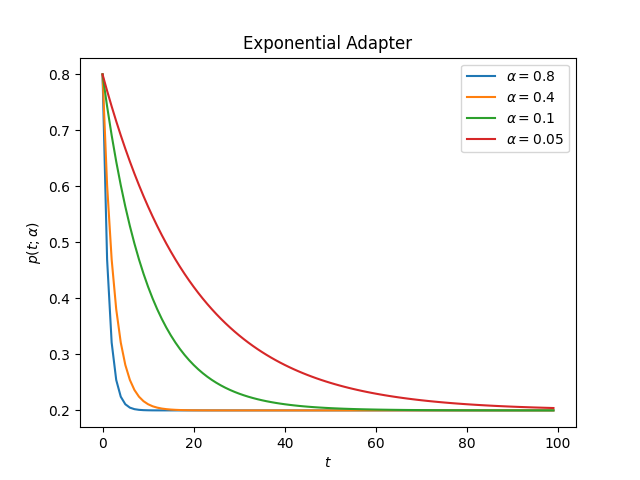
升序:
import matplotlib.pyplot as plt
from sklearn_genetic.schedules import ExponentialAdapter
values = [{"initial_value": 0.8, "end_value": 0.2}, # Decay
{"initial_value": 0.2, "end_value": 0.8}] # Ascend
alphas = [0.8, 0.4, 0.1, 0.05]
for value in values:
for alpha in alphas:
adapter = ExponentialAdapter(**value, adaptive_rate=alpha)
adapter_result = [adapter.step() for _ in range(100)]
plt.plot(adapter_result, label=r'$\alpha={}$'.format(alpha))
plt.xlabel(r'$t$')
plt.ylabel(r'$p(t; \alpha)$')
plt.title("Exponential Adapter")
plt.legend()
plt.show()
InverseAdapter
逆向适配器采用以下形式来改变初始值
使用示例:
from sklearn_genetic.schedules import InverseAdapter
# Decay over initial_value
adapter = InverseAdapter(initial_value=0.8, end_value=0.2, adaptive_rate=0.1)
# Run a few iterations
for _ in range(3):
adapter.step() # 0.8, 0.75, 0.7
这是适配器在不同alpha值下的表现
衰减:
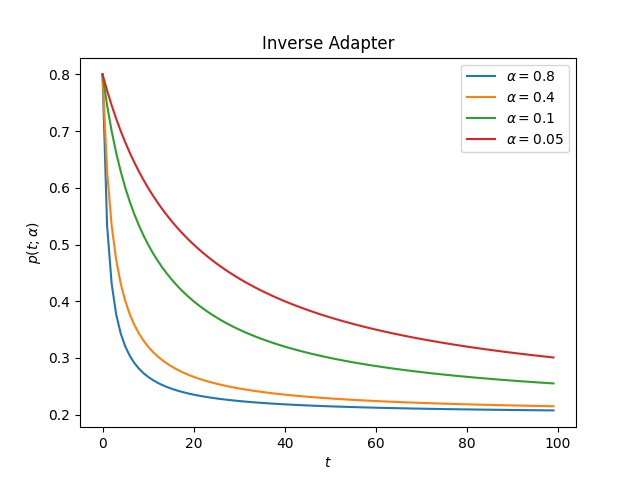
升序:
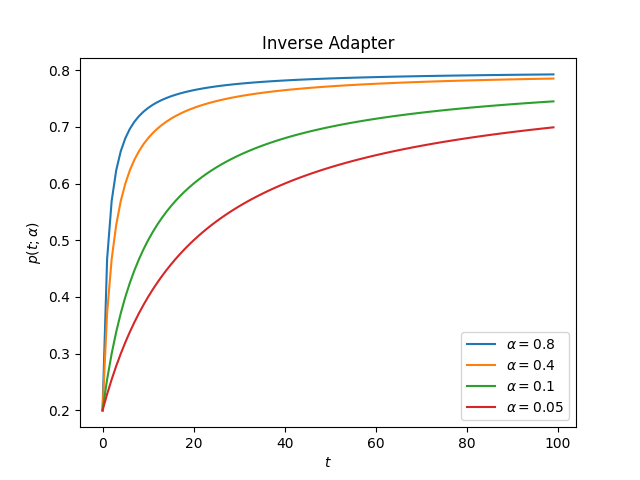
PotentialAdapter
逆向适配器采用以下形式来改变初始值
使用示例:
from sklearn_genetic.schedules import PotentialAdapter
# Decay over initial_value
adapter = PotentialAdapter(initial_value=0.8, end_value=0.2, adaptive_rate=0.1)
# Run a few iterations
for _ in range(3):
adapter.step() # 0.8, 0.26, 0.206
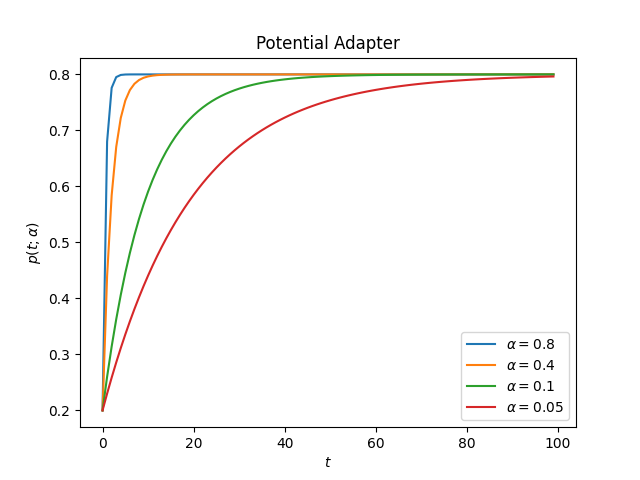
这是适配器在不同alpha值下的表现
衰减:
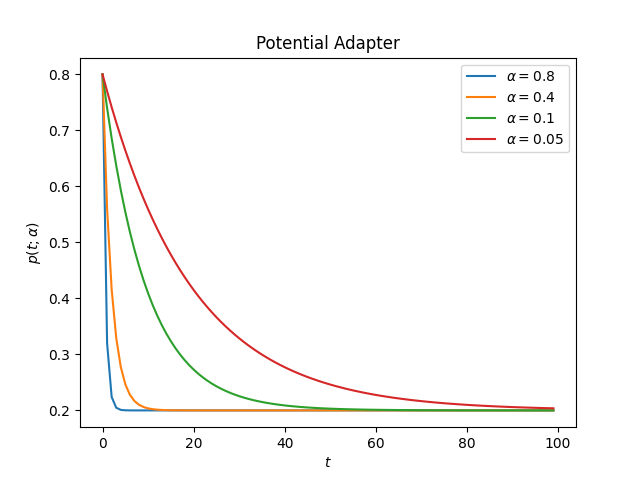
升序:
对比
这是所有适配器在相同alpha值下的外观
衰减:
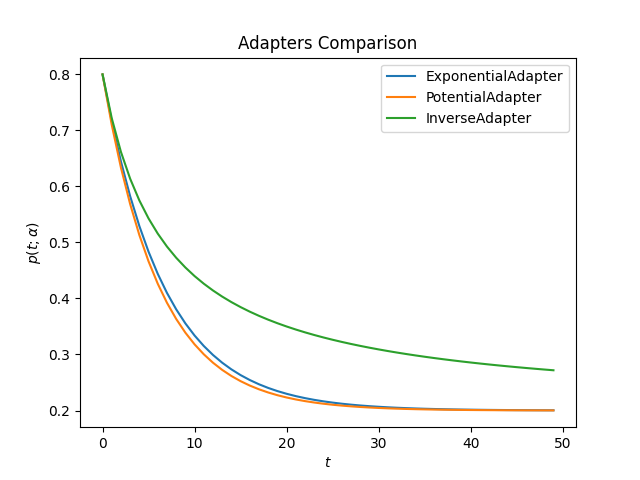
升序:

import matplotlib.pyplot as plt
from sklearn_genetic.schedules import ExponentialAdapter, PotentialAdapter, InverseAdapter
params = {"initial_value": 0.2, "end_value": 0.8, "adaptive_rate": 0.15} # Ascend
adapters = [ExponentialAdapter(**params), PotentialAdapter(**params), InverseAdapter(**params)]
for adapter in adapters:
adapter_result = [adapter.step() for _ in range(50)]
plt.plot(adapter_result, label=f"{type(adapter).__name__}")
plt.xlabel(r'$t$')
plt.ylabel(r'$p(t; \alpha)$')
plt.title("Adapters Comparison")
plt.legend()
plt.show()
完整示例
在本示例中,我们希望为变异概率创建一个衰减策略,并为交叉概率创建一个递增策略,分别称之为\(p_{mt}(t; \alpha)\)和\(p_{cr}(t; \alpha)\);这将使优化器在初始迭代中探索更多样化的解决方案。需要注意的是,在此场景下,我们必须谨慎选择\(\alpha, p_0, p_f\),这是因为进化实现要求:
同样的思路可以应用于超参数调优或特征选择。
from sklearn_genetic import GASearchCV
from sklearn_genetic import ExponentialAdapter
from sklearn_genetic.space import Continuous, Categorical, Integer
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import train_test_split, StratifiedKFold
from sklearn.datasets import load_digits
from sklearn.metrics import accuracy_score
data = load_digits()
n_samples = len(data.images)
X = data.images.reshape((n_samples, -1))
y = data['target']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=42)
clf = RandomForestClassifier()
mutation_adapter = ExponentialAdapter(initial_value=0.8, end_value=0.2, adaptive_rate=0.1)
crossover_adapter = ExponentialAdapter(initial_value=0.2, end_value=0.8, adaptive_rate=0.1)
param_grid = {'min_weight_fraction_leaf': Continuous(0.01, 0.5, distribution='log-uniform'),
'bootstrap': Categorical([True, False]),
'max_depth': Integer(2, 30),
'max_leaf_nodes': Integer(2, 35),
'n_estimators': Integer(100, 300)}
cv = StratifiedKFold(n_splits=3, shuffle=True)
evolved_estimator = GASearchCV(estimator=clf,
cv=cv,
scoring='accuracy',
population_size=20,
generations=25,
mutation_probability=mutation_adapter,
crossover_probability=crossover_adapter,
param_grid=param_grid,
n_jobs=-1)
# Train and optimize the estimator
evolved_estimator.fit(X_train, y_train)
# Best parameters found
print(evolved_estimator.best_params_)
# Use the model fitted with the best parameters
y_predict_ga = evolved_estimator.predict(X_test)
print(accuracy_score(y_test, y_predict_ga))
# Saved metadata for further analysis
print("Stats achieved in each generation: ", evolved_estimator.history)
print("Best k solutions: ", evolved_estimator.hof)