如何使用Sklearn-genetic-opt
简介
Sklearn-genetic-opt利用进化算法来微调scikit-learn机器学习算法并执行特征选择。 它设计用于接受scikit-learn回归或分类模型(或包含其中一个模型的管道)。
这个包的核心理念是定义我们需要调优的超参数集,以及这些参数可取值范围的上下界。 我们可以自定义不同的优化算法、回调函数和内置参数来控制优化过程。 入门阶段,我们将仅使用最基本的功能和选项。
优化过程通过进化算法实现,并借助deap包完成。 其工作原理是:首先定义需要调优的超参数集合,初始时随机采样一组选项(种群)。 然后通过交配、变异、选择和评估等进化算子,在每一代中生成新的候选方案以提升交叉验证分数。 该过程将持续进行,直到达到指定迭代次数或满足回调函数设定的终止条件。
调优示例
首先让我们导入一些数据集和其他scikit-learn标准模块,我们将使用 digits数据集。 这是一个分类问题,我们将针对这个任务微调随机森林分类器。
import matplotlib.pyplot as plt
from sklearn_genetic import GASearchCV
from sklearn_genetic.space import Categorical, Integer, Continuous
from sklearn.model_selection import train_test_split, StratifiedKFold
from sklearn.neural_network import MLPClassifier
from sklearn.datasets import load_digits
from sklearn.metrics import accuracy_score
首先我们读取数据,将其划分为训练集和测试集,并可视化部分数据点:
data = load_digits()
n_samples = len(data.images)
X = data.images.reshape((n_samples, -1))
y = data['target']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=42)
_, axes = plt.subplots(nrows=1, ncols=4, figsize=(10, 3))
for ax, image, label in zip(axes, data.images, data.target):
ax.set_axis_off()
ax.imshow(image, cmap=plt.cm.gray_r, interpolation='nearest')
ax.set_title('Training: %i' % label)
我们应该会看到类似这样的内容:

现在,我们必须定义我们的param_grid,类似于scikit-learn,它是一个包含模型超参数的字典。 与例如sckit-learn的GridSearchCv的主要区别在于, 我们不需要预先定义搜索中要使用的值, 而是定义每个参数的边界范围。
因此如果我们有一个名为'n_estimators'的参数,我们只需要告诉sckit-learn-genetic-opt这是一个整数值, 并且我们希望设置下限为100,上限为500,这样优化器就会在这个范围内设定一个值。 我们需要对所有想要调优的超参数都进行这样的设置,如下所示:
# this library even performs well on weird ranges
# adjust this to better ranges for better results
param_grid = {'tol': Continuous(1e-2, 1e10, distribution='log-uniform'),
'alpha': Continuous(1e-5, 2e-5),
'activation': Categorical(['logistic', 'tanh']),
'batch_size': Integer(300, 350)
}
请注意,对于'boostrap'这个分类变量,我们必须明确定义其所有可能的取值。 同样,在'min_weight_fraction_leaf'参数中,我们使用了一个名为distribution的附加参数, 这有助于告知优化器在优化过程中可以从哪些数据分布中采样随机值。
现在,我们已经准备好设置GASearchCV,这个对象将允许我们使用进化算法运行拟合过程。 它有几个可用的选项,在第一个示例中,我们将保持非常简单:
# The base classifier to tune
clf = MLPClassifier(hidden_layer_sizes=(50, 30))
# Our cross-validation strategy (it could be just an int)
cv = StratifiedKFold(n_splits=3, shuffle=True)
# The main class from sklearn-genetic-opt
evolved_estimator = GASearchCV(estimator=clf,
cv=cv,
scoring='accuracy',
param_grid=param_grid,
n_jobs=-1,
verbose=True,
population_size=10,
generations=20)
现在设置已经完成,需要注意的是GASearchCV中还可以指定其他参数。我们使用的这些参数与scikit-learn中的含义相同,除了已经解释过的参数外,值得一提的是"metric"将作为优化变量使用,因此算法会尝试找到能最大化该指标的最佳参数组合。
我们已准备好运行优化程序:
# Train and optimize the estimator
evolved_estimator.fit(X_train, y_train)
在训练过程中,您应该会看到类似这样的日志:
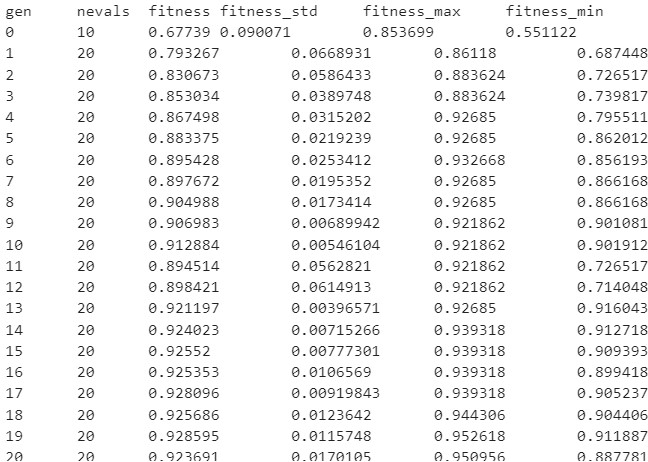
此日志展示了我们在每次迭代(代)中获得的指标,以下是每个条目的含义:
gen: 代数编号
nevals: 当前世代中有多少个个体
fitness: 交叉验证中的平均得分指标(验证集)。 在本例中,指所有超参数组合在交叉验证折叠中的平均准确率。
fitness_std: 交叉验证准确率的标准差。
fitness_max: 本代所有模型中的最高个体得分。
fitness_min: 本代所有模型中的最低个体得分。
在模型拟合完成后,我们还有一些额外的方法可以立即使用该模型。 默认情况下,它将基于交叉验证分数使用找到的最佳超参数组合:
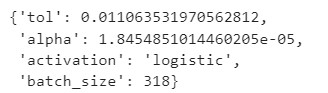
# Best parameters found
print(evolved_estimator.best_params_)
# Use the model fitted with the best parameters
y_predict_ga = evolved_estimator.predict(X_test)
print(accuracy_score(y_test, y_predict_ga))
在本例中,我们在测试集上获得了0.96的准确率分数

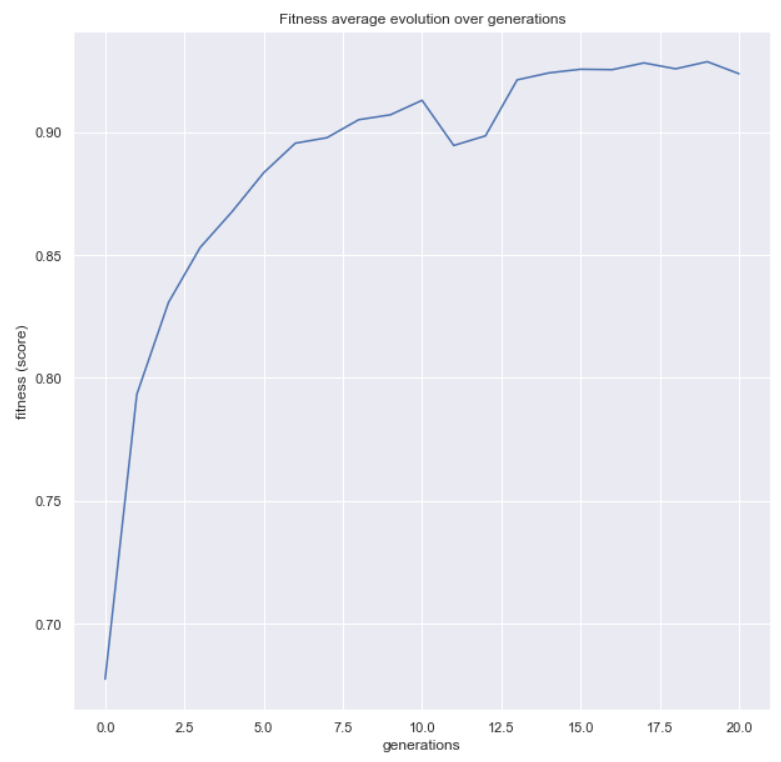
现在,让我们使用该包中提供的更多函数。 第一个函数将帮助我们查看指标在代际间的演变情况
from sklearn_genetic.plots import plot_fitness_evolution
plot_fitness_evolution(evolved_estimator)
plt.show()
最后,我们可以检查名为evolved_estimator.logbook的属性,
这是一个DEAP的日志记录本,存储了每个个体拟合模型的所有结果。
sklearn-genetic-opt自带了一个绘图函数来分析这个日志:
from sklearn_genetic.plots import plot_search_space
plot_search_space(evolved_estimator, features=['tol', 'batch_size', 'alpha'])
plt.show()
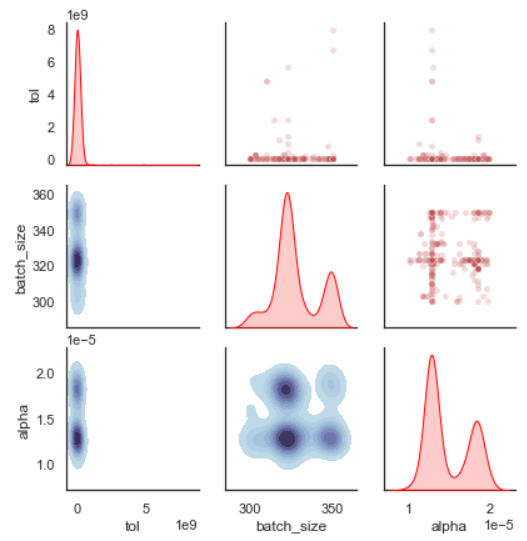
这张图向我们展示了每个超参数的采样值分布情况。 例如我们可以看到在'tol'参数上,算法主要采样了非常小的值 (这个范围需要在第二次迭代时进行调整)。您还可以查看每个变量组合 以及代表采样值的等高线图。
特征选择示例
在本示例中,我们将使用著名的鸢尾花数据集,这是一个包含四个特征的分类问题。 我们还将模拟一些随机噪声来表示不重要的特征:
import matplotlib.pyplot as plt
from sklearn_genetic import GAFeatureSelectionCV
from sklearn_genetic.plots import plot_fitness_evolution
from sklearn.model_selection import train_test_split, StratifiedKFold
from sklearn.svm import SVC
from sklearn.datasets import load_iris
from sklearn.metrics import accuracy_score
import numpy as np
data = load_iris()
X, y = data["data"], data["target"]
noise = np.random.uniform(0, 10, size=(X.shape[0], 10))
X = np.hstack((X, noise))
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=0)
这将为我们的训练集和测试集额外添加10个带噪声的特征。
现在我们可以创建GAFeatureSelectionCV对象,它与GASearchCV非常相似,并且共享大部分参数。主要区别在于GAFeatureSelectionCV不运行超参数优化,因此param_grid参数不可用,而且评估器应该使用其超参数来定义。
特征选择的方式是通过创建包含特征子集的模型并评估其交叉验证分数,子集的生成利用了现有的进化算法。该方法同时会尝试最小化所选特征的数量,因此属于多目标优化问题。
让我们创建特征选择对象,我们将使用的估计器是SVM:
clf = SVC(gamma='auto')
evolved_estimator = GAFeatureSelectionCV(
estimator=clf,
cv=3,
scoring="accuracy",
population_size=30,
generations=20,
n_jobs=-1,
verbose=True,
keep_top_k=2,
elitism=True,
)
我们已准备好运行优化程序:
# Train and select the features
evolved_estimator.fit(X_train, y_train)
在训练过程中,日志格式与之前显示的相同:
在拟合模型之后,我们还有一些额外的方法可以立即使用该模型。默认情况下,它将使用找到的最佳特征集,请记住由于算法仅使用了特征子集,当您使用predict、predict_proba等方法时,这些方法只会使用support_特征。
features = evolved_estimator.support_
# Predict only with the subset of selected features
y_predict_ga = evolved_estimator.predict(X_test)
accuracy = accuracy_score(y_test, y_predict_ga)
在这种情况下,我们在测试集上获得了0.98的准确率分数。
请注意support_是一个布尔值向量,每个位置代表特征(列)的索引,其值表示该特征是否被算法选中(True表示选中,False表示未选中)。在本例中,算法丢弃了我们创建的所有噪声随机变量,并选择了原始变量。
我们还可以绘制适应度进化曲线:
from sklearn_genetic.plots import plot_fitness_evolution
plot_fitness_evolution(evolved_estimator)
plt.show()
以上就是我们对sklearn-genetic-opt基础用法的介绍。 后续教程将涵盖GASearchCV和GAFeatureSelectionCV参数、回调函数、 不同优化算法以及更高级的使用场景。