更多示例和配方¶
本节将通过一些示例和配方来帮助您使用lifelines。
工作示例¶
如果你在寻找一些lifelines的完整示例,这里有完整的Jupyter笔记本和脚本,以及在开发博客上的示例和想法。
统计比较两个群体¶
研究人员经常希望比较不同人群的生存率。以下是一些实现这一目标的技术:
Logrank 检验¶
注意
当比例风险假设成立时,对数秩检验具有最大的功效。因此,如果生存函数交叉,对数秩检验将给出不准确的差异评估。
lifelines.statistics.logrank_test() 函数比较两个群体的“死亡”生成过程是否相等:
from lifelines.statistics import logrank_test
from lifelines.datasets import load_waltons
df = load_waltons()
ix = df['group'] == 'miR-137'
T_exp, E_exp = df.loc[ix, 'T'], df.loc[ix, 'E']
T_con, E_con = df.loc[~ix, 'T'], df.loc[~ix, 'E']
results = logrank_test(T_exp, T_con, event_observed_A=E_exp, event_observed_B=E_con)
results.print_summary()
"""
t_0 = -1
alpha = 0.95
null_distribution = chi squared
df = 1
use_bonferroni = True
---
test_statistic p
3.528 0.00034 **
"""
print(results.p_value) # 0.46759
print(results.test_statistic) # 0.528
如果你有两个以上的群体,你可以使用pairwise_logrank_test()(它以与上述相同的方式比较每一对),或者multivariate_logrank_test()(它测试所有群体具有相同的“死亡”生成过程的假设)。
import pandas as pd
from lifelines.statistics import multivariate_logrank_test
df = pd.DataFrame({
'durations': [5, 3, 9, 8, 7, 4, 4, 3, 2, 5, 6, 7],
'groups': [0, 0, 0, 0, 1, 1, 1, 1, 1, 2, 2, 2], # could be strings too
'events': [1, 1, 1, 1, 1, 1, 0, 0, 1, 1, 1, 0],
})
results = multivariate_logrank_test(df['durations'], df['groups'], df['events'])
results.print_summary()
"""
t_0 = -1
alpha = 0.95
null_distribution = chi squared
df = 2
---
test_statistic p
1.0800 0.5827
---
"""
对数秩检验统计量是根据在零假设下,所有组共享相同的生存曲线,从观察到的死亡人数与预期死亡人数之间的差异计算得出的,该差异在所有有序死亡时间上求和。因此,它在每个死亡时间上对生存曲线之间的差异进行同等加权,当比例风险假设成立时,具有最大的功效。为了测试组间生存的早期或晚期差异,对非比例风险更敏感的加权对数秩检验可能是更好的选择。
目前在lifelines中通过weightings参数提供了四种类型的加权对数秩检验:
Wilcoxon(weightings='wilcoxon')、Tarone-Ware(weightings='tarone-ware')、Peto(weightings='peto')
和Fleming-Harrington(weightings='fleming-harrington')检验。
以下权重应用于第i个有序失效时间\(t_{i}\):
\[\text{Wilcoxon:}\quad n_i\]\[\text{Tarone-Ware:}\quad \sqrt{n_i}\]\[\text{Peto:}\quad \bar{S}(t_i)\]\[\text{Fleming-Harrington}\quad \hat{S}(t_i)^p \times (1 - \hat{S}(t_i))^q\]
其中 \(n_i\) 是在时间 \(t_{i}\) 之前的风险数量,\(\bar{S}(t_i)\) 是 Peto-Peto 的修正生存估计,\(\hat{S}(t_i)\) 是在时间 \(t_{i}\) 的左连续 Kaplan-Meier 生存估计。
Wilcoxon、Tarone-Ware和Peto检验对早期死亡时间赋予更多权重。当许多观察结果被截尾时,Peto检验比Wilcoxon或Tarone-Ware检验更稳健。当p > q时,Fleming-Harrington检验对早期死亡时间赋予更多权重,而当p < q时,它对晚期差异更敏感(当p=q=0时,它简化为未加权的对数秩检验)。选择执行哪种检验应提前决定,而不是事后决定,以避免引入偏差。
import pandas as pd
from lifelines.statistics import multivariate_logrank_test
df = pd.DataFrame({
'durations': [5, 3, 9, 8, 7, 4, 4, 3, 2, 5, 6, 7],
'groups': [0, 0, 0, 0, 1, 1, 1, 1, 1, 2, 2, 2], # could be strings too
'events': [1, 1, 1, 1, 1, 1, 0, 0, 1, 1, 1, 0],
})
results = multivariate_logrank_test(df['durations'], df['groups'], df['events'], weightings='peto')
results.print_summary()
"""
t_0 = -1
null_distribution = chi squared
degrees_of_freedom = 2
test_name = multivariate_Peto_test
---
test_statistic p -log2(p)
0.95 0.62 0.68
"""
时间点上的生存差异¶
通常分析师希望比较特定时间点的群体生存率,而不是比较整个生存曲线。例如,分析师可能对5年生存率感兴趣。在特定时间点统计比较原始的Kaplan-Meier点实际上会降低统计效力。通过对Kaplan-Meier曲线进行转换,我们可以恢复更多的统计效力。函数lifelines.statistics.survival_difference_at_fixed_point_in_time_test()隐式使用log(-log)转换,并使用卡方检验比较特定时间点的群体生存率。
from lifelines.statistics import survival_difference_at_fixed_point_in_time_test
from lifelines.datasets import load_waltons
df = load_waltons()
ix = df['group'] == 'miR-137'
T_exp, E_exp = df.loc[ix, 'T'], df.loc[ix, 'E']
T_con, E_con = df.loc[~ix, 'T'], df.loc[~ix, 'E']
kmf_exp = KaplanMeierFitter(label="exp").fit(T_exp, E_exp)
kmf_con = KaplanMeierFitter(label="con").fit(T_con, E_con)
point_in_time = 10.
results = survival_difference_at_fixed_point_in_time_test(point_in_time, kmf_exp, kmf_con)
results.print_summary()
"""
t_0 = -1
null_distribution = chi squared
degrees_of_freedom = 1
point_in_time = 10.0
test_name = survival_difference_at_fixed_point_in_time_test
---
test_statistic p -log2(p)
4.77 0.03 5.11
"""
此外,我们可以在固定时间点绘制两条生存曲线并进行比较:
kmf_exp.plot_survival_function(point_in_time=point_in_time)
kmf_con.plot_survival_function(point_in_time=point_in_time)

我们可以看到,实验组的生存函数值(蓝色)低于对照组的生存函数值(橙色)。 值得注意的是,在那个特定的时间点,两组的置信区间在一定程度上重叠,这在其他时间点并不总是观察到。
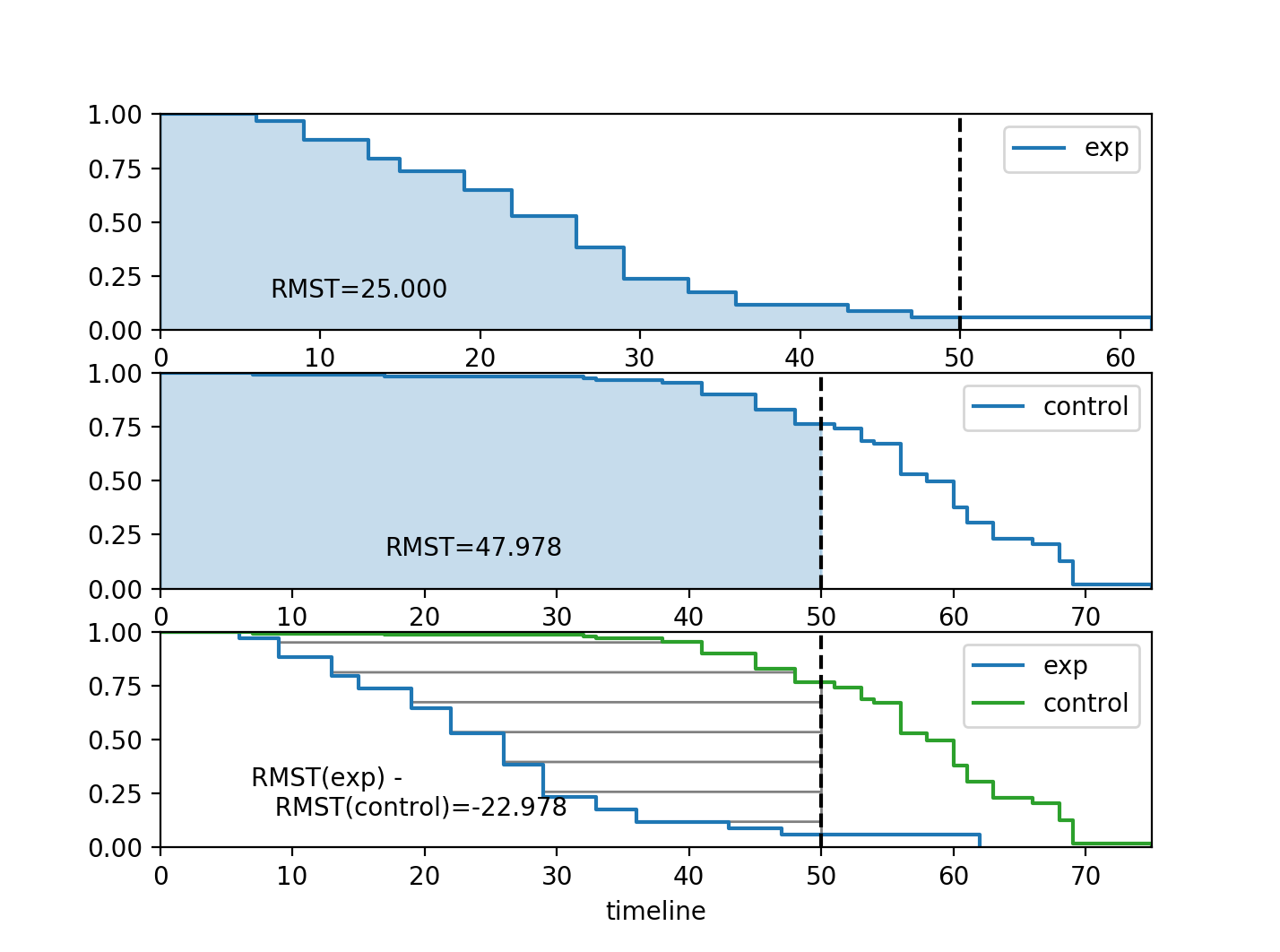
受限平均生存时间 (RMST)¶
lifelines 有一个函数可以准确计算受限平均生存时间,定义为
\[\text{RMST}(t) = \int_0^t S(\tau) d\tau\]
这是一个用于比较两条生存曲线的良好指标,因为它们的差异代表了曲线之间的区域(见下图),这是“时间损失”的度量。上述积分的上限通常是有限的,因为估计的生存曲线的尾部具有高方差,并且可能强烈影响积分。
from lifelines.utils import restricted_mean_survival_time
from lifelines.datasets import load_waltons
from lifelines import KaplanMeierFitter
df = load_waltons()
ix = df['group'] == 'miR-137'
T, E = df['T'], df['E']
time_limit = 50
kmf_exp = KaplanMeierFitter().fit(T[ix], E[ix], label='exp')
rmst_exp = restricted_mean_survival_time(kmf_exp, t=time_limit)
kmf_con = KaplanMeierFitter().fit(T[~ix], E[~ix], label='control')
rmst_con = restricted_mean_survival_time(kmf_con, t=time_limit)
此外,存在绘制RMST的绘图函数:
from matplotlib import pyplot as plt
from lifelines.plotting import rmst_plot
ax = plt.subplot(311)
rmst_plot(kmf_exp, t=time_limit, ax=ax)
ax = plt.subplot(312)
rmst_plot(kmf_con, t=time_limit, ax=ax)
ax = plt.subplot(313)
rmst_plot(kmf_exp, model2=kmf_con, t=time_limit, ax=ax)
使用lifelines进行模型选择¶
如果使用lifelines进行预测工作,理想情况下你应该执行某种类型的交叉验证方案。这种交叉验证可以让你确信你的样本外预测在实际中会表现良好。它还允许你在多个模型之间进行选择。
lifelines 有一个内置的 k 折交叉验证函数。例如,考虑以下示例:
import numpy as np
from lifelines import AalenAdditiveFitter, CoxPHFitter
from lifelines.datasets import load_regression_dataset
from lifelines.utils import k_fold_cross_validation
df = load_regression_dataset()
#create the three models we'd like to compare.
aaf_1 = AalenAdditiveFitter(coef_penalizer=0.5)
aaf_2 = AalenAdditiveFitter(coef_penalizer=10)
cph = CoxPHFitter()
print(np.mean(k_fold_cross_validation(cph, df, duration_col='T', event_col='E', scoring_method="concordance_index")))
print(np.mean(k_fold_cross_validation(aaf_1, df, duration_col='T', event_col='E', scoring_method="concordance_index")))
print(np.mean(k_fold_cross_validation(aaf_2, df, duration_col='T', event_col='E', scoring_method="concordance_index")))
从这些结果来看,带有惩罚因子10的Aalen加法模型是预测未来生存时间的最佳模型。
lifelines 也有包装器可以使用 scikit-learn 的交叉验证和网格搜索工具。参见 如何将 lifelines 与 scikit learn 结合使用。
使用QQ图选择参数模型¶
QQ图通常通过排序值来构建。然而,当存在截尾数据时,这不合适。在lifelines中,有例程仍然可以创建带有截尾数据的QQ图。这些功能在lifelines.plotting.qq_plots()下可用,并接受拟合的参数化lifelines模型。
from lifelines import *
from lifelines.plotting import qq_plot
# generate some fake log-normal data
N = 1000
T_actual = np.exp(np.random.randn(N))
C = np.exp(np.random.randn(N))
E = T_actual < C
T = np.minimum(T_actual, C)
fig, axes = plt.subplots(2, 2, figsize=(8, 6))
axes = axes.reshape(4,)
for i, model in enumerate([WeibullFitter(), LogNormalFitter(), LogLogisticFitter(), ExponentialFitter()]):
model.fit(T, E)
qq_plot(model, ax=axes[i])

此图形测试可用于使模型无效。例如,在上图中,我们可以看到只有对数正态参数模型是合适的(我们期望尾部有偏差,但不要太多)。另一个用例是选择正确的参数AFT模型。
qq_plots() 也适用于左截断的情况。
使用AIC选择参数模型¶
比较不同模型的一种自然方法是AIC:
其中 \(k\) 是模型的参数数量(自由度),\(\text{ll}\) 是最大对数似然。具有最低AIC的模型是可取的,因为这是在尽可能少的参数下最大化对数似然之间的权衡。
所有生命线模型在拟合后都具有AIC_属性。
此外,lifelines 有一个内置函数来自动化单变量参数模型之间的AIC比较:
from lifelines.utils import find_best_parametric_model
from lifelines.datasets import load_lymph_node
T = load_lymph_node()['rectime']
E = load_lymph_node()['censrec']
best_model, best_aic_ = find_best_parametric_model(T, E, scoring_method="AIC")
print(best_model)
# <lifelines.SplineFitter:"Spline_estimate", fitted with 686 total observations, 387 right-censored observations>
best_model.plot_hazard()

在图表上绘制多个图形¶
当调用.plot时,会返回一个axis对象,该对象可以传递给未来的.plot调用:
kmf.fit(data1)
ax = kmf.plot_survival_function()
kmf.fit(data2)
ax = kmf.plot_survival_function(ax=ax)
如果你有一个包含“T”、“E”和一些分类变量的pandas DataFrame,那么类似以下的内容将会起作用:
from matplotlib import pyplot as plt
from lifelines.datasets import load_waltons
from lifelines import KaplanMeierFitter
df = load_waltons()
ax = plt.subplot(111)
kmf = KaplanMeierFitter()
for name, grouped_df in df.groupby('group'):
kmf.fit(grouped_df["T"], grouped_df["E"], label=name)
kmf.plot_survival_function(ax=ax)
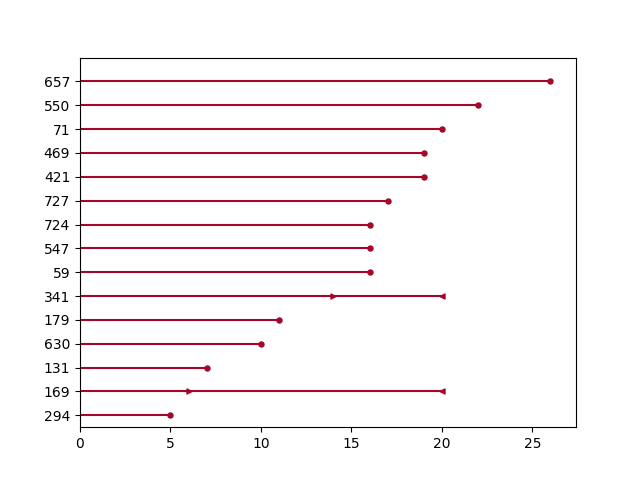
绘制区间删失数据¶
注意
lifelines v0.24.6 中的新功能
from lifelines.datasets import load_diabetes
from lifelines.plotting import plot_interval_censored_lifetimes
df_sample = load_diabetes().sample(frac=0.02)
ax = plot_interval_censored_lifetimes(df_sample['left'], df_sample['right'])
绘图选项和样式¶
让我们加载一些数据
from lifelines.datasets import load_waltons
waltons = load_waltons()
T = waltons['T']
E = waltons['E']
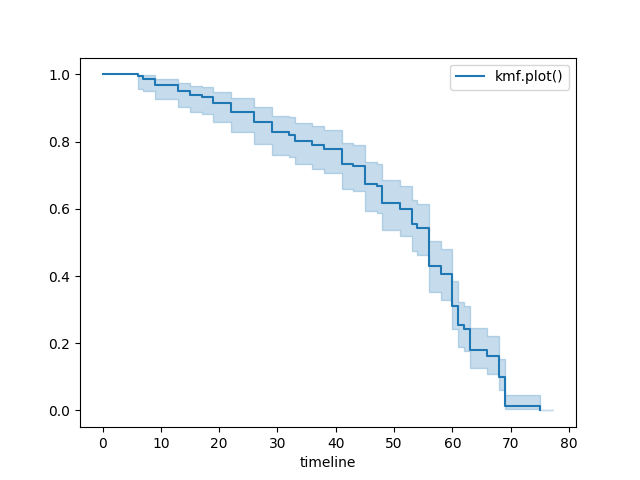
标准¶
kmf = KaplanMeierFitter()
kmf.fit(T, E, label="kmf.plot_survival_function()")
kmf.plot_survival_function()
显示审查和编辑标记¶
kmf.fit(T, E, label="kmf.plot_survival_function(show_censors=True, \ncensor_styles={'ms': 6, 'marker': 's'})")
kmf.plot_survival_function(show_censors=True, censor_styles={'ms': 6, 'marker': 's'})
隐藏置信区间¶
kmf.fit(T, E, label="kmf.plot_survival_function(ci_show=False)")
kmf.plot_survival_function(ci_show=False)
在图表下方显示风险计数¶
kmf.fit(T, E, label="label name")
kmf.plot_survival_function(at_risk_counts=True)
plt.tight_layout()
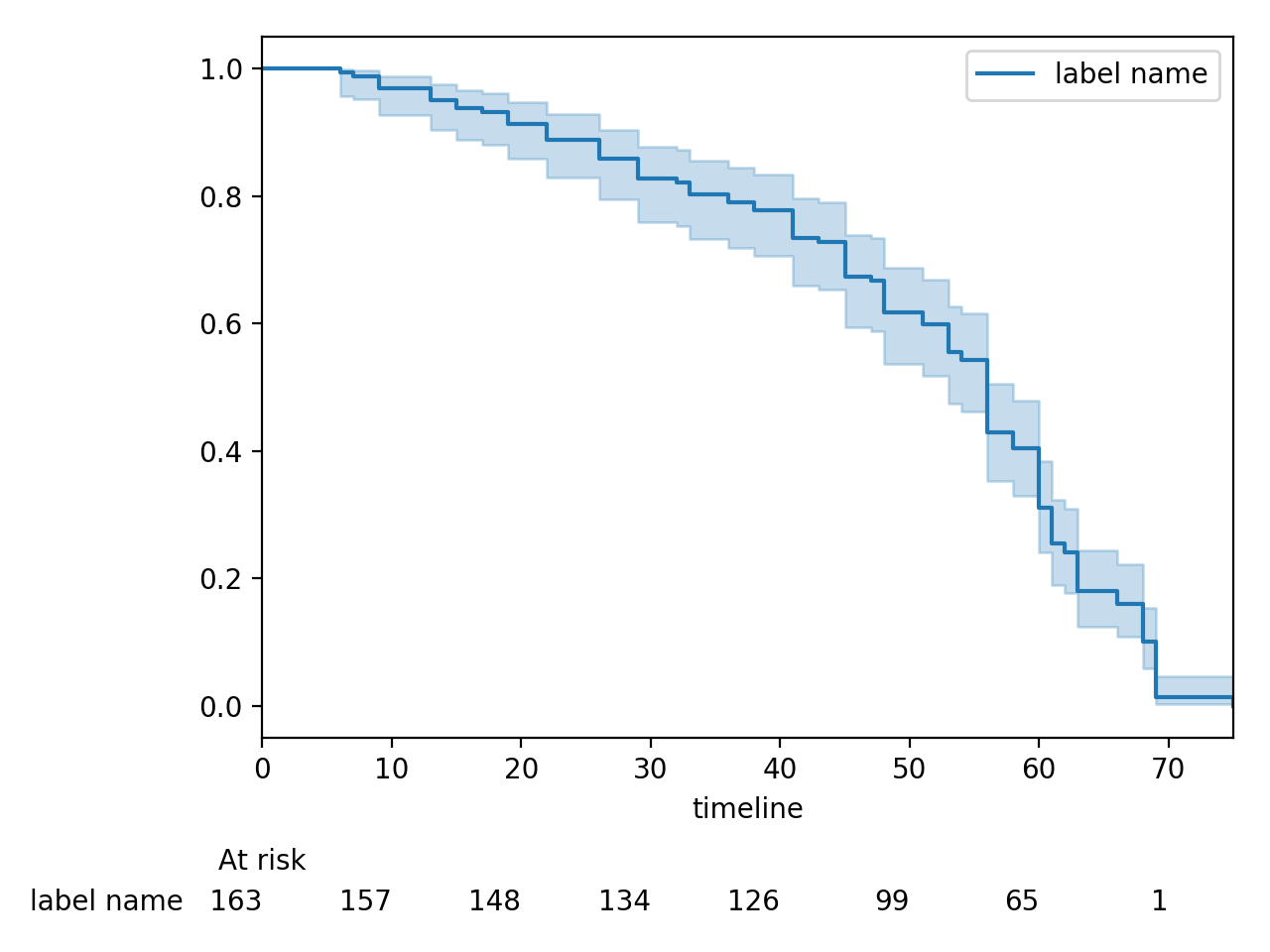
在图表下方显示多个风险计数¶
函数 lifelines.plotting.add_at_risk_counts() 允许你在图表的底部添加计数。例如:
from lifelines import KaplanMeierFitter
from lifelines.datasets import load_waltons
waltons = load_waltons()
ix = waltons['group'] == 'control'
ax = plt.subplot(111)
kmf_control = KaplanMeierFitter()
ax = kmf_control.fit(waltons.loc[ix]['T'], waltons.loc[ix]['E'], label='control').plot_survival_function(ax=ax)
kmf_exp = KaplanMeierFitter()
ax = kmf_exp.fit(waltons.loc[~ix]['T'], waltons.loc[~ix]['E'], label='exp').plot_survival_function(ax=ax)
from lifelines.plotting import add_at_risk_counts
add_at_risk_counts(kmf_exp, kmf_control, ax=ax)
plt.tight_layout()
将显示
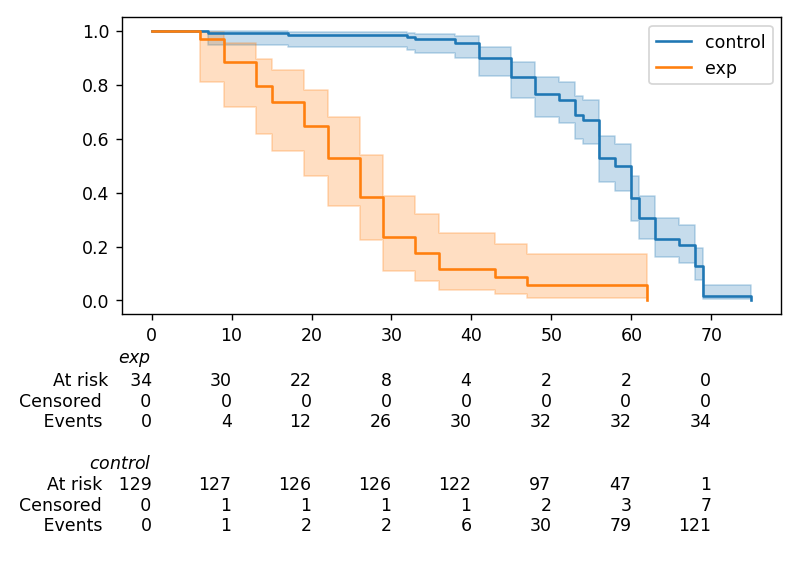
将生存表数据转换为lifelines格式¶
一些lifelines类是为每行代表一个个体的列表或数组设计的。如果你的数据是survival table格式的,有一个实用方法可以将其转换为lifelines格式。
示例: 假设你有一个CSV文件,数据如下所示:
时间 |
观察到的死亡人数 |
已审查 |
|---|---|---|
0 |
7 |
0 |
1 |
1 |
1 |
2 |
2 |
0 |
3 |
1 |
2 |
4 |
5 |
2 |
… |
… |
… |
import pandas as pd
from lifelines.utils import survival_events_from_table
df = pd.read_csv('file.csv')
df = df.set_index('time')
T, E, W = survival_events_from_table(df, observed_deaths_col='observed deaths', censored_col='censored')
# weights, W, is the number of occurrences of each observation - helps with data compression.
kmf = KaplanMeierFitter().fit(T, E, weights=W)
将观测数据转换为生存表格式¶
也许你有兴趣查看给定一些持续时间和删失向量的生存表。
from lifelines.utils import survival_table_from_events
table = survival_table_from_events(T, E)
print(table.head())
"""
removed observed censored entrance at_risk
event_at
0 0 0 0 60 60
2 2 1 1 0 60
3 3 1 2 0 58
4 5 3 2 0 55
5 12 6 6 0 50
"""
设置估计的索引/时间线¶
假设您的数据集在时间60附近有寿命分组,因此在拟合lifelines.fitters.kaplan_meier_fitter.KaplanMeierFitter后,您的生存函数可能看起来像这样:
print(kmf.survival_function_)
"""
KM-estimate
0 1.00
47 0.99
49 0.97
50 0.96
51 0.95
52 0.91
53 0.86
54 0.84
55 0.79
56 0.74
57 0.71
58 0.67
59 0.58
60 0.49
61 0.41
62 0.31
63 0.24
64 0.19
65 0.14
66 0.10
68 0.07
69 0.04
70 0.02
71 0.01
74 0.00
"""
你希望的是有一个可预测且完整的索引,从40到75。(注意在上面的索引中,最后两个时间点不相邻——原因是观察到在时间72或73时没有寿命存在)。这对于在特定时间点比较多个生存函数特别有用。为此,所有拟合方法都接受一个timeline参数:
kmf.fit(T, timeline=range(40,75))
print(kmf.survival_function_)
"""
KM-estimate
40 1.00
41 1.00
42 1.00
43 1.00
44 1.00
45 1.00
46 1.00
47 0.99
48 0.99
49 0.97
50 0.96
51 0.95
52 0.91
53 0.86
54 0.84
55 0.79
56 0.74
57 0.71
58 0.67
59 0.58
60 0.49
61 0.41
62 0.31
63 0.24
64 0.19
65 0.14
66 0.10
67 0.10
68 0.07
69 0.04
70 0.02
71 0.01
72 0.01
73 0.01
74 0.00
"""
lifelines 将智能地向前填充估计值到未观察到的时间点。
从表中获取生存数据的SQL查询示例¶
以下是从关系数据库中获取示例数据集的一种方法(这可能因您的数据库而异):
SELECT
id,
DATEDIFF('dd', started_at, COALESCE(ended_at, CURRENT_DATE)) AS "T",
(ended_at IS NOT NULL) AS "E"
FROM table
解释¶
每一行是一个id,一个持续时间,以及一个布尔值,表示事件是否发生。回想一下,如果事件确实发生了,我们表示为“True”,即ended_at被填写(我们观察到了ended_at)。例如:
id |
T |
E |
|---|---|---|
10 |
40 |
真 |
11 |
42 |
假 |
12 |
42 |
假 |
13 |
36 |
真 |
14 |
33 |
真 |
获取时间变化数据的示例SQL查询和转换¶
对于Cox时变模型,我们在时变回归的数据集创建中讨论了数据集应该是什么样子的。通常我们有一个基础数据集,然后我们合并协变量数据集。以下是一些端到端的SQL查询和Python转换。
基础数据集: base_df¶
SELECT
id,
group,
DATEDIFF('dd', dt.started_at, COALESCE(dt.ended_at, CURRENT_DATE)) AS "T",
(ended_at IS NOT NULL) AS "E"
FROM dimension_table dt
时变变量: cv¶
-- this could produce more than 1 row per subject
SELECT
id,
DATEDIFF('dd', dt.started_at, ft.event_at) AS "time",
ft.var1
FROM fact_table ft
JOIN dimension_table dt
USING(id)
from lifelines.utils import to_long_format
from lifelines.utils import add_covariate_to_timeline
base_df = to_long_format(base_df, duration_col="T")
df = add_covariate_to_timeline(base_df, cv, duration_col="time", id_col="id", event_col="E")
事件变量: event_df¶
另一个非常常见的操作是将事件数据添加到我们的时变数据集中。例如,一个包含事件日期信息的数据集/SQL表(如果事件未发生则为NULL)。一个示例SQL查询可能如下所示:
SELECT
id,
DATEDIFF('dd', dt.started_at, ft.event1_at) AS "E1",
DATEDIFF('dd', dt.started_at, ft.event2_at) AS "E2",
DATEDIFF('dd', dt.started_at, ft.event3_at) AS "E3"
...
FROM dimension_table dt
在Pandas中,这可能看起来像:
"""
id E1 E2 E3
0 1 1.0 NaN 2.0
1 2 NaN 5.0 NaN
2 3 3.0 5.0 7.0
...
"""
最初,这不能添加到我们的基线时变数据集中。使用 lifelines.utils.covariates_from_event_matrix() 我们可以将这样的 DataFrame 转换为可以轻松添加的格式。
from lifelines.utils import covariates_from_event_matrix
cv = covariates_from_event_matrix(event_df, id_col='id')
print(cv)
"""
id duration E1 E2 E3
0 1 1.0 1 0 0
1 1 2.0 0 1 0
2 2 5.0 0 1 0
3 3 3.0 1 0 0
4 3 5.0 0 1 0
5 3 7.0 0 0 1
"""
base_df = add_covariate_to_timeline(base_df, cv, duration_col="time", id_col="id", event_col="E")
随时间变化的协变量的累积和示例¶
通常我们会有交易协变量数据集或状态协变量数据集。在交易数据集中,将协变量相加以表示随时间推移的治疗管理可能是有意义的。例如,在充满风险的初创企业世界中,我们可能希望汇总在某个时间点收到的资金金额。我们可能还对最后一轮融资的金额感兴趣。以下是一个执行此操作的示例:
假设我们有一个初始的初创公司DataFrame,如下所示:
seed_df = pd.DataFrame([
{'id': 'FB', 'E': True, 'T': 12, 'funding': 0},
{'id': 'SU', 'E': True, 'T': 10, 'funding': 0},
])
以及一个表示融资轮次的协变量DataFrame,如下所示:
cv = pd.DataFrame([
{'id': 'FB', 'funding': 30, 't': 5},
{'id': 'FB', 'funding': 15, 't': 10},
{'id': 'FB', 'funding': 50, 't': 15},
{'id': 'SU', 'funding': 10, 't': 6},
{'id': 'SU', 'funding': 9, 't': 10},
])
我们可以执行以下操作来获取累计收到的资金和最新一轮的资金:
from lifelines.utils import to_long_format
from lifelines.utils import add_covariate_to_timeline
df = seed_df.pipe(to_long_format, 'T')\
.pipe(add_covariate_to_timeline, cv, 'id', 't', 'E', cumulative_sum=True)\
.pipe(add_covariate_to_timeline, cv, 'id', 't', 'E', cumulative_sum=False)
"""
start cumsum_funding funding stop id E
0 0 0.0 0.0 5.0 FB False
1 5 30.0 30.0 10.0 FB False
2 10 45.0 15.0 12.0 FB True
3 0 0.0 0.0 6.0 SU False
4 6 10.0 10.0 10.0 SU False
5 10 19.0 9.0 10.0 SU True
"""
在CoxPH模型下的样本量确定¶
假设您希望在CoxPH模型下测量两个群体之间的风险比。也就是说,我们想要评估假设
H0: 相对风险比 = 1 vs H1: 相对风险比 != 1,其中相对风险比是实验组与对照组的\(\exp{\left(\beta\right)}\)。我们事先对达到一定统计功效所需的两组样本量感兴趣。要在lifelines中实现这一点,可以使用lifelines.statistics.sample_size_necessary_under_cph()函数。例如:
from lifelines.statistics import sample_size_necessary_under_cph
desired_power = 0.8
ratio_of_participants = 1.
p_exp = 0.25
p_con = 0.35
postulated_hazard_ratio = 0.7
n_exp, n_con = sample_size_necessary_under_cph(desired_power, ratio_of_participants, p_exp, p_con, postulated_hazard_ratio)
# (421, 421)
这假设您已经估计了实验组和对照组中事件发生的概率。这可以从之前的实验中确定。
CoxPH模型下的功率确定¶
假设你希望在CoxPH模型下测量两个群体之间的风险比。为了确定风险比假设检验的统计功效,在CoxPH模型下,我们可以使用lifelines.statistics.power_under_cph()。也就是说,假设我们想知道拒绝相对风险比为1的零假设的概率,假设相对风险比确实与1不同。这个函数将给你那个概率。
from lifelines.statistics import power_under_cph
n_exp = 50
n_con = 100
p_exp = 0.25
p_con = 0.35
postulated_hazard_ratio = 0.5
power = power_under_cph(n_exp, n_con, p_exp, p_con, postulated_hazard_ratio)
# 0.4957
Cox比例风险模型中的收敛问题¶
由于Cox比例风险模型中的系数估计是使用Newton-Raphson算法完成的,有时会出现收敛问题。以下是一些常见症状和解决方法:
首次检查:在输出中查找
ConvergenceWarning。大多数情况下,收敛问题是由于数据集中的问题引起的。lifelines在拟合之前会对数据集进行检查,并向用户输出警告。delta contains nan value(s).: 首先尝试在fit函数中添加show_progress=True。如果delta中的值无限增长,可能是step_size太大。尝试将其设置为一个较小的值(0.1-0.5)。Convergence halted due to matrix inversion problems: 这意味着您的数据集中存在高度共线性。也就是说,某一列等于其他一列或多列的线性组合。此错误的常见原因是对分类变量进行虚拟化但没有删除一列,或者数据集中存在某种层次结构。尝试通过以下方式找到关系:向模型添加惩罚项,例如:CoxPHFitter(penalizer=0.1).fit(…),直到模型收敛。在print_summary()中,具有高共线性的系数将在coefs列中具有较大的(绝对值)幅度。
使用方差膨胀因子(VIF)来查找冗余变量。
查看数据集的相关系数矩阵,或
一些系数比其他系数大很多数量级,而且系数的标准误差也很大或者结果中有
nan。这可以通过在拟合的CoxPHFitter对象上使用print_summary方法来查看。寻找关于方差过小的
ConvergenceWarning。数据集可能包含一个常量列,这对回归没有提供任何信息(Cox模型不像其他回归模型那样有传统的“截距”项)。数据是完全可分的,这意味着存在一个协变量完全决定了事件是否发生。例如,对于数据集中所有的“死亡”事件,存在一个在所有事件中保持不变的协变量。在
fit调用后查找ConvergenceWarning。参见https://stats.stackexchange.com/questions/11109/how-to-deal-with-perfect-separation-in-logistic-regression与上述相关,协变量与持续时间之间的关系可能完全确定。例如,如果协变量与持续时间之间的秩相关非常接近1或-1,那么仅使用该协变量就可以任意增加对数似然。在
fit调用后查找ConvergenceWarning。另一个问题可能是数据集中存在共线性关系。请参见上面的第3点。
如果添加一个非常小的
penalizer显著改变了结果(CoxPHFitter(penalizer=0.0001)),那么这可能意味着迭代算法中的步长太大。尝试减小它(.fit(..., step_size=0.50)或更小),并将penalizer项返回到0。如果使用
strata参数,请确保您的分层组大小不要太小。尝试df.groupby(strata).size()。
在Cox模型中为观测值添加权重¶
在模型中有两种常见的权重使用方式。第一种是作为数据大小缩减技术(称为案例权重)。如果数据集中有多个具有相同属性(包括持续时间和事件)的个体,那么它们的似然贡献也是相同的。因此,我们可以计算一次对数似然,并将其乘以具有相同属性的用户数量,而不是为每个个体计算对数似然。在实践中,这涉及首先按协变量对个体进行分组和计数。例如,使用Rossi数据集,我们将使用Pandas按属性进行分组(但其他数据处理工具,如Spark,也可以做到这一点):
from lifelines.datasets import load_rossi
rossi = load_rossi()
rossi_weights = rossi.copy()
rossi_weights['weights'] = 1.
rossi_weights = rossi_weights.groupby(rossi.columns.tolist())['weights'].sum()\
.reset_index()
原始数据集有432行,而分组后的数据集有387行,并增加了一个weights列。CoxPHFitter有一个额外的参数用于指定哪一列是权重列。
from lifelines import CoxPHFitter
cph = CoxPHFitter()
cph.fit(rossi_weights, 'week', 'arrest', weights_col='weights')
拟合应该更快,结果应与未加权数据集相同。此选项也可在CoxTimeVaryingFitter中使用。
权重的第二个用途是抽样权重。这些通常是正的、非整数的权重,代表了对观测值的一些人为的欠/过采样(例如:治疗权重的逆概率)。建议在调用fit时设置robust=True,因为对于抽样权重,通常的标准误差是不正确的。robust标志将使用三明治估计器来计算标准误差。
警告
三明治估计器的实现在处理结(ties)时(在Efron处理结的方法下)并不正确,并且会根据结的频率给出与其他软件略有或显著不同的结果。
Cox模型中科目之间的相关性¶
在某些情况下,您的数据集可能包含相关的主题,这违反了独立同分布的假设。这种情况可能发生在哪些情况下?
如果一个主题在数据集中出现多次(当主题可以多次发生事件时很常见)
如果使用匹配技术,如倾向得分匹配,配对之间存在相关性。
在这两种情况下,从未调整的Cox模型报告的标准误差将是错误的。为了调整这些相关性,fit()中有一个cluster_col关键字,允许您指定DataFrame中包含相关主题指定信息的列。例如,如果第1行和第2行的主题是相关的,但没有其他主题是相关的,那么cluster_col列应该在第1行和第2行具有相同的值,而所有其他行的值应该是唯一的。另一个例子:对于匹配的对,每对中的每个主题应该具有相同的值。
from lifelines.datasets import load_rossi
from lifelines import CoxPHFitter
rossi = load_rossi()
# this may come from a database, or other libraries that specialize in matching
matched_pairs = [
(156, 230),
(275, 228),
(61, 252),
(364, 201),
(54, 340),
(130, 33),
(183, 145),
(268, 140),
(332, 259),
(314, 413),
(330, 211),
(372, 255),
# ...
]
rossi['id'] = None # we will populate this column
for i, pair in enumerate(matched_pairs):
subjectA, subjectB = pair
rossi.loc[subjectA, 'id'] = i
rossi.loc[subjectB, 'id'] = i
rossi = rossi.dropna(subset=['id'])
cph = CoxPHFitter()
cph.fit(rossi, 'week', 'arrest', cluster_col='id')
指定cluster_col将处理相关性,并调用稳健的三明治估计器来计算标准误差(与设置robust=True相同)。
将lifelines模型序列化到磁盘¶
当你想将lifelines模型保存到磁盘(并在以后加载)时,你可以使用最流行的序列化库(dill、pickle、joblib)中的loads和dumps API:
from dill import loads, dumps
from pickle import loads, dumps
s_cph = dumps(cph)
cph_new = loads(s_cph)
cph_new.summary
s_kmf = dumps(kmf)
kmf_new = loads(s_kmf)
kmf_new.survival_function_
上面的代码将训练好的模型保存为内存中的二进制对象。要将lifelines模型序列化到磁盘上的指定路径:
import pickle
with open('/path/my.pickle', 'wb') as f:
pickle.dump(cph, f) # saving my trained cph model as my.pickle
with open('/path/my.pickle', 'rb') as f:
cph_new = pickle.load(f)
cph_new.summary # should produce the same output as cph.summary
生成一个LaTex或HTML表格¶
在版本0.23.1中新增,lifelines 模型现在能够从print_summary选项中输出LaTeX或HTML表格:
from lifelines.datasets import load_rossi
from lifelines import CoxPHFitter
rossi = load_rossi()
cph = CoxPHFitter().fit(rossi, 'week', 'arrest')
# print a LaTeX table:
cph.print_summary(style="latex")
# print a HTML summary and table:
cph.print_summary(style="html")
为了在LaTeX中使用生成的表格摘要,请确保在导言区导入booktabs包(\usepackage{booktabs}),因为这是正确显示表格所必需的。
过滤一个print_summary表格¶
由print_summary提供的信息可能很多,甚至对于一些屏幕来说太多了。你可以使用columns关键字参数过滤到特定的列(默认是显示所有列):
from lifelines.datasets import load_rossi
from lifelines import CoxPHFitter
rossi = load_rossi()
cph = CoxPHFitter().fit(rossi, 'week', 'arrest')
cph.print_summary(columns=["coef", "se(coef)", "p"])
修复FormulaSyntaxError¶
作为 lifelines v0.25.0 的一部分,公式可以用来建模你的数据框。如果你的数据框列名包含空格、句点或其他字符,这可能会导致问题。最简单的解决方法就是更改你的列名:
df = pd.DataFrame({
'T': [1, 2, 3, 4],
'column with spaces': [1.5, 1.0, 2.5, 1.0],
'column.with.periods': [2.5, -1.0, -2.5, 1.0],
'column': [2.0, 1.0, 3.0, 4.0]
})
cph = CoxPHFitter().fit(df, 'T')
"""
FormulaSyntaxError:
...
"""
df.columns = df.columns.str.replace(' ', '')
df.columns = df.columns.str.replace('.', '')
cph = CoxPHFitter().fit(df, 'T')
"""
👍
"""
另一个选择是使用公式语法来处理这个问题:
df = pd.DataFrame({
'T': [1, 2, 3, 4],
'column with spaces': [1.5, 1.0, 2.5, 1.0],
'column.with.periods': [2.5, -1.0, -2.5, 1.0],
'column': [2.0, 1.0, 3.0, 4.0]
})
cph = CoxPHFitter().fit(df, 'T', formula="column + Q('column with spaces') + Q('column.with.periods')")