使用BART的分位数回归#
Running on PyMC v5.9.2+10.g547bcb481
%config InlineBackend.figure_format = "retina"
RANDOM_SEED = 5781
np.random.seed(RANDOM_SEED)
az.style.use("arviz-darkgrid")
通常在进行回归时,我们会建模某个分布的条件均值。常见的情况包括对连续无界响应使用正态分布,对计数数据使用泊松分布等。
分位数回归,估计的是响应变量的条件分位数。如果分位数是0.5,那么我们将估计中位数(而不是均值),这可以作为一种执行稳健回归的方式,类似于使用学生t分布而不是正态分布。但对于某些问题,我们实际上关心的是响应变量在均值(或中位数)之外的行为。例如,在医学研究中,病理或潜在健康风险发生在高或低分位数,例如,超重和体重不足。在其他一些领域,如生态学,由于变量之间存在复杂的相互作用,分位数回归是合理的,其中一个变量对另一个变量的影响在变量的不同范围内是不同的。
非对称拉普拉斯分布#
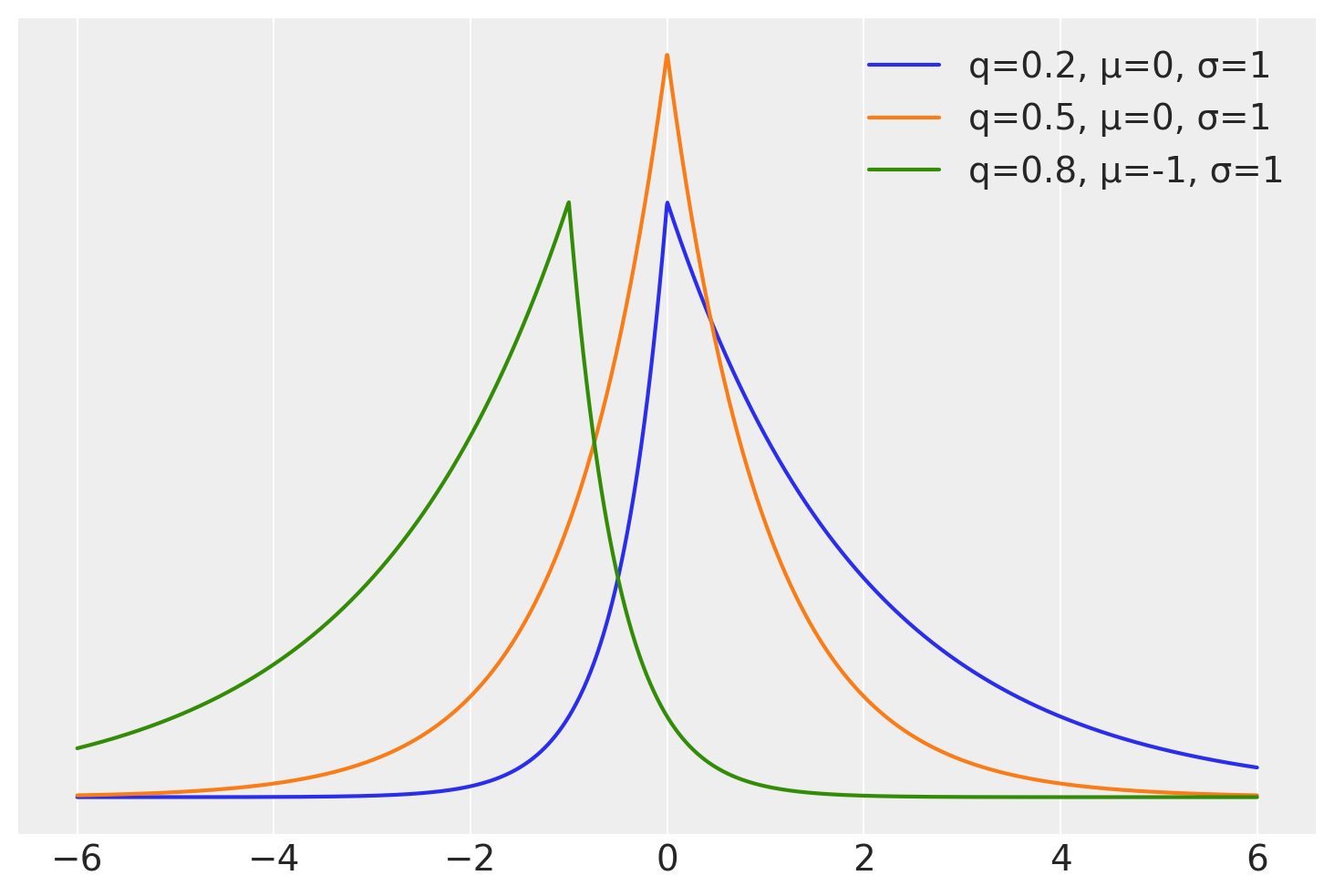
起初,思考我们应该使用哪种分布作为分位数回归的可能性或如何编写分位数回归的贝叶斯模型可能会显得有些奇怪。但事实证明,答案非常简单,我们只需要使用非对称拉普拉斯分布。这个分布有一个参数控制均值,另一个控制尺度,第三个控制不对称性。关于这个不对称参数,至少有两种替代的参数化方法。在 \(\kappa\) 参数方面,它从 0 到 \(\infty\),而在 \(q\) 参数方面,它是一个介于 0 和 1 之间的数字。后一种参数化方法对于分位数回归来说更为直观,因为我们可以直接将其解释为感兴趣的分位数。
在下一个单元格中,我们计算来自非对称拉普拉斯族的3个分布的概率密度函数
x = np.linspace(-6, 6, 2000)
for q, m in zip([0.2, 0.5, 0.8], [0, 0, -1]):
κ = (q / (1 - q)) ** 0.5
plt.plot(x, stats.laplace_asymmetric(κ, m, 1).pdf(x), label=f"q={q:}, μ={m}, σ=1")
plt.yticks([])
plt.legend();
我们将使用一个简单的数据集来模拟荷兰儿童和年轻男性的体重指数(BMI)作为他们年龄的函数。
try:
bmi = pd.read_csv(Path("..", "data", "bmi.csv"))
except FileNotFoundError:
bmi = pd.read_csv(pm.get_data("bmi.csv"))
bmi.plot(x="age", y="bmi", kind="scatter");
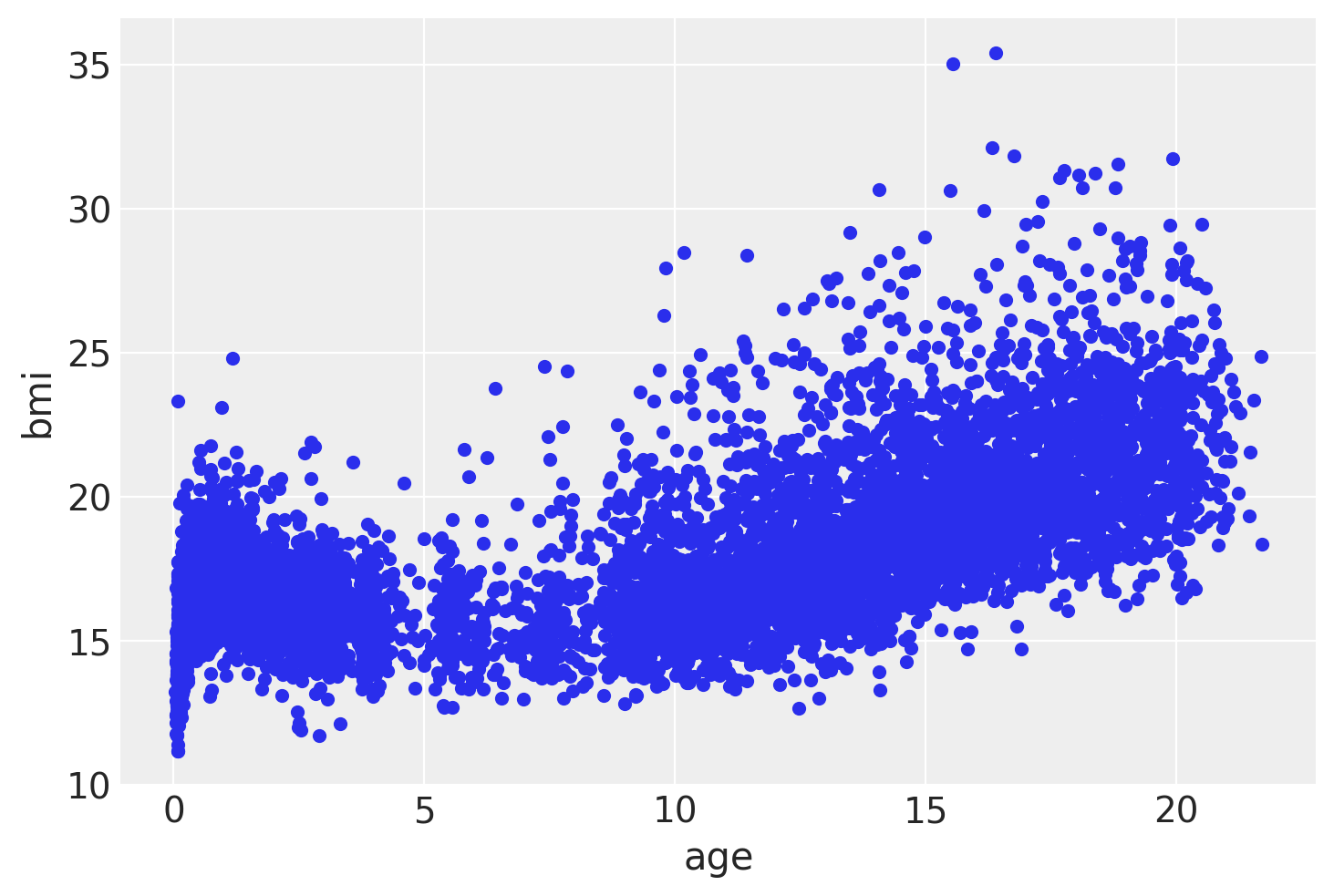
从上图可以看出,BMI与年龄之间的关系远非线性,因此我们将使用BART。
我们将对3个分位数0.1、0.5和0.9进行建模。我们可以通过拟合3个单独的模型来计算这个量,唯一的不同是q的值,即非对称拉普拉斯分布的值。或者,我们可以像在y_stack中那样堆叠观测值,并拟合一个单一的模型。
array([[0.1],
[0.5],
[0.9]])
with pm.Model() as model:
μ = pmb.BART("μ", X, y, shape=(3, 7294))
σ = pm.HalfNormal("σ", 5)
obs = pm.AsymmetricLaplace("obs", mu=μ, b=σ, q=quantiles, observed=y_stack)
idata = pm.sample(compute_convergence_checks=False)
/home/osvaldo/anaconda3/envs/pymc/lib/python3.11/site-packages/pytensor/tensor/math.py:1107: FutureWarning: sgn is deprecated and will stop working in the future, use sign instead.
warnings.warn(
/home/osvaldo/anaconda3/envs/pymc/lib/python3.11/site-packages/pytensor/tensor/math.py:1107: FutureWarning: sgn is deprecated and will stop working in the future, use sign instead.
warnings.warn(
/home/osvaldo/anaconda3/envs/pymc/lib/python3.11/site-packages/pytensor/tensor/math.py:1107: FutureWarning: sgn is deprecated and will stop working in the future, use sign instead.
warnings.warn(
/home/osvaldo/anaconda3/envs/pymc/lib/python3.11/site-packages/pytensor/tensor/math.py:1107: FutureWarning: sgn is deprecated and will stop working in the future, use sign instead.
warnings.warn(
/home/osvaldo/anaconda3/envs/pymc/lib/python3.11/site-packages/pytensor/tensor/math.py:1107: FutureWarning: sgn is deprecated and will stop working in the future, use sign instead.
warnings.warn(
/home/osvaldo/anaconda3/envs/pymc/lib/python3.11/site-packages/pytensor/tensor/math.py:1107: FutureWarning: sgn is deprecated and will stop working in the future, use sign instead.
warnings.warn(
/home/osvaldo/anaconda3/envs/pymc/lib/python3.11/site-packages/pytensor/tensor/math.py:1107: FutureWarning: sgn is deprecated and will stop working in the future, use sign instead.
warnings.warn(
Multiprocess sampling (4 chains in 4 jobs)
CompoundStep
>PGBART: [μ]
>NUTS: [σ]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 376 seconds.
我们可以在下图中看到3条拟合曲线的结果。一个突出的特点是,中位数(橙色)线与另外两条线之间的差距或距离并不相同。此外,尽管曲线的形状遵循相似的模式,但并不完全相同。
plt.plot(bmi.age, bmi.bmi, ".", color="0.5")
for idx, q in enumerate(quantiles[:, 0]):
plt.plot(
bmi.age,
idata.posterior["μ"].mean(("chain", "draw")).sel(μ_dim_0=idx),
label=f"q={q:}",
lw=3,
)
plt.legend();
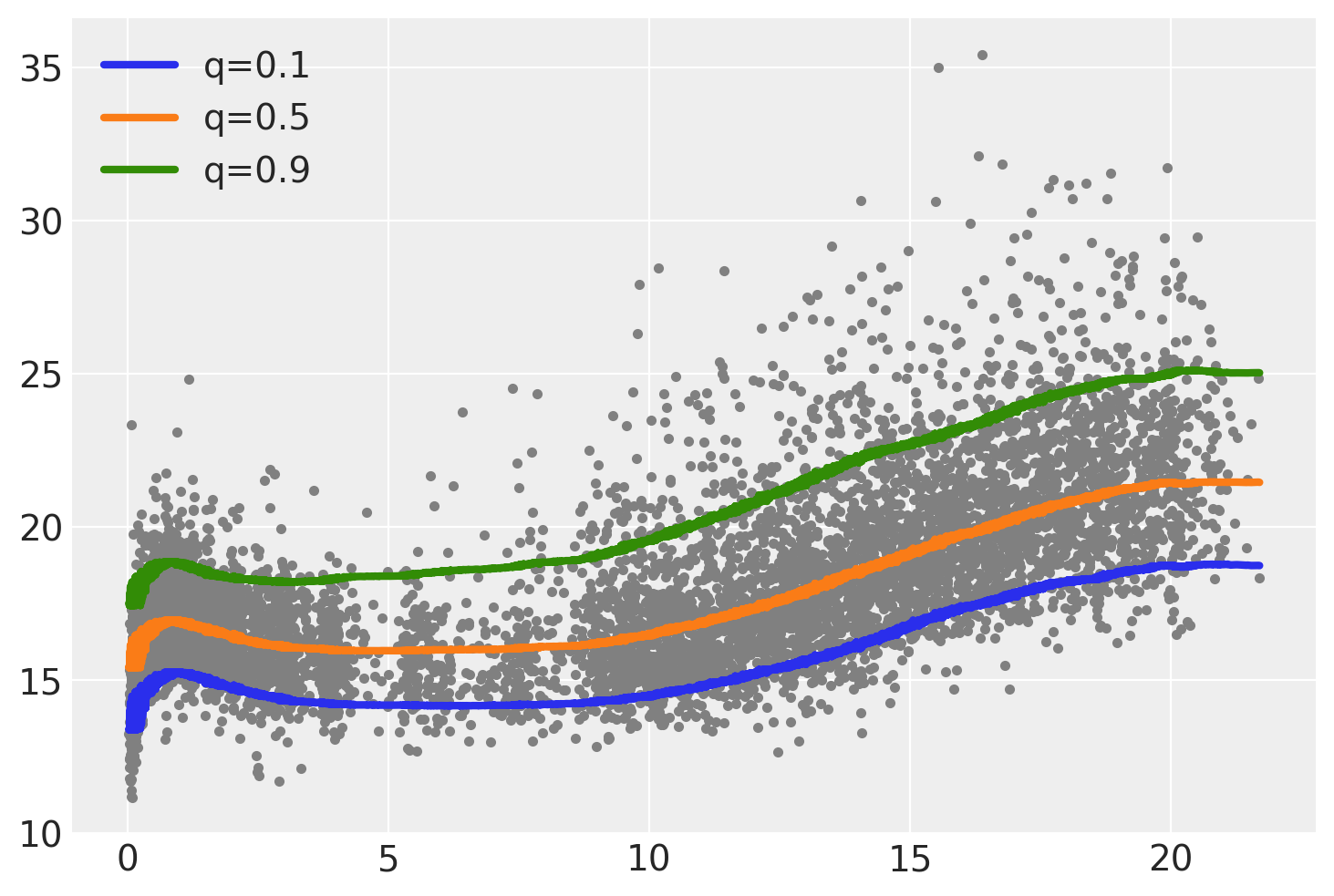
为了更好地理解这些评论,让我们使用正态似然计算一个BART回归,然后从该拟合中计算相同的3个分位数。
y = bmi.bmi.values
x = bmi.age.values[:, None]
with pm.Model() as model:
μ = pmb.BART("μ", x, y)
σ = pm.HalfNormal("σ", 5)
obs = pm.Normal("obs", mu=μ, sigma=σ, observed=y)
idata_g = pm.sample(compute_convergence_checks=False)
idata_g.extend(pm.sample_posterior_predictive(idata_g))
Multiprocess sampling (4 chains in 4 jobs)
CompoundStep
>PGBART: [μ]
>NUTS: [σ]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 85 seconds.
Sampling: [obs]
idata_g_mean_quantiles = idata_g.posterior_predictive["obs"].quantile(
quantiles[:, 0], ("chain", "draw")
)
plt.plot(bmi.age, bmi.bmi, ".", color="0.5")
for q in quantiles[:, 0]:
plt.plot(bmi.age.values, idata_g_mean_quantiles.sel(quantile=q), label=f"q={q:}")
plt.legend()
plt.xlabel("Age")
plt.ylabel("BMI");
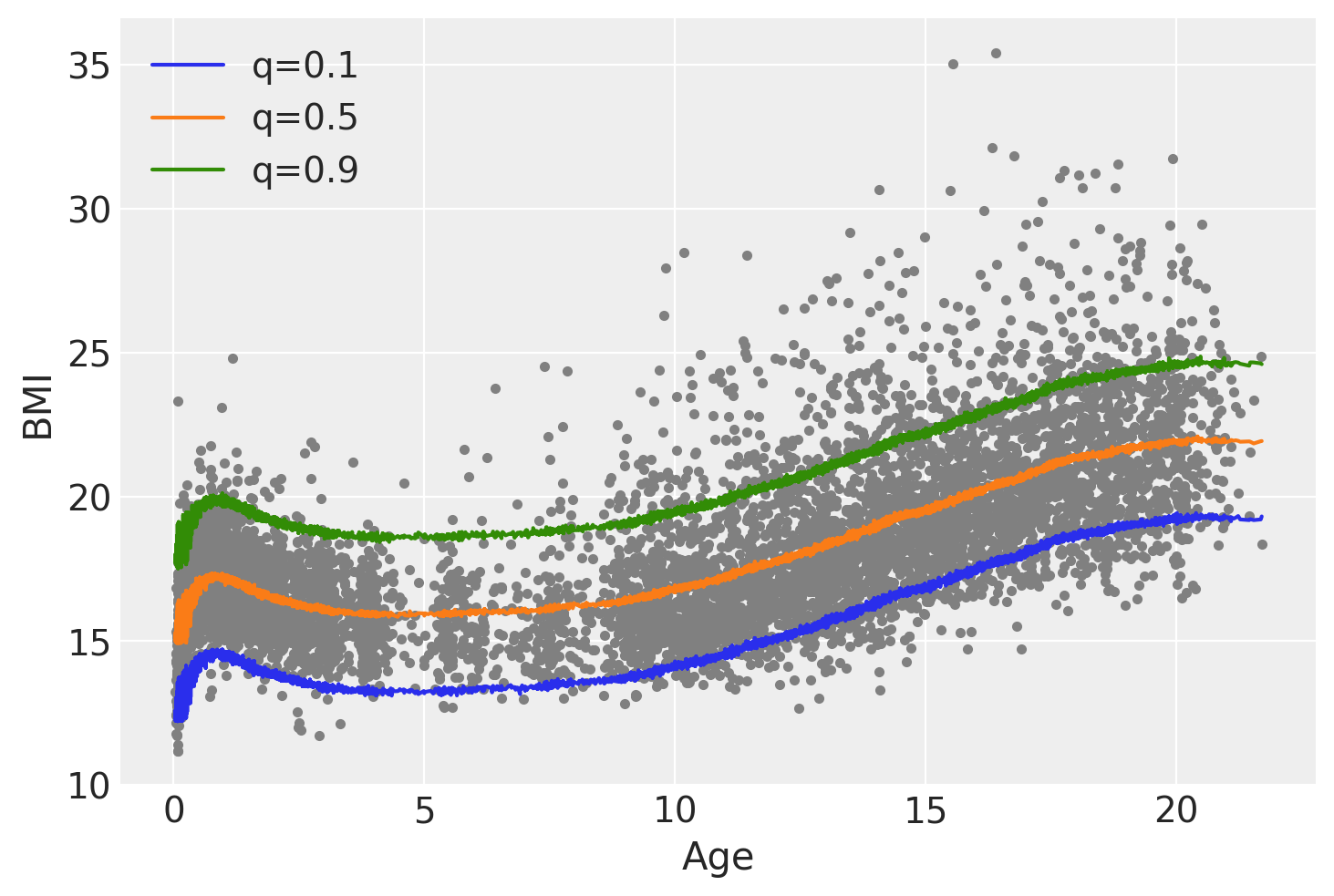
我们可以看到,当我们使用正态似然时,从该拟合中我们计算分位数,分位数 q=0.1 和 q=0.9 相对于 q=0.5 是对称的,曲线的形状基本上是相同的,只是上下移动。此外,非对称拉普拉斯族允许模型考虑随着年龄增加BMI的变异性增加,而对于高斯族,这种变异性始终保持不变。
参考资料#
Miriana Quiroga, Pablo G Garay, Juan M. Alonso, Juan Martin Loyola, 和 Osvaldo A Martin. 贝叶斯加性回归树用于概率编程。2022年。URL: https://arxiv.org/abs/2206.03619, doi:10.48550/ARXIV.2206.03619.
Osvaldo A Martin, Ravin Kumar, 和 Junpeng Lao. Python中的贝叶斯建模与计算. Chapman and Hall/CRC, 2021. doi:10.1201/9781003019169.
水印#
%load_ext watermark
%watermark -n -u -v -iv -w -p pytensor,xarray
Last updated: Sat Nov 18 2023
Python implementation: CPython
Python version : 3.11.5
IPython version : 8.16.1
pytensor: 2.17.3
xarray : 2023.10.1
pymc : 5.9.2+10.g547bcb481
matplotlib: 3.8.0
pandas : 2.1.2
arviz : 0.16.1
numpy : 1.24.4
pymc_bart : 0.5.3
scipy : 1.11.3
Watermark: 2.4.3