反事实推理:计算因COVID-19导致的超额死亡#
因果推理和反事实思维是非常有趣但复杂的主题!尽管如此,我们可以通过相对简单的例子来理解这些概念。本笔记本专注于贝叶斯因果推理的概念及其使用PyMC的实际实现。
我们使用计算因COVID-19导致的超额死亡的令人警醒但重要的例子来做到这一点。因此,本笔记本中的想法与Google的CausalImpact(参见Brodersen 等人 [2015])有很强的重叠。实际上,我们将尝试使用来自英格兰和威尔士的数据,估计自COVID-19爆发以来的“超额死亡”数量。超额死亡定义为:
关于超额死亡的声明需要因果/反事实推理。虽然报告的死亡人数只是现实世界中一个真实可观察事实的(可能是噪声和/或滞后的)衡量标准,但预期死亡人数是无法测量的,因为这些在我们的时间线上从未实现。也就是说,预期死亡人数是一个反事实的思想实验,我们可以问“如果……会发生/将会发生什么?”
总体策略#
我们实际上如何进行呢?我们将遵循这个策略:
导入报告的死亡总数(我们的结果变量)以及一些合理的预测变量:
平均月温度
一年中的月份,我们用它来模拟季节性效应
以及用于模拟任何潜在线性趋势的时间。
拆分为
pre和post新冠疫情的数据集。这是一个重要的步骤。我们希望基于我们在新冠疫情之前所知道的信息来构建一个模型,以便我们可以根据新冠疫情之前的数据来构建我们的反事实预测。基于
pre数据集估计模型参数。反向预测模型在COVID-19前时期预期的死亡人数。这不是一个反事实,而是告诉我们模型在解释已观察数据方面的能力。
反事实推理 - 我们使用我们的模型来构建一个反事实预测。如果没有COVID-19,我们未来会看到什么?这可以通过使用著名的do-operator来实现。实际上,我们通过对样本外数据进行后验预测来实现这一点。
通过将报告的死亡人数与我们的反事实(预期死亡人数)进行比较,计算超额死亡人数。
建模策略#
我们可以采用许多不同的方法进行建模。由于我们处理的是时间序列数据,因此使用时间序列建模方法是非常合理的。例如,Google的CausalImpact使用了一种贝叶斯结构时间序列模型,但我们还可以选择许多其他的时间序列模型。
但由于本案例研究的重点是反事实推理,而不是时间序列建模的具体细节,我选择了更简单的线性回归方法来构建时间序列模型(参见Martin 等 [2021]了解更多)。
因果推断免责声明#
读者应该注意,我们在这里可以做出的因果声明当然是有一定限制的。如果我们处理的是一个营销案例,在这个案例中我们进行了一段时间的促销活动,并希望对超额销售做出推论,那么我们只有在进行了尽职调查,考虑了促销期间可能发生的其他因素后,才能做出强有力的因果声明。
同样,自2020年1月(首次记录COVID-19病例的时间)以来,英国(英格兰和威尔士)发生了许多其他变化。因此,如果我们想要做到万无一失,我们应该考虑其他可能相关的因素。
最后,我们不声称有\(x\)人直接死于COVID-19病毒。超额死亡概念的优点在于它捕捉了所有超出我们预期的死亡原因。因此,它不仅涵盖了直接死于COVID-19病毒的人,还包括了病毒及其护理可用性的所有下游影响,例如。
import calendar
import os
import arviz as az
import matplotlib.dates as mdates
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import pymc as pm
import pytensor.tensor as pt
import seaborn as sns
import xarray as xr
%config InlineBackend.figure_format = 'retina'
RANDOM_SEED = 8927
rng = np.random.default_rng(RANDOM_SEED)
az.style.use("arviz-darkgrid")
现在让我们定义一些辅助函数
Show code cell content
def ZeroSumNormal(name, *, sigma=None, active_dims=None, dims, model=None):
model = pm.modelcontext(model=model)
if isinstance(dims, str):
dims = [dims]
if isinstance(active_dims, str):
active_dims = [active_dims]
if active_dims is None:
active_dims = dims[-1]
def extend_axis(value, axis):
n_out = value.shape[axis] + 1
sum_vals = value.sum(axis, keepdims=True)
norm = sum_vals / (pt.sqrt(n_out) + n_out)
fill_val = norm - sum_vals / pt.sqrt(n_out)
out = pt.concatenate([value, fill_val], axis=axis)
return out - norm
dims_reduced = []
active_axes = []
for i, dim in enumerate(dims):
if dim in active_dims:
active_axes.append(i)
dim_name = f"{dim}_reduced"
if name not in model.coords:
model.add_coord(dim_name, length=len(model.coords[dim]) - 1, mutable=False)
dims_reduced.append(dim_name)
else:
dims_reduced.append(dim)
raw = pm.Normal(f"{name}_raw", sigma=sigma, dims=dims_reduced)
for axis in active_axes:
raw = extend_axis(raw, axis)
return pm.Deterministic(name, raw, dims=dims)
def format_x_axis(ax, minor=False):
# major ticks
ax.xaxis.set_major_formatter(mdates.DateFormatter("%Y %b"))
ax.xaxis.set_major_locator(mdates.YearLocator())
ax.grid(which="major", linestyle="-", axis="x")
# minor ticks
if minor:
ax.xaxis.set_minor_formatter(mdates.DateFormatter("%Y %b"))
ax.xaxis.set_minor_locator(mdates.MonthLocator())
ax.grid(which="minor", linestyle=":", axis="x")
# rotate labels
for label in ax.get_xticklabels(which="both"):
label.set(rotation=70, horizontalalignment="right")
def plot_xY(x, Y, ax):
quantiles = Y.quantile((0.025, 0.25, 0.5, 0.75, 0.975), dim=("chain", "draw")).transpose()
az.plot_hdi(
x,
hdi_data=quantiles.sel(quantile=[0.025, 0.975]),
fill_kwargs={"alpha": 0.25},
smooth=False,
ax=ax,
)
az.plot_hdi(
x,
hdi_data=quantiles.sel(quantile=[0.25, 0.75]),
fill_kwargs={"alpha": 0.5},
smooth=False,
ax=ax,
)
ax.plot(x, quantiles.sel(quantile=0.5), color="C1", lw=3)
# default figure sizes
figsize = (10, 5)
# create a list of month strings, for plotting purposes
month_strings = calendar.month_name[1:]
导入数据#
为了我们的目的,我们将获取英格兰和威尔士报告的死亡人数(每月)。这些数据可以从国家统计局的数据集 英格兰和威尔士每月登记的死亡人数 中获得。我手动下载了2006年至2022年的数据,并将其汇总到一个 .csv 文件中。我还添加了作为预测因子的英国月平均气温数据,该数据来自 英国气象局提供的英国平均气温 数据集。
try:
df = pd.read_csv(os.path.join("..", "data", "deaths_and_temps_england_wales.csv"))
except FileNotFoundError:
df = pd.read_csv(pm.get_data("deaths_and_temps_england_wales.csv"))
df["date"] = pd.to_datetime(df["date"])
df = df.set_index("date")
# split into separate dataframes for pre and post onset of COVID-19
pre = df[df.index < "2020"]
post = df[df.index >= "2020"]
可视化数据#
报告的死亡人数随时间变化#
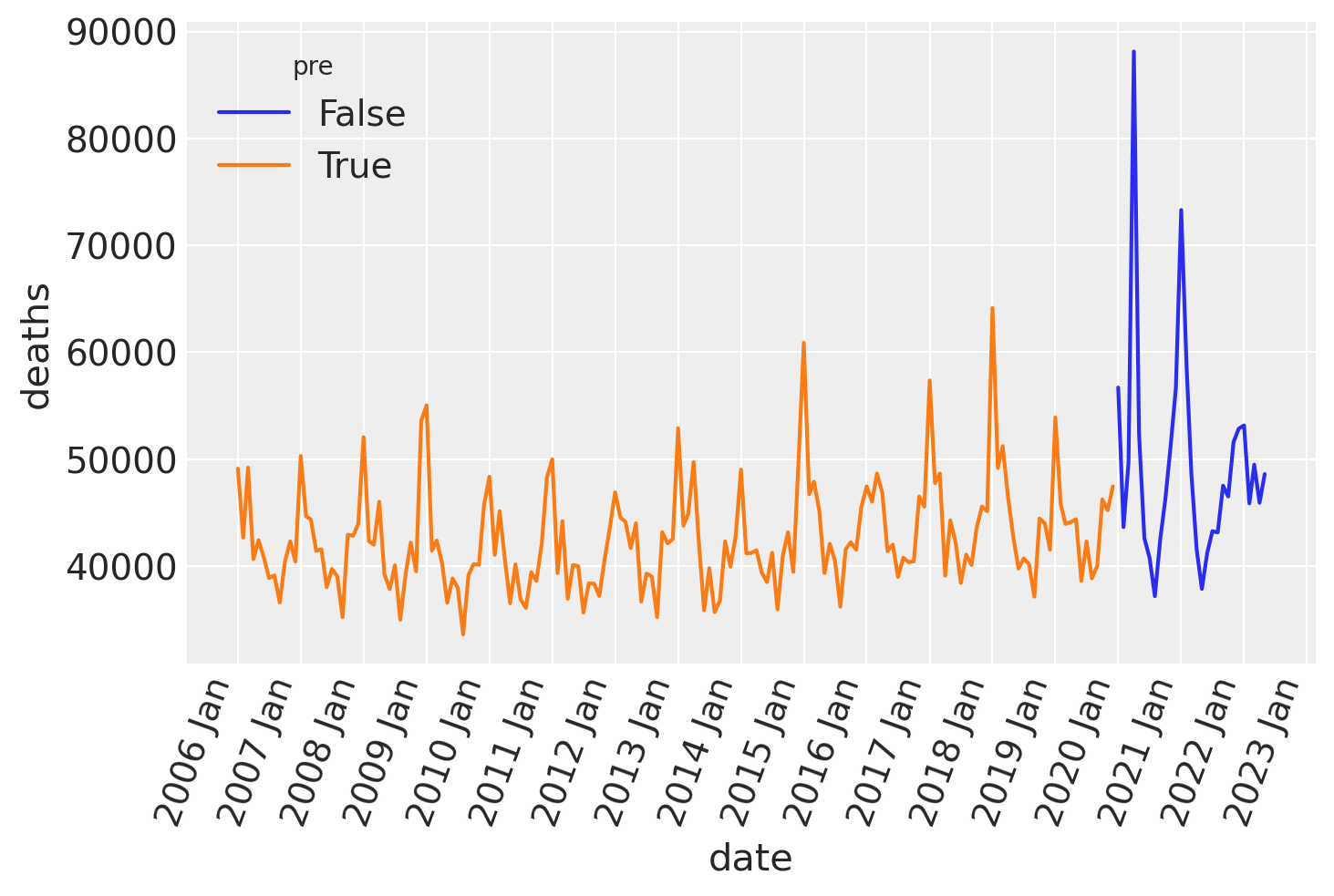
绘制时间序列图显示,死亡人数存在明显的季节性,我们也可以猜测每年平均死亡人数可能有所增加。
ax = sns.lineplot(data=df, x="date", y="deaths", hue="pre")
format_x_axis(ax)
季节性#
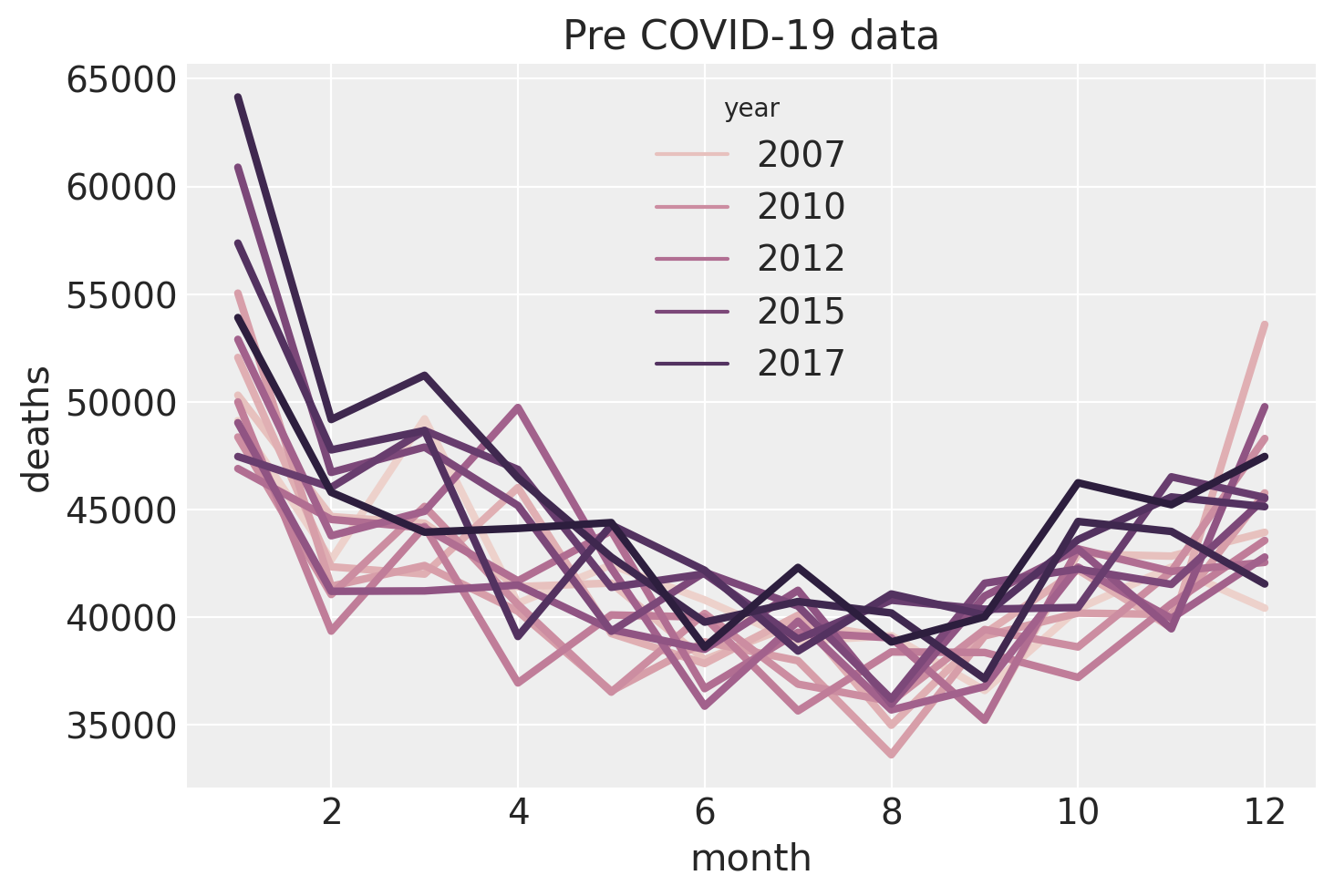
让我们更仔细地观察季节性模式(仅限于疫情前的数据),通过绘制死亡人数与月份的关系图,并用颜色编码年份。这证实了我们对死亡人数存在季节性趋势的怀疑,即冬季的死亡人数多于夏季。我们还可以看到一月份有大量死亡,随后二月份略有下降,三月份又有所回升。这可能是由于以下因素的组合:
push-back将实际上发生在12月的死亡事件推迟到1月登记或者
pull-forward,其中许多原本会在二月死亡的脆弱人群最终在一月死亡,可能是由于寒冷的天气条件。
颜色编码支持了我们的怀疑,即年份有一个积极的主要影响——每年的基线死亡人数正在增加。
ax = sns.lineplot(data=pre, x="month", y="deaths", hue="year", lw=3)
ax.set(title="Pre COVID-19 data");
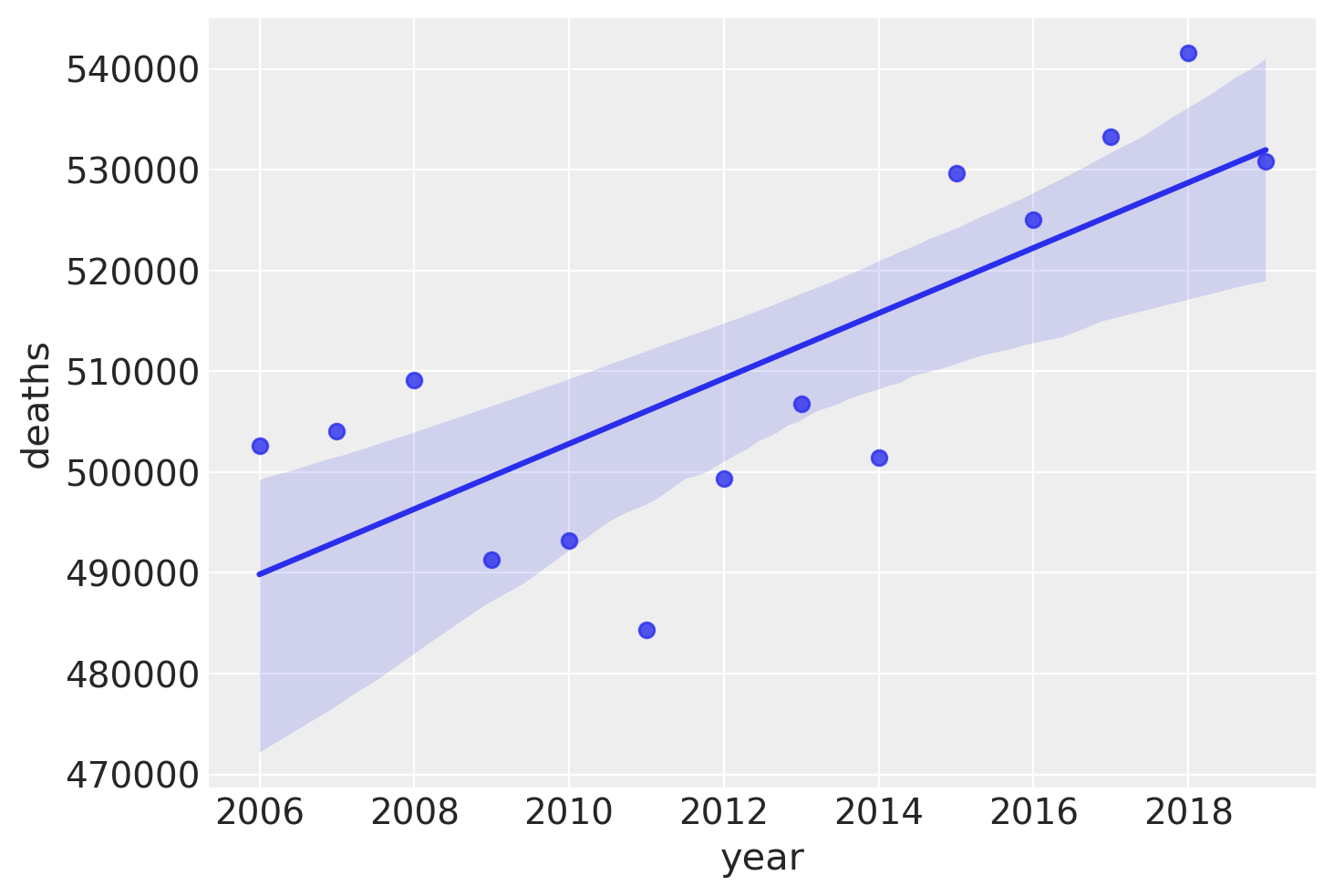
线性趋势#
让我们通过绘制COVID-19之前的总死亡人数随时间的变化来更仔细地观察这一点。虽然这里有一些变化,但似乎添加一个线性趋势作为预测因子将捕捉到报告死亡人数的一些方差,从而使报告死亡人数的模型更好。
annual_deaths = pd.DataFrame(pre.groupby("year")["deaths"].sum()).reset_index()
sns.regplot(x="year", y="deaths", data=annual_deaths);
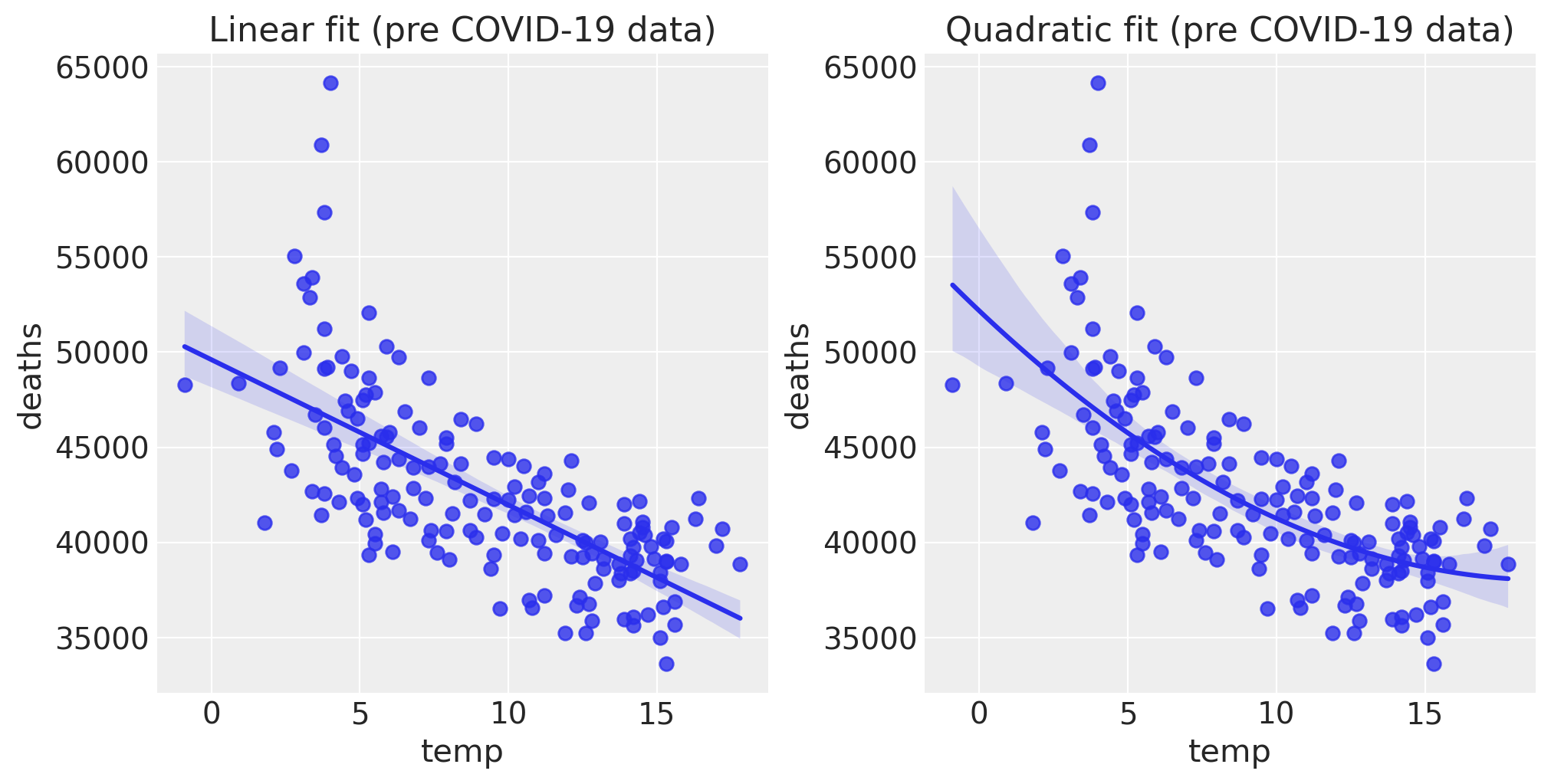
温度对死亡的影响#
仅从pre数据来看,月平均温度与死亡人数之间存在明显的负相关关系。在更广泛的温度范围内,可以清楚地看到死亡人数与温度之间存在U形关系。但在英格兰和威尔士的气候条件下,我们只能看到这条曲线的较低部分。尽管如此,这种关系可能近似于二次关系,但对于我们的目的来说,线性关系似乎是一个合理的起点。
fig, ax = plt.subplots(1, 2, figsize=figsize)
sns.regplot(x="temp", y="deaths", data=pre, scatter_kws={"s": 40}, order=1, ax=ax[0])
ax[0].set(title="Linear fit (pre COVID-19 data)")
sns.regplot(x="temp", y="deaths", data=pre, scatter_kws={"s": 40}, order=2, ax=ax[1])
ax[1].set(title="Quadratic fit (pre COVID-19 data)");
让我们来研究这个关系的斜率,这在我们模型中定义温度系数的先验时会很有用。
# NOTE: results are returned from higher to lower polynomial powers
slope, intercept = np.polyfit(pre["temp"], pre["deaths"], 1)
print(f"{slope:.0f} deaths/degree")
-764 deaths/degree
基于此,如果我们仅关注温度与死亡率之间的关系,我们预计平均每月温度每升高\(1^\circ C\),死亡人数将减少764人。因此,在定义温度效应系数的先验时,我们可以使用这一数据。
建模#
我们将使用一个截距、一个线性趋势、季节性偏差(针对每个月)和平均月温度来估计随时间变化的报告死亡人数。因此,这是一个非常直接的线性模型。唯一值得注意的是,我们将正态分布的月度偏差转换为均值为零,以减少模型的自由度,这应该有助于参数的可识别性。
with pm.Model(coords={"month": month_strings}) as model:
# observed predictors and outcome
month = pm.MutableData("month", pre["month"].to_numpy(), dims="t")
time = pm.MutableData("time", pre["t"].to_numpy(), dims="t")
temp = pm.MutableData("temp", pre["temp"].to_numpy(), dims="t")
deaths = pm.MutableData("deaths", pre["deaths"].to_numpy(), dims="t")
# priors
intercept = pm.Normal("intercept", 40_000, 10_000)
month_mu = ZeroSumNormal("month mu", sigma=3000, dims="month")
linear_trend = pm.TruncatedNormal("linear trend", 0, 50, lower=0)
temp_coeff = pm.Normal("temp coeff", 0, 200)
# the actual linear model
mu = pm.Deterministic(
"mu",
intercept + (linear_trend * time) + month_mu[month - 1] + (temp_coeff * temp),
dims="t",
)
sigma = pm.HalfNormal("sigma", 2_000)
# likelihood
pm.TruncatedNormal("obs", mu=mu, sigma=sigma, lower=0, observed=deaths, dims="t")
pm.model_to_graphviz(model)
先验预测检查#
作为贝叶斯工作流程的一部分,我们将绘制先验预测图,以查看模型在观察到任何数据之前得出的结果。
with model:
idata = pm.sample_prior_predictive(random_seed=RANDOM_SEED)
fig, ax = plt.subplots(figsize=figsize)
plot_xY(pre.index, idata.prior_predictive["obs"], ax)
format_x_axis(ax)
ax.plot(pre.index, pre["deaths"], label="observed")
ax.set(title="Prior predictive distribution in the pre COVID-19 era")
plt.legend();
Sampling: [intercept, linear trend, month mu_raw, obs, sigma, temp coeff]
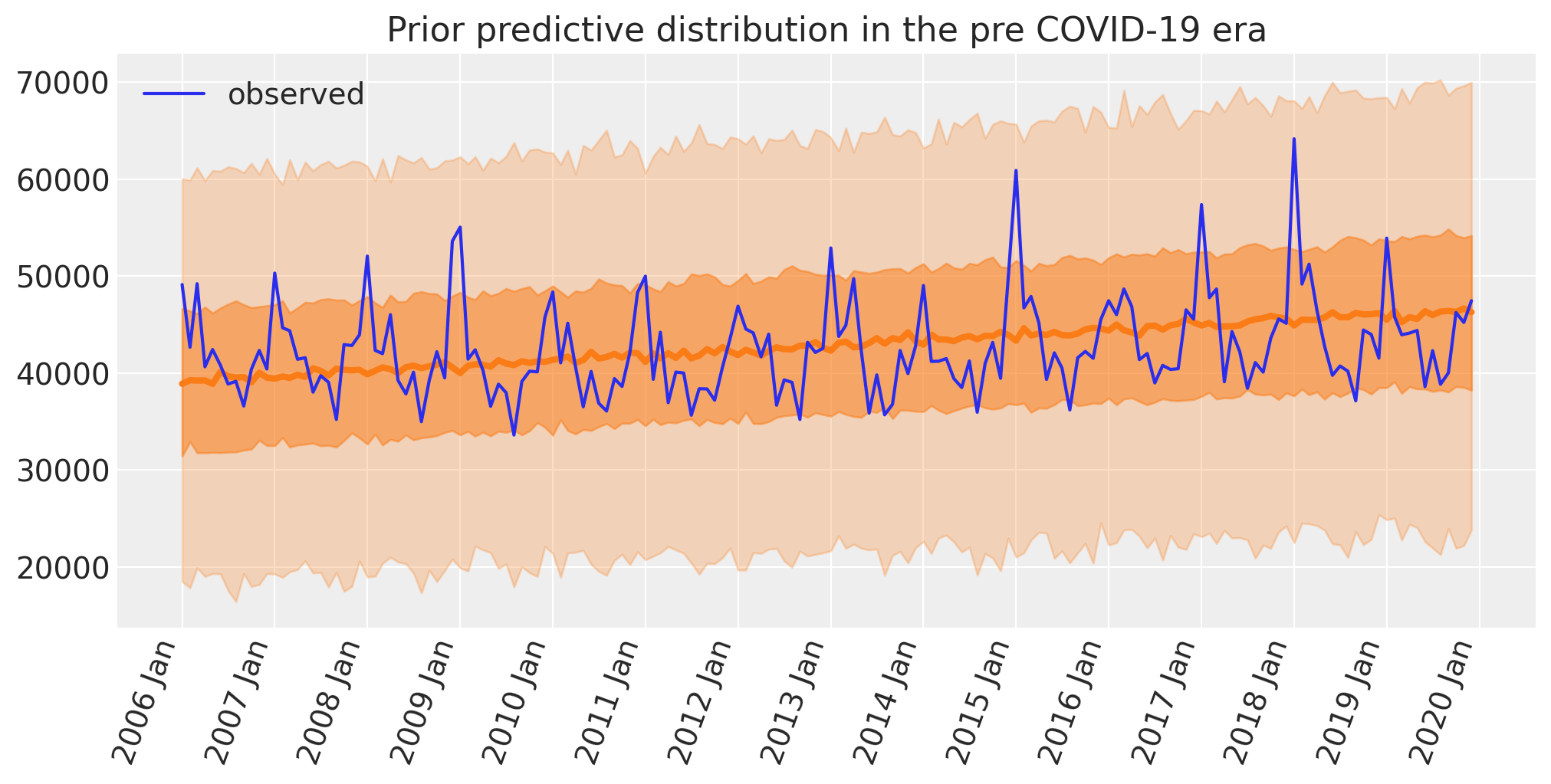
这看起来很合理:
先验的死亡人数看起来集中在观察到的数字上。
给定先验,预测的死亡范围相当广泛,因此不太可能过度约束模型。
该模型不会预测每月死亡人数为负数。
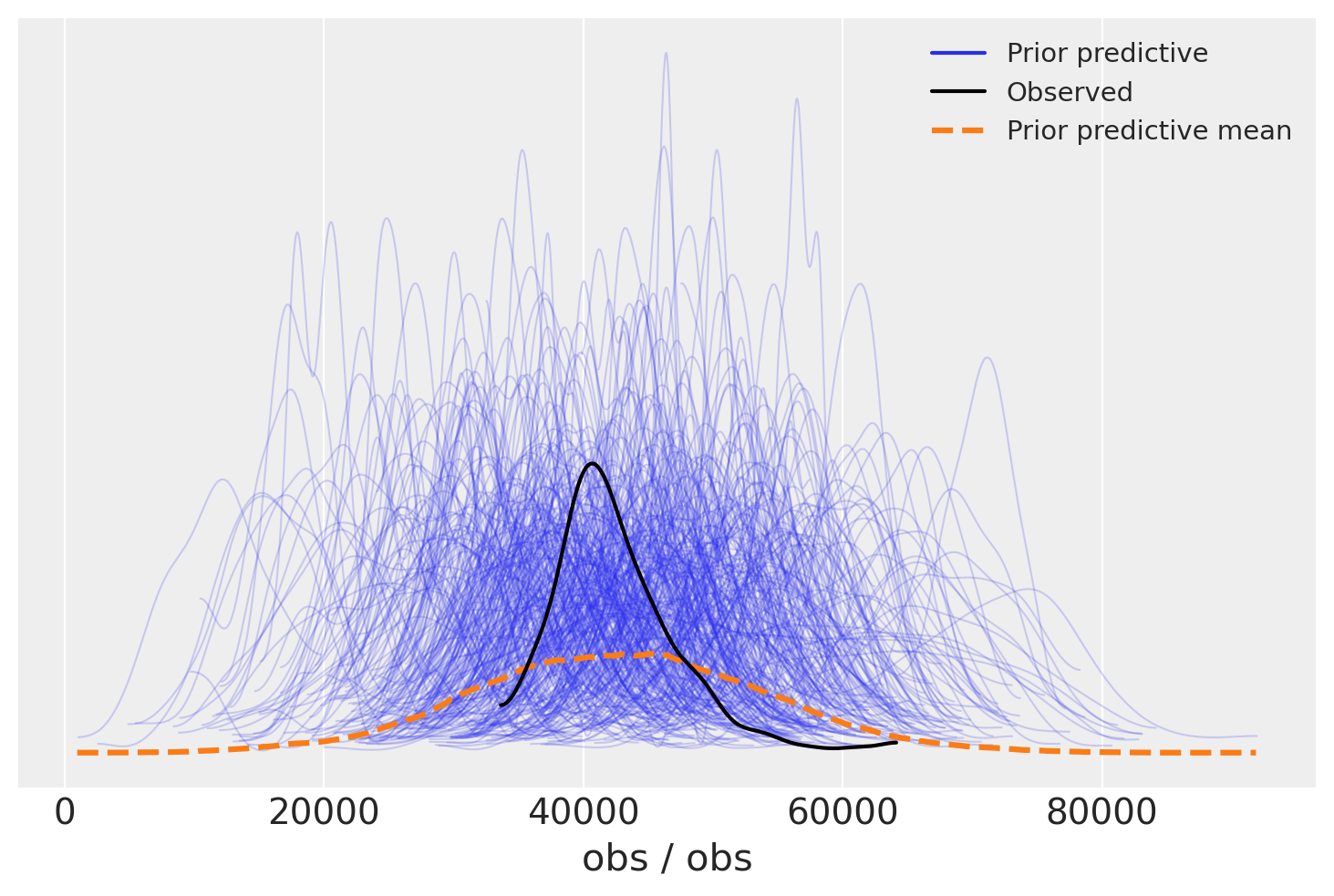
我们可以通过Arviz先验预测检查(ppc)图更详细地查看这一点。再次我们看到观测值的分布以实际观测值为中心,但分布更广。这很有用,因为我们可以知道先验分布不是太严格,不太可能系统地影响我们的后验预测向上或向下。
az.plot_ppc(idata, group="prior");
推理#
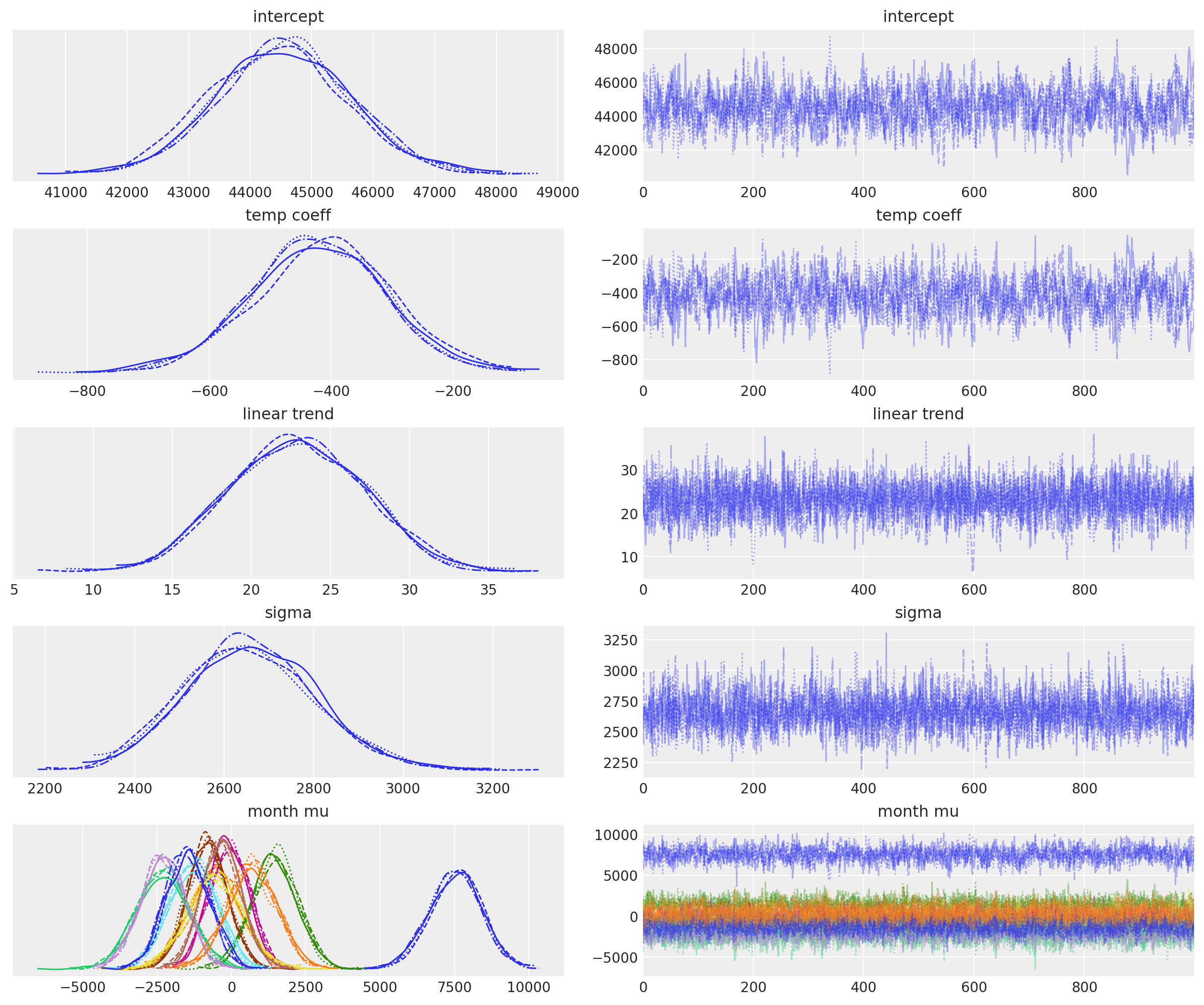
绘制后验分布的样本,并记住我们仅对COVID-19疫情前的数据进行此操作。
with model:
idata.extend(pm.sample(random_seed=RANDOM_SEED))
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [intercept, month mu_raw, linear trend, temp coeff, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 6 seconds.
az.plot_trace(idata, var_names=["~mu", "~month mu_raw"]);
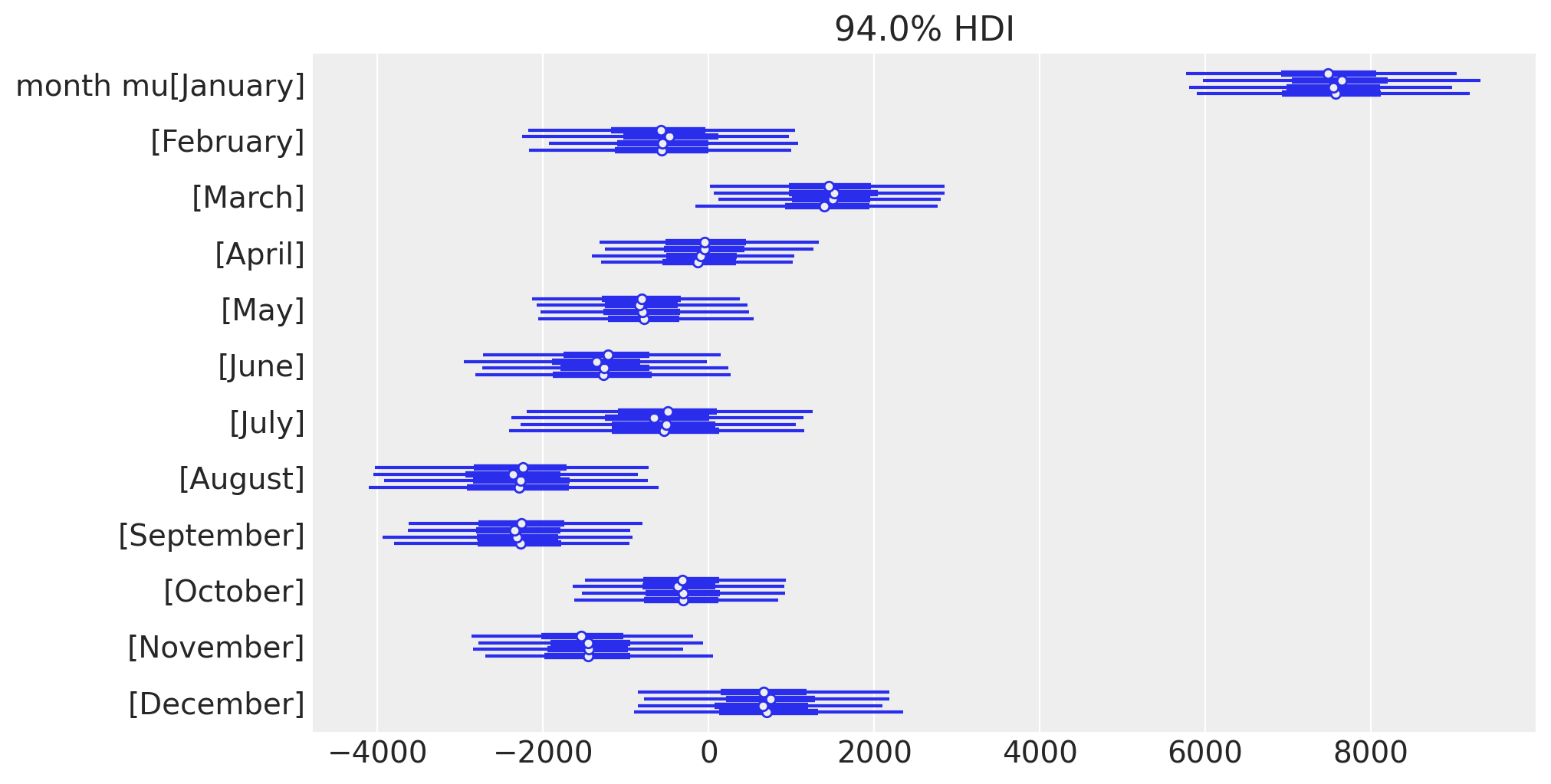
让我们也以一种不同的方式来看待月度偏转的后验估计,以聚焦于季节性效应。
az.plot_forest(idata.posterior, var_names="month mu", figsize=figsize);
后验预测检查#
贝叶斯工作流程的另一个重要方面是绘制模型的后验预测图,使我们能够看到模型对已观察数据的回溯预测效果如何。正是在这一点上,我们可以决定模型是否过于简单(那么我们会在模型中增加更多复杂性)或者是否已经足够好。
with model:
idata.extend(pm.sample_posterior_predictive(idata, random_seed=RANDOM_SEED))
fig, ax = plt.subplots(figsize=figsize)
az.plot_hdi(pre.index, idata.posterior_predictive["obs"], hdi_prob=0.5, smooth=False)
az.plot_hdi(pre.index, idata.posterior_predictive["obs"], hdi_prob=0.95, smooth=False)
ax.plot(pre.index, pre["deaths"], label="observed")
format_x_axis(ax)
ax.set(title="Posterior predictive distribution in the pre COVID-19 era")
plt.legend();
Sampling: [obs]
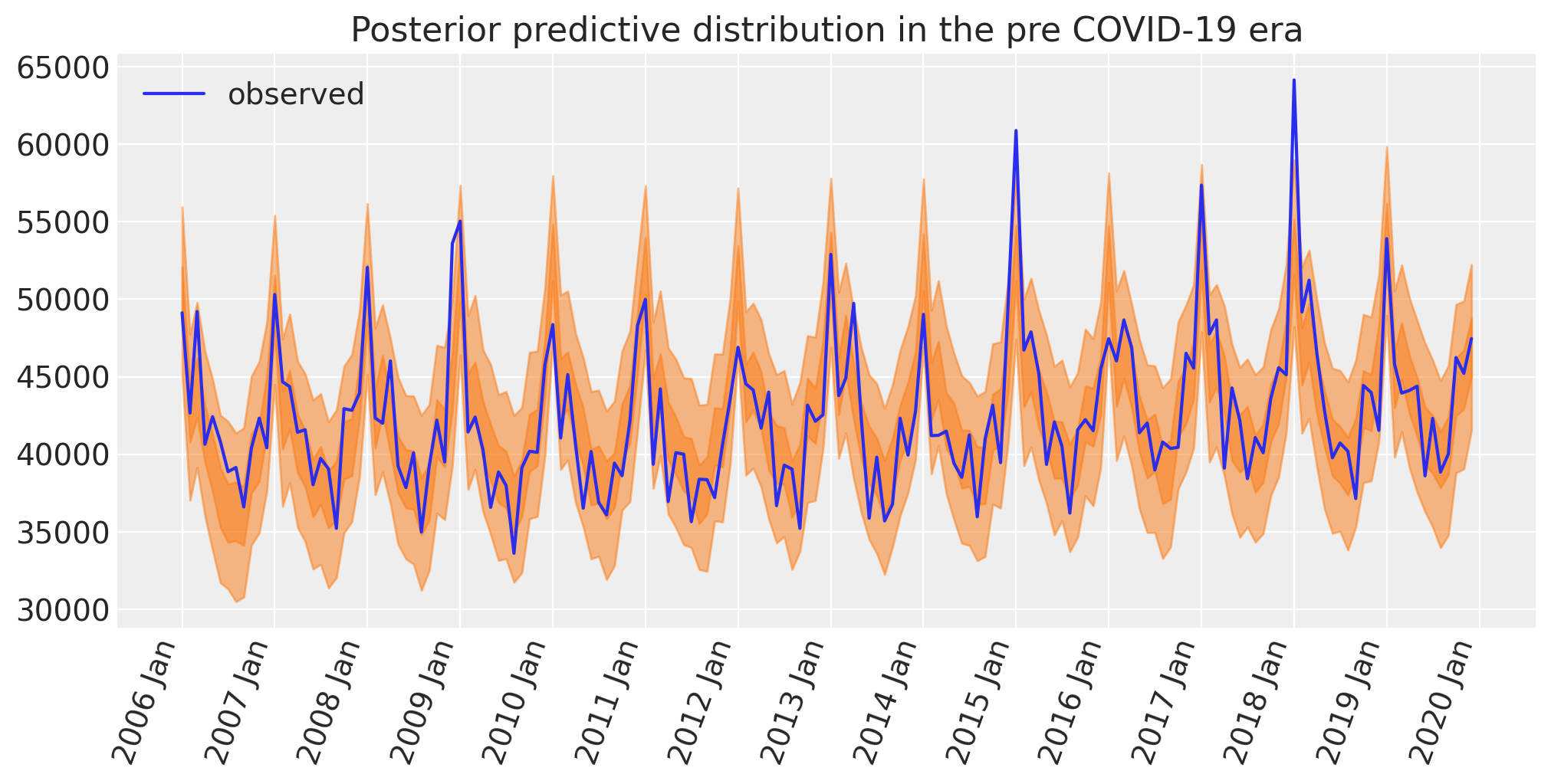
现在让我们再进行一次检查,但这次重点放在季节性效应上。我们将复制上面死亡人数随月份变化的图表。为了不让图表变得一团糟,我们只绘制后验均值。因此,这不是一个后验预测检查,而是一个后验检查。
temp = idata.posterior["mu"].mean(dim=["chain", "draw"]).to_dataframe()
pre = pre.assign(deaths_predicted=temp["mu"].values)
fig, ax = plt.subplots(1, 2, figsize=figsize, sharey=True)
sns.lineplot(data=pre, x="month", y="deaths", hue="year", ax=ax[0], lw=3)
ax[0].set(title="Observed")
sns.lineplot(data=pre, x="month", y="deaths_predicted", hue="year", ax=ax[1], lw=3)
ax[1].set(title="Model predicted mean");
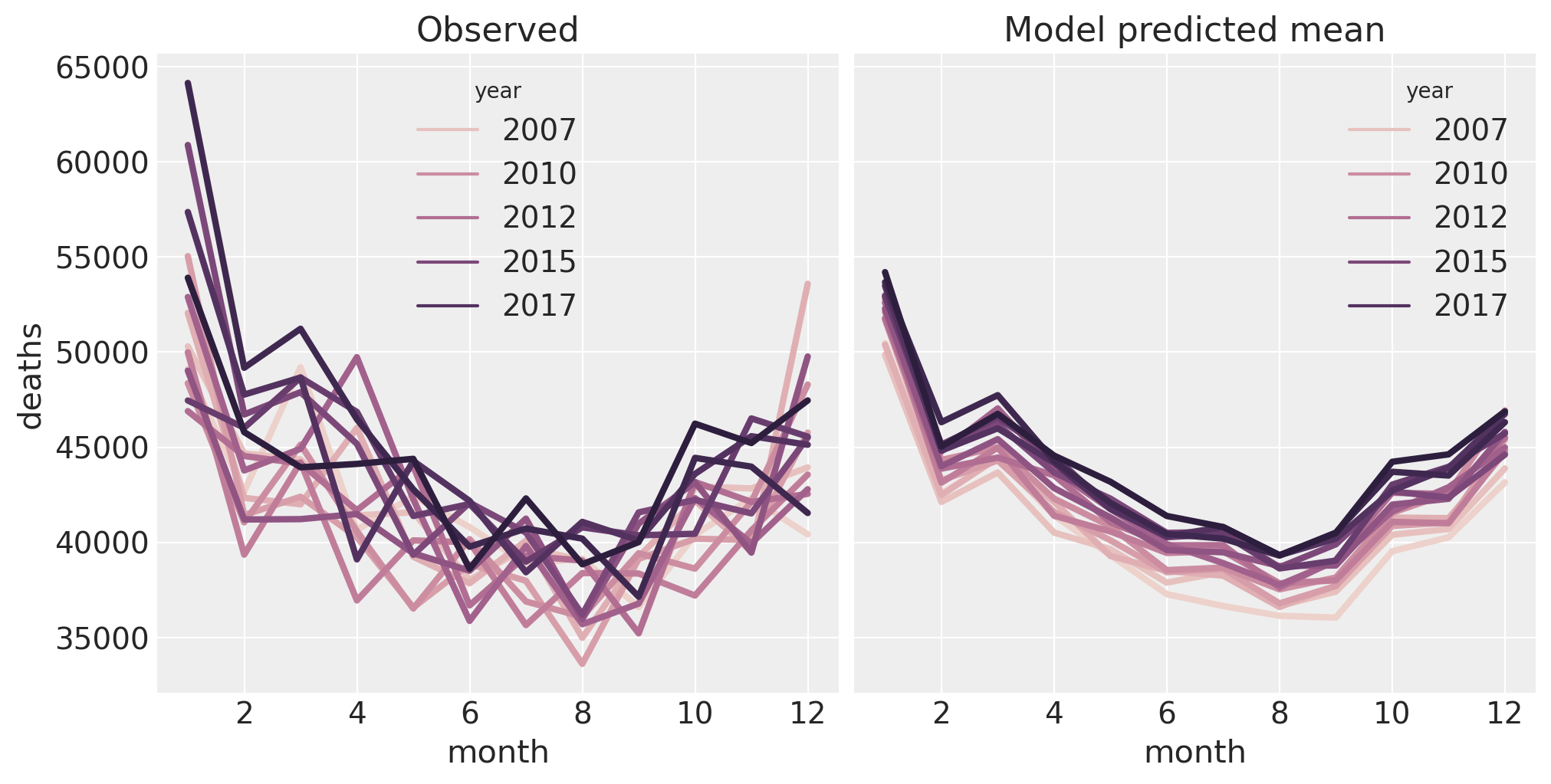
模型在捕捉数据属性方面做得相当不错。在右侧,我们可以清楚地看到月份和年份的主要影响。然而,我们可以看到数据(左侧)在一月份发生了一些有趣的事情,而模型并未捕捉到这一点。这可能可以通过在月份和年份之间添加交互项来捕捉,但这留给读者作为练习。
超额死亡:新冠疫情前#
这一步并不是严格必要的,但我们可以将超额死亡公式应用于模型对pre时期的回溯预测。这是有用的,因为我们可以检查模型的表现如何。
Show code cell source
# convert deaths into an XArray object with a labelled dimension to help in the next step
deaths = xr.DataArray(pre["deaths"].to_numpy(), dims=["t"])
# do the calculation by taking the difference
excess_deaths = deaths - idata.posterior_predictive["obs"]
fig, ax = plt.subplots(figsize=figsize)
# the transpose is to keep arviz happy, ordering the dimensions as (chain, draw, t)
az.plot_hdi(pre.index, excess_deaths.transpose(..., "t"), hdi_prob=0.5, smooth=False)
az.plot_hdi(pre.index, excess_deaths.transpose(..., "t"), hdi_prob=0.95, smooth=False)
format_x_axis(ax)
ax.axhline(y=0, color="k")
ax.set(title="Excess deaths, pre COVID-19");
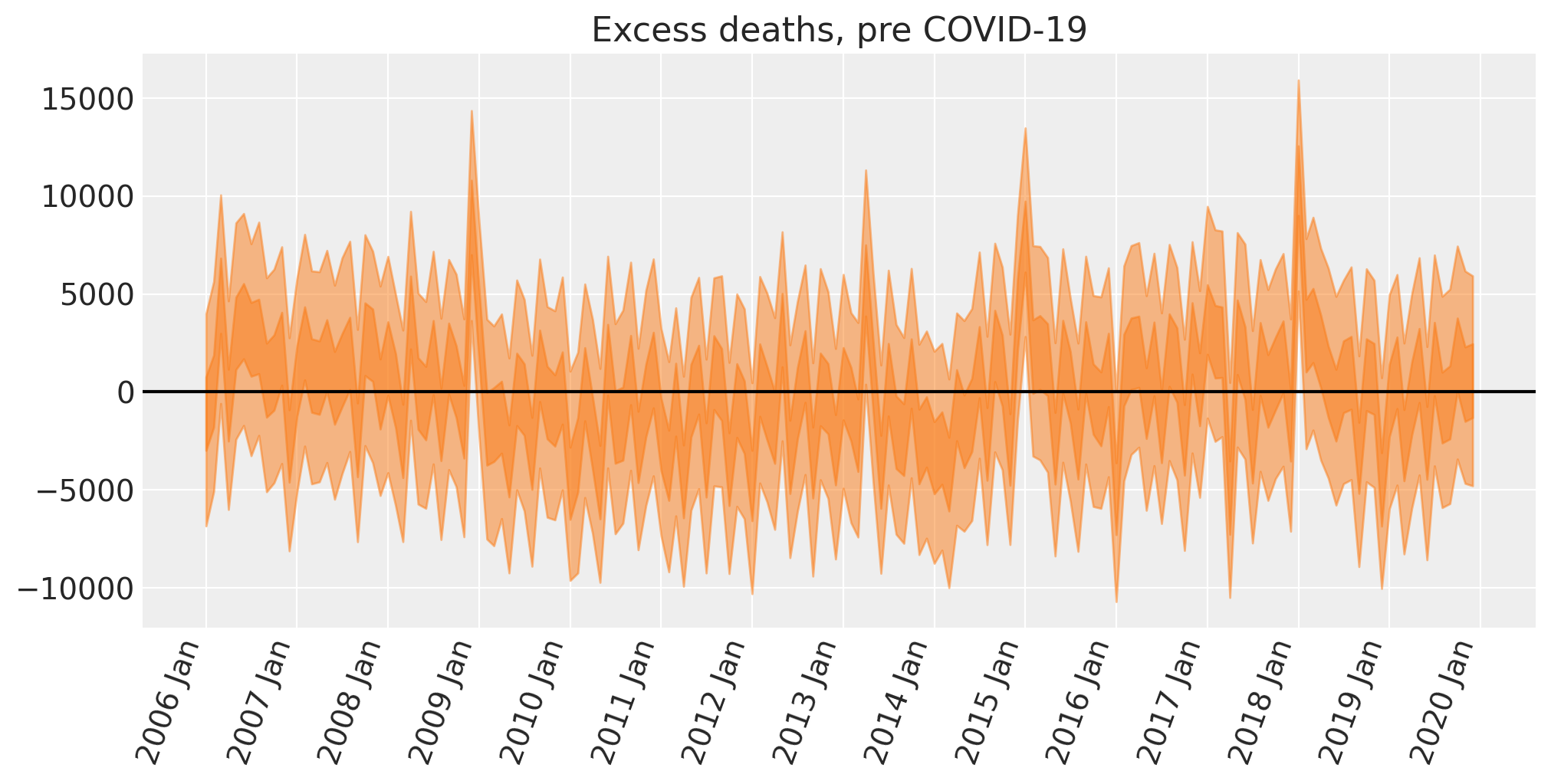
我们可以看到这里有一些峰值,超额死亡人数可能大于零。这些情况超出了我们可以从以下因素中预期的范围:a) 季节性影响,b) 线性增长趋势,c) 寒冷冬季的影响。
如果我们感兴趣,那么我们可以开始生成关于哪些额外的预测因子可能解释这一现象的假设。一些想法可能包括普通感冒的流行程度,或最低月温度,这些可能提供平均值未捕捉到的额外预测信息。
我们还可以看到,模型并没有完全捕捉到一些额外的时序趋势。从后验均值来看,存在一些系统性的低频漂移偏离零点。也就是说,数据中存在我们的预测因子未能完全捕捉到的额外方差,这可能是由于易感人群规模随时间变化所导致的。
但我们已经接近在COVID-19期间计算超额死亡的目标,因此我们将继续前进,因为这里的主要目的是进行反事实思考,而不是构建有史以来最全面的报告死亡模型。
反事实推理#
现在我们将使用我们的模型来预测在“假设”情景下,即在正常业务情况下,报告的死亡人数。
因此,我们使用month和时间(t)以及temp数据从post数据框更新模型,并运行后验预测采样以预测在此反事实情景中我们将观察到的报告死亡人数。我们也可以称之为“预测”。
with model:
pm.set_data(
{
"month": post["month"].to_numpy(),
"time": post["t"].to_numpy(),
"temp": post["temp"].to_numpy(),
}
)
counterfactual = pm.sample_posterior_predictive(
idata, var_names=["obs"], random_seed=RANDOM_SEED
)
Sampling: [obs]
Show code cell source
fig, ax = plt.subplots(figsize=figsize)
plot_xY(post.index, counterfactual.posterior_predictive["obs"], ax)
format_x_axis(ax, minor=True)
ax.plot(post.index, post["deaths"], label="reported deaths")
ax.set(title="Counterfactual: Posterior predictive forecast of deaths if COVID-19 had not appeared")
plt.legend();
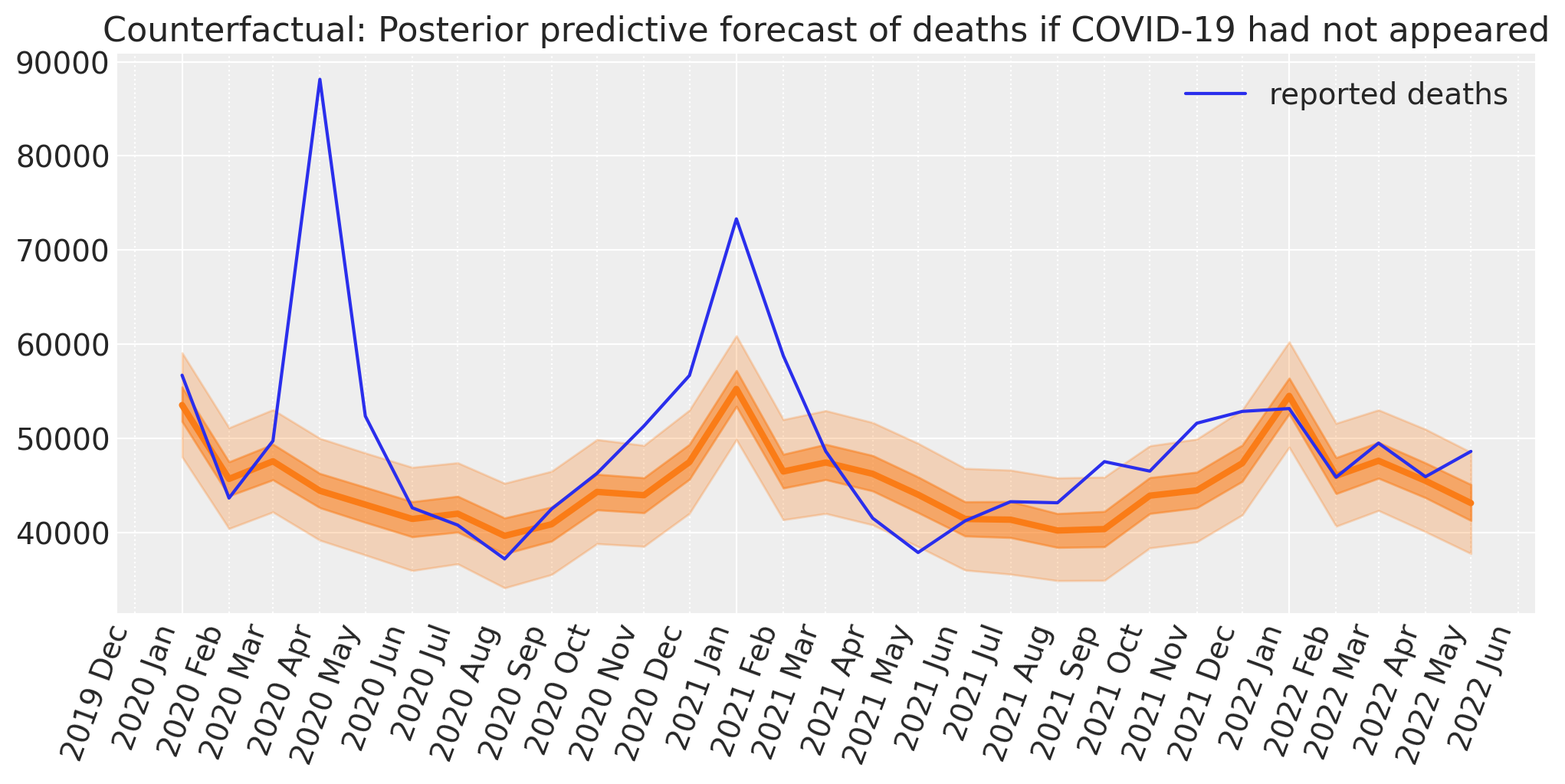
我们现在有了计算超额死亡所需的要素。即报告的死亡人数,以及贝叶斯反事实预测,即如果从新冠疫情前到疫情后没有任何变化,会有多少人死亡。
超额死亡:自新冠疫情开始#
现在我们将使用在反事实情景下预测的死亡人数,并将其与报告的死亡人数进行比较,以得出我们对超额死亡的反事实估计。
# convert deaths into an XArray object with a labelled dimension to help in the next step
deaths = xr.DataArray(post["deaths"].to_numpy(), dims=["t"])
# do the calculation by taking the difference
excess_deaths = deaths - counterfactual.posterior_predictive["obs"]
我们可以轻松计算累计超额死亡人数
# calculate the cumulative excess deaths
cumsum = excess_deaths.cumsum(dim="t")
Show code cell source
fig, ax = plt.subplots(2, 1, figsize=(figsize[0], 9), sharex=True)
# Plot the excess deaths
# The transpose is to keep arviz happy, ordering the dimensions as (chain, draw, t)
plot_xY(post.index, excess_deaths.transpose(..., "t"), ax[0])
format_x_axis(ax[0], minor=True)
ax[0].axhline(y=0, color="k")
ax[0].set(title="Excess deaths, since COVID-19 onset")
# Plot the cumulative excess deaths
plot_xY(post.index, cumsum.transpose(..., "t"), ax[1])
format_x_axis(ax[1], minor=True)
ax[1].axhline(y=0, color="k")
ax[1].set(title="Cumulative excess deaths, since COVID-19 onset");
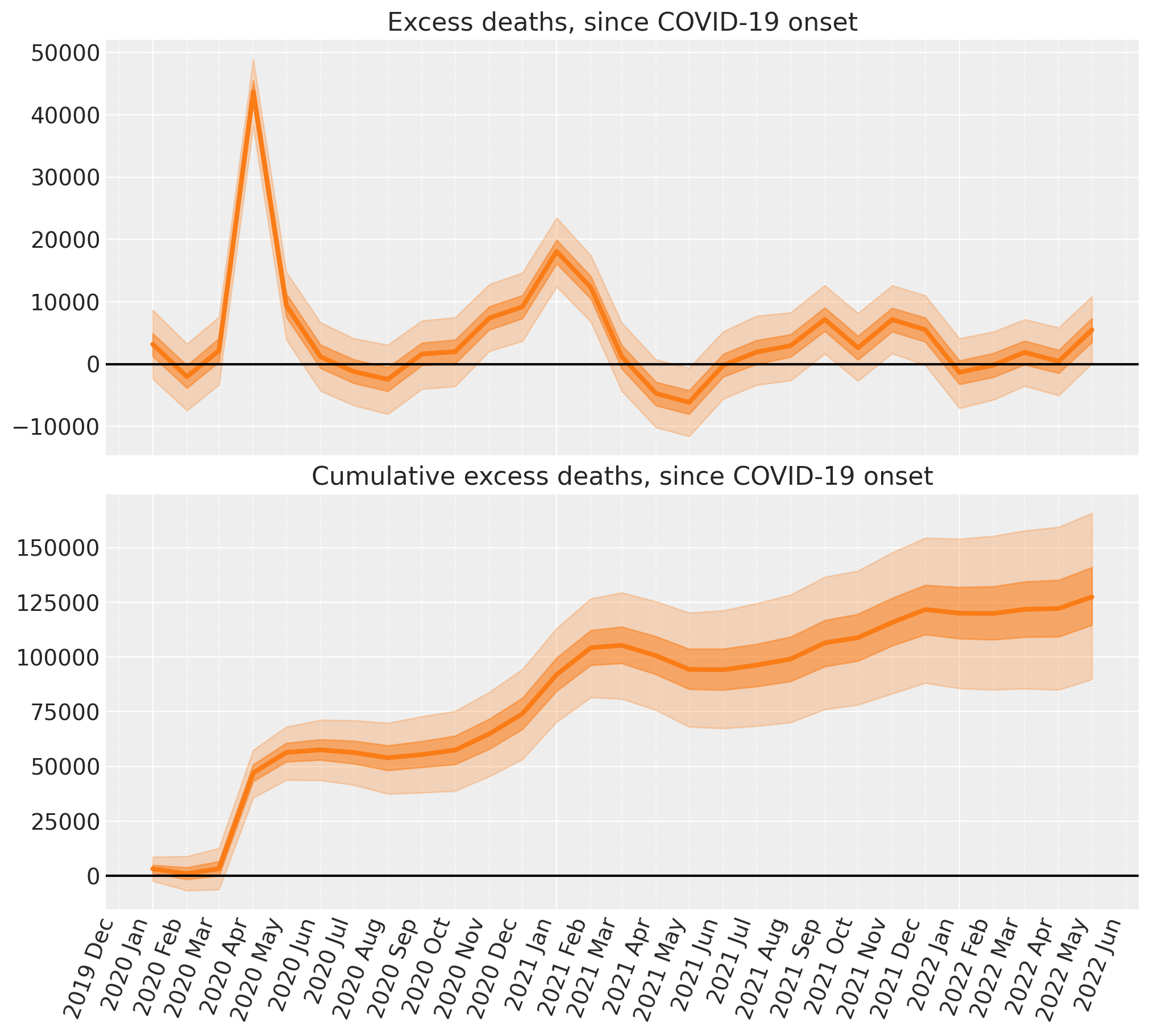
就这样,我们在PyMC中完成了一些贝叶斯反事实推理!在短短几个步骤中,我们:
构建了一个简单的线性回归模型。
基于疫情前数据推断模型参数,运行先验和后验预测检查。我们注意到模型相当不错,但一如既往,未来可能会有改进模型的方法。
使用模型创建了反事实预测,预测如果没有任何改变,未来(COVID-19时期)会发生什么。
通过将报告的死亡人数与我们的反事实预期死亡人数进行比较,计算了超额死亡人数(以及累计超额死亡人数)。
当然,坏消息是,截至最后一个数据点(2022年5月),英格兰和威尔士的超额死亡人数又开始上升了。
参考资料#
Kay H. Brodersen, Fabian Gallusser, Jim Koehler, Nicolas Remy, 和 Steven L. Scott. 使用贝叶斯结构时间序列模型推断因果影响。应用统计年鉴,9:247–274, 2015.
Osvaldo A Martin, Ravin Kumar, 和 Junpeng Lao. Python中的贝叶斯建模与计算. Chapman and Hall/CRC, 2021. doi:10.1201/9781003019169.
水印#
%load_ext watermark
%watermark -n -u -v -iv -w -p pytensor,aeppl,xarray
Last updated: Wed Feb 01 2023
Python implementation: CPython
Python version : 3.11.0
IPython version : 8.9.0
pytensor: 2.8.11
aeppl : not installed
xarray : 2023.1.0
pymc : 5.0.1
arviz : 0.14.0
matplotlib: 3.6.3
pandas : 1.5.3
numpy : 1.24.1
xarray : 2023.1.0
seaborn : 0.12.2
pytensor : 2.8.11
Watermark: 2.3.1