多层次建模贝叶斯方法入门#
层次或分层建模是回归建模的泛化。
多层次模型是回归模型,其中构成模型的参数被赋予概率模型。这意味着模型参数可以按组变化。
观察单位通常是自然聚集的。尽管对群组进行随机抽样并在群组内进行随机抽样,但聚集会导致观察结果之间的依赖性。
一个层次模型是一种特定的多层次模型,其中参数相互嵌套。
一些多级结构并不是层次化的。
例如,“country”和“year”不是嵌套的,但可能代表独立的、但重叠的参数集群
我们将使用一个环境流行病学的例子来激发这个话题。
示例:氡污染 Gelman 和 Hill [2006]#
氡是一种放射性气体,通过与地面的接触点进入家庭。它是一种致癌物质,是非吸烟者肺癌的主要原因。氡水平在家庭之间差异很大。
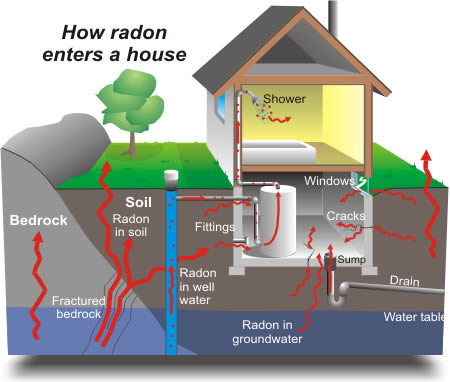
美国环保署对80,000所房屋的氡水平进行了研究。有两个重要的预测因素:
在地下室或一楼进行测量(地下室的氡气较高)
县铀含量水平(与氡水平呈正相关)
我们将重点关注明尼苏达州的氡气水平建模。
此示例中的层次结构是县内的家庭。
数据组织#
首先,我们从本地文件导入数据,并提取明尼苏达州的数据。
import os
import warnings
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import pymc as pm
import pytensor.tensor as pt
import seaborn as sns
import xarray as xr
warnings.filterwarnings("ignore", module="scipy")
print(f"Running on PyMC v{pm.__version__}")
Running on PyMC v4.4.0
RANDOM_SEED = 8924
az.style.use("arviz-darkgrid")
原始数据存在于几个独立的数据集中,我们将在这里导入、合并和处理这些数据。首先是来自美国各地各个家庭的测量数据。我们将提取明尼苏达州的子集。
try:
srrs2 = pd.read_csv(os.path.join("..", "data", "srrs2.dat"))
except FileNotFoundError:
srrs2 = pd.read_csv(pm.get_data("srrs2.dat"))
srrs2.columns = srrs2.columns.map(str.strip)
srrs_mn = srrs2[srrs2.state == "MN"].copy()
接下来,通过组合两个变量来获取县级预测变量,铀。
try:
cty = pd.read_csv(os.path.join("..", "data", "cty.dat"))
except FileNotFoundError:
cty = pd.read_csv(pm.get_data("cty.dat"))
srrs_mn["fips"] = srrs_mn.stfips * 1000 + srrs_mn.cntyfips
cty_mn = cty[cty.st == "MN"].copy()
cty_mn["fips"] = 1000 * cty_mn.stfips + cty_mn.ctfips
使用 merge 方法将家庭和县级的信息合并到一个DataFrame中。
srrs_mn = srrs_mn.merge(cty_mn[["fips", "Uppm"]], on="fips")
srrs_mn = srrs_mn.drop_duplicates(subset="idnum")
u = np.log(srrs_mn.Uppm).unique()
n = len(srrs_mn)
让我们对县名进行编码,并制作我们将使用的变量的本地副本。
我们还需要一个查找表(dict)用于每个唯一的县,以便进行索引。
srrs_mn.county = srrs_mn.county.map(str.strip)
county, mn_counties = srrs_mn.county.factorize()
srrs_mn["county_code"] = county
radon = srrs_mn.activity
srrs_mn["log_radon"] = log_radon = np.log(radon + 0.1).values
floor_measure = srrs_mn.floor.values
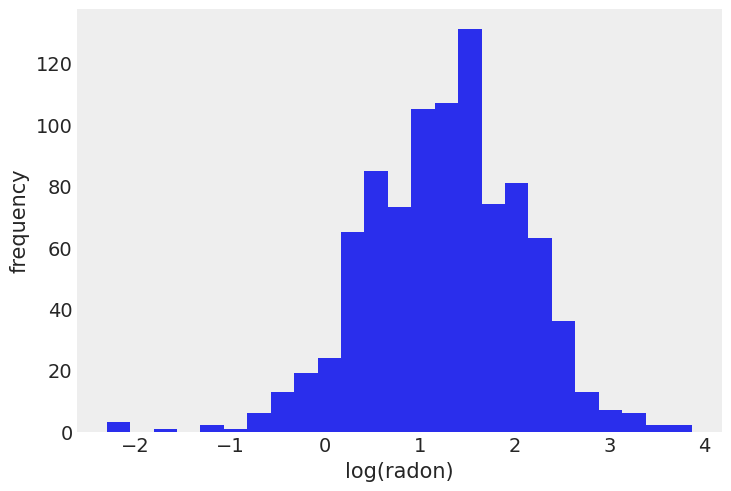
明尼苏达州氡水平分布(对数尺度):
srrs_mn.log_radon.hist(bins=25, grid=False)
plt.xlabel("log(radon)")
plt.ylabel("frequency");
传统方法#
建模氡暴露的两种传统替代方法代表了偏差-方差权衡的两个极端:
完全池化:
将所有县视为相同,并估计一个单一的氡水平。
无池化:
在每个县独立建模氡。
其中 \(j = 1,\ldots,85\)
误差 \(\epsilon_i\) 可能代表测量误差、房屋内的时变差异或房屋间的差异。
以下是完全池化模型的斜率和截距的点估计值:
with pm.Model() as pooled_model:
floor_ind = pm.MutableData("floor_ind", floor_measure, dims="obs_id")
alpha = pm.Normal("alpha", 0, sigma=10)
beta = pm.Normal("beta", mu=0, sigma=10)
sigma = pm.Exponential("sigma", 5)
theta = alpha + beta * floor_ind
y = pm.Normal("y", theta, sigma=sigma, observed=log_radon, dims="obs_id")
pm.model_to_graphviz(pooled_model)
您可能会好奇,尽管变量 floor_ind 既不是观测变量也不是模型的参数,为什么我们还要使用 pm.Data 容器。正如您将看到的,这将使我们在绘制和诊断模型时的生活变得更加轻松。ArviZ 会将 floor_ind 作为变量包含在结果 InferenceData 对象的 constant_data 组中。此外,将 floor_ind 包含在 InferenceData 对象中,使得共享和复现分析变得更加容易,所有用于分析或重新运行模型所需的数据都存储在那里。
在运行模型之前,我们先进行一些先验预测检查。
事实上,拥有合理的先验不仅是一种将科学知识融入模型的方式,它还可以帮助并使MCMC机制更快——这里我们处理的是一个简单的线性回归,因此没有链接函数来扭曲结果空间;但总有一天这种情况会发生在你身上,你需要仔细思考你的先验以帮助你的MCMC采样器。所以,最好在事情相对简单的时候训练自己,而不是在非常困难的时候才去学习。
在PyMC中有一个方便的函数用于先验预测采样:
with pooled_model:
prior_checks = pm.sample_prior_predictive(random_seed=RANDOM_SEED)
Sampling: [alpha, beta, sigma, y]
ArviZ InferenceData 在底层使用了 xarray.Dataset,这使得我们可以通过 .plot 访问几种常见的绘图函数。在这种情况下,我们希望绘制每个考虑的两个水平下的平均对数氡水平的散点图(存储在变量 a 中)。如果我们的期望绘图受 xarray 绘图功能的支持,我们可以利用 xarray 来自动生成绘图和标签。请注意,所有内容都是直接绘制和注释的,我们唯一需要做的更改是将 y 轴标签从 a 重命名为 Mean log radon level。
prior = prior_checks.prior.squeeze(drop=True)
xr.concat((prior["alpha"], prior["alpha"] + prior["beta"]), dim="location").rename(
"log_radon"
).assign_coords(location=["basement", "floor"]).plot.scatter(
x="location", y="log_radon", edgecolors="none"
);

我不是氡专家,但在看到数据之前,这些先验似乎允许平均对数氡水平的范围相当广泛,无论是在地下室还是在楼层测量。但别担心,如果采样给我们提示这些先验可能不合适,我们总是可以改变它们——毕竟,先验是假设,不是誓言;而且与大多数假设一样,它们可以被检验。
然而,我们已经可以想到一种改进:记住我们提到氡水平往往在地下室较高,因此我们可以通过强制楼层效应(beta)为负来将这一先验科学知识纳入我们的模型中。目前,我们将保持模型不变,并相信数据中的信息将足够充分。
说到采样,让我们启动贝叶斯机器吧!
with pooled_model:
pooled_trace = pm.sample(random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [alpha, beta, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 4 seconds.
没有分歧,采样只用了几秒钟!这里的链看起来非常好(良好的R帽,良好的有效样本大小,小的标准差)。模型还估计了一个负的地板效应,正如我们所预期的。
az.summary(pooled_trace, round_to=2)
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| alpha | 1.36 | 0.03 | 1.31 | 1.42 | 0.0 | 0.0 | 4200.11 | 3082.69 | 1.0 |
| beta | -0.59 | 0.07 | -0.73 | -0.46 | 0.0 | 0.0 | 3944.89 | 3146.38 | 1.0 |
| sigma | 0.79 | 0.02 | 0.75 | 0.83 | 0.0 | 0.0 | 4816.78 | 3116.53 | 1.0 |
让我们绘制地下室预期氡水平(alpha)和楼层预期氡水平(alpha + beta)与用于拟合模型的数据的关系:
post_mean = pooled_trace.posterior.mean(dim=("chain", "draw"))
plt.scatter(srrs_mn.floor, np.log(srrs_mn.activity + 0.1))
xvals = xr.DataArray(np.linspace(-0.2, 1.2))
plt.plot(xvals, post_mean["beta"] * xvals + post_mean["alpha"], "r--");
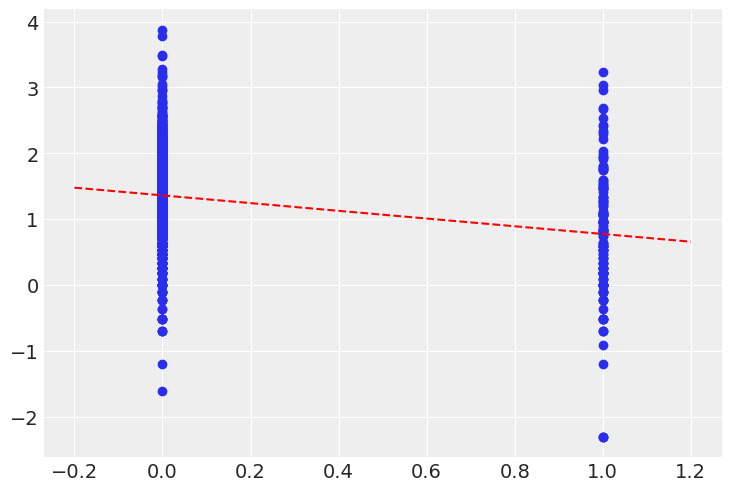
这看起来是合理的,尽管注意到数据中存在大量的残差变异性。
现在让我们将注意力转向未合并的模型,并看看它在比较中的表现如何。
coords = {"county": mn_counties}
with pm.Model(coords=coords) as unpooled_model:
floor_ind = pm.MutableData("floor_ind", floor_measure, dims="obs_id")
alpha = pm.Normal("alpha", 0, sigma=10, dims="county")
beta = pm.Normal("beta", 0, sigma=10)
sigma = pm.Exponential("sigma", 1)
theta = alpha[county] + beta * floor_ind
y = pm.Normal("y", theta, sigma=sigma, observed=log_radon, dims="obs_id")
pm.model_to_graphviz(unpooled_model)
with unpooled_model:
unpooled_trace = pm.sample(random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [alpha, beta, sigma]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 7 seconds.
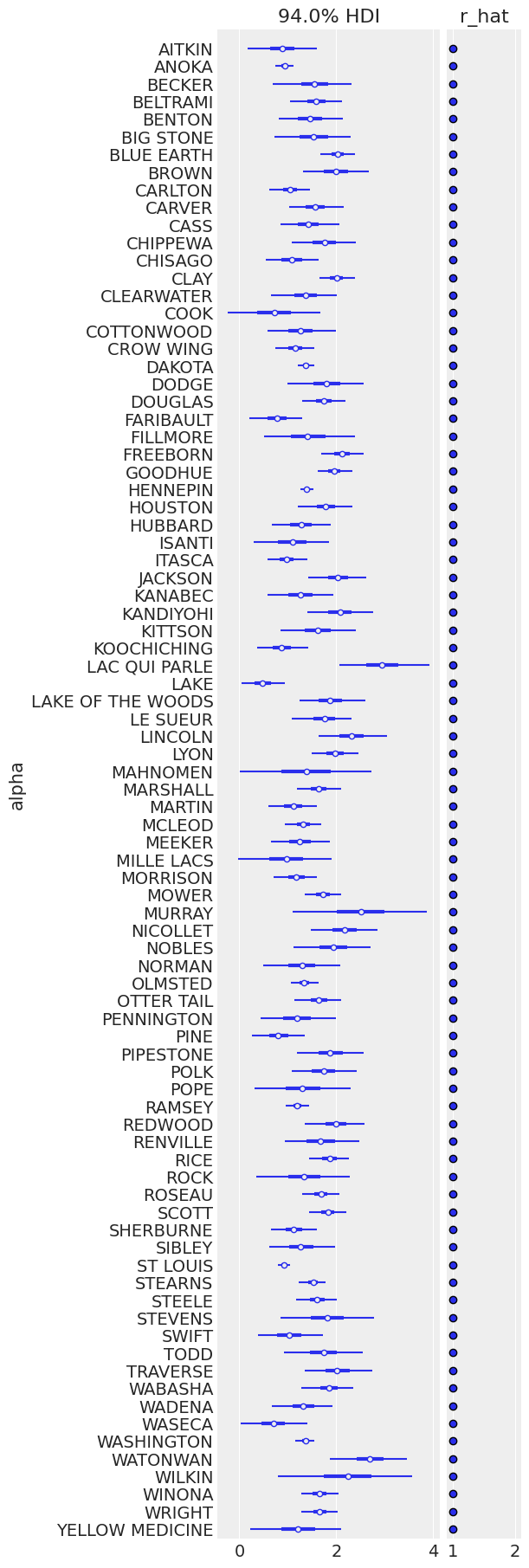
这里的采样也很干净;让我们看看每个县地下室(维度0)和地面(维度1)的期望值:
ax = az.plot_forest(
unpooled_trace,
var_names=["alpha"],
r_hat=True,
combined=True,
figsize=(6, 18),
labeller=az.labels.NoVarLabeller(),
)
ax[0].set_ylabel("alpha");
为了识别高氡水平的县,我们可以绘制有序的平均估计值,以及它们的94% HPD:
unpooled_means = unpooled_trace.posterior.mean(dim=("chain", "draw"))
unpooled_hdi = az.hdi(unpooled_trace)
unpooled_means_iter = unpooled_means.sortby("alpha")
unpooled_hdi_iter = unpooled_hdi.sortby(unpooled_means_iter.alpha)
_, ax = plt.subplots(figsize=(12, 5))
xticks = np.arange(0, 86, 6)
unpooled_means_iter.plot.scatter(x="county", y="alpha", ax=ax, alpha=0.8)
ax.vlines(
np.arange(mn_counties.size),
unpooled_hdi_iter.alpha.sel(hdi="lower"),
unpooled_hdi_iter.alpha.sel(hdi="higher"),
color="orange",
alpha=0.6,
)
ax.set(ylabel="Radon estimate", ylim=(-2, 4.5))
ax.set_xticks(xticks)
ax.set_xticklabels(unpooled_means_iter.county.values[xticks])
ax.tick_params(rotation=90);
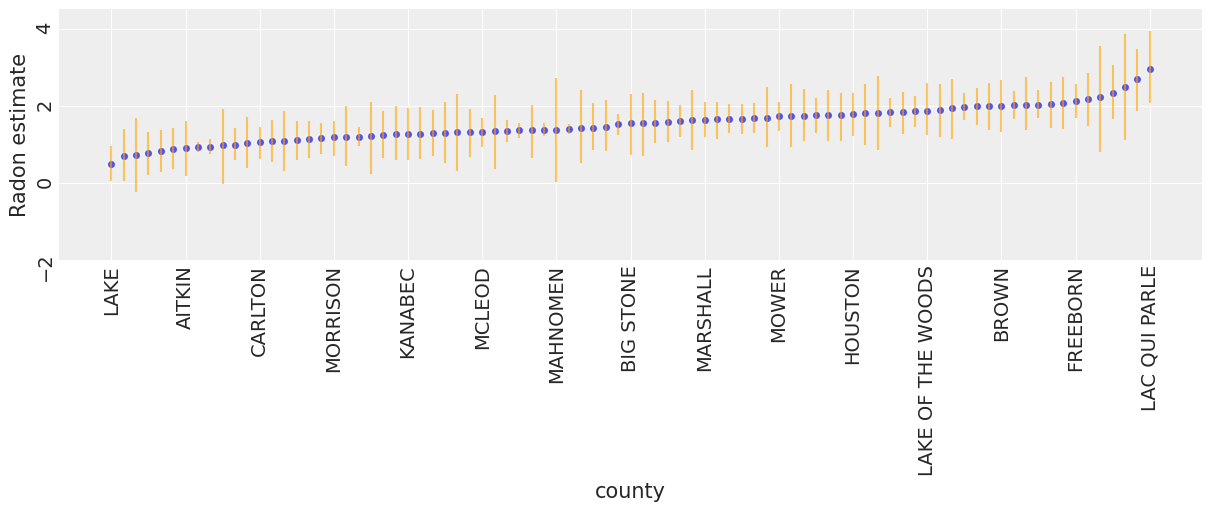
现在我们已经拟合了传统的(即非层次的)模型,让我们看看它们的推论有何不同。以下是汇集估计和非汇集估计在代表不同样本量的一组县中的可视化比较。
sample_counties = (
"LAC QUI PARLE",
"AITKIN",
"KOOCHICHING",
"DOUGLAS",
"CLAY",
"STEARNS",
"RAMSEY",
"ST LOUIS",
)
fig, axes = plt.subplots(2, 4, figsize=(12, 6), sharey=True, sharex=True)
axes = axes.ravel()
m = unpooled_means["beta"]
for i, c in enumerate(sample_counties):
y = srrs_mn.log_radon[srrs_mn.county == c]
x = srrs_mn.floor[srrs_mn.county == c]
axes[i].scatter(x + np.random.randn(len(x)) * 0.01, y, alpha=0.4)
# No pooling model
b = unpooled_means["alpha"].sel(county=c)
# Plot both models and data
xvals = xr.DataArray(np.linspace(0, 1))
axes[i].plot(xvals, m * xvals + b)
axes[i].plot(xvals, post_mean["beta"] * xvals + post_mean["alpha"], "r--")
axes[i].set_xticks([0, 1])
axes[i].set_xticklabels(["basement", "floor"])
axes[i].set_ylim(-1, 3)
axes[i].set_title(c)
if not i % 2:
axes[i].set_ylabel("log radon level")
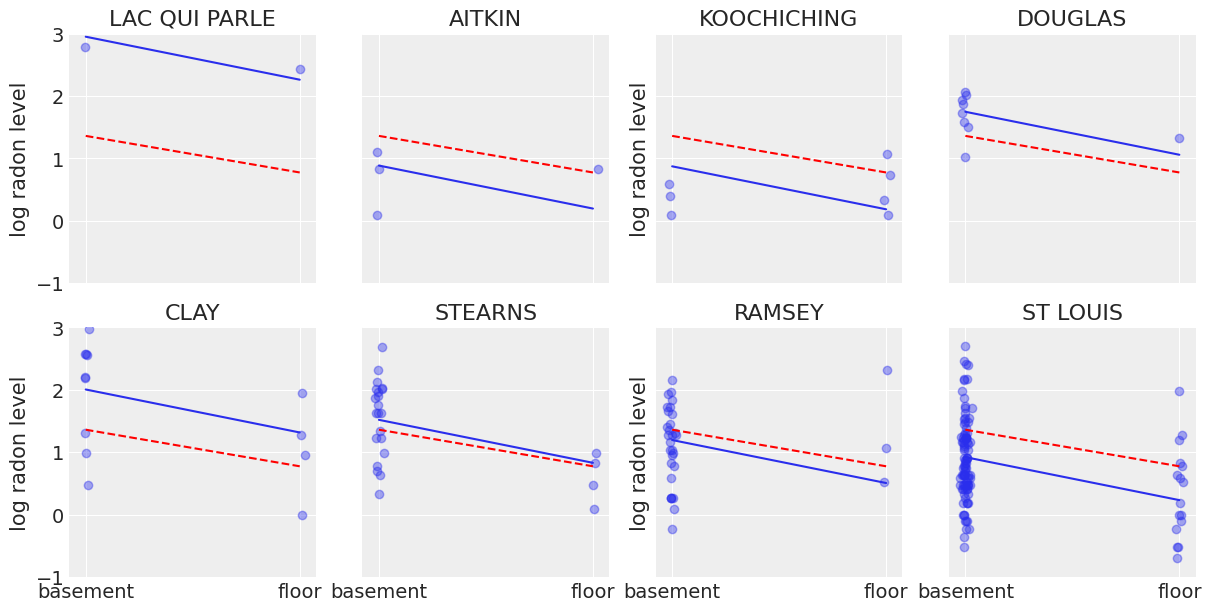
这两个模型都不令人满意:
如果我们试图识别高氡含量的县,合并数据是无用的——因为根据定义,合并模型估计的是州一级的氡含量。换句话说,合并数据导致了最大程度的欠拟合:没有考虑到各县之间的差异,只估计了总体人口。
我们不信任由使用少量观测值的模型产生的极端非汇集估计。这导致了最大程度的过拟合:只考虑了县内的变化,而总体人口(即州级水平,它告诉我们各县之间的相似性)未被估计。
这个问题在小样本量的情况下尤为突出,如上所述:在我们拥有较少地板测量数据的县中,如果这些数据点的氡水平高于地下室的数据点(Aitkin、Koochiching、Ramsey),模型将估计这些县的地板氡水平高于地下室。但我们不应相信这一结论,因为科学知识和其他县的实际情况告诉我们,通常情况是相反的(地下室氡 > 地板氡)。因此,除非我们有大量的观测数据表明某个县的氡水平相反,否则我们应该持怀疑态度,并将我们的县估计值缩小到州估计值——换句话说,我们应该在集群级别和总体级别信息之间取得平衡,而收缩的程度将取决于每个集群中数据的极端程度和数量。
这里是分层模型发挥作用的地方。
多层次和层次模型#
当我们合并数据时,我们暗示它们是从相同的模型中采样的。这忽略了采样单位之间的任何变化(除了采样方差)——我们假设各县都是相同的:

当我们进行非合并数据分析时,意味着这些数据是从独立的单独模型中采样的。与合并情况相反,这种方法声称采样单位之间的差异太大,以至于无法将它们合并——我们假设各县之间没有任何相似之处:

在层次模型中,参数被视为来自参数总体分布的样本。因此,我们将它们视为既不完全不同,也不完全相同。这就是部分池化:

我们可以使用 PyMC 轻松指定多层模型,并使用马尔可夫链蒙特卡罗方法拟合它们。
部分池化模型#
家庭氡数据集的最简单部分池化模型是仅估计氡水平,而不在任何级别使用任何预测变量的模型。部分池化模型代表了池化与非池化极端之间的折衷,大约是基于样本大小的非池化县估计值和池化估计值的加权平均值。
对于样本量较小的县,估计值会向全州平均值收缩,而对于样本量较大的县,估计值会更接近未合并的县估计值。
让我们从最简单的模型开始,该模型忽略了楼层与地下室测量的影响。
with pm.Model(coords=coords) as partial_pooling:
county_idx = pm.MutableData("county_idx", county, dims="obs_id")
# Priors
mu_a = pm.Normal("mu_a", mu=0.0, sigma=10)
sigma_a = pm.Exponential("sigma_a", 1)
# Random intercepts
alpha = pm.Normal("alpha", mu=mu_a, sigma=sigma_a, dims="county")
# Model error
sigma_y = pm.Exponential("sigma_y", 1)
# Expected value
y_hat = alpha[county_idx]
# Data likelihood
y_like = pm.Normal("y_like", mu=y_hat, sigma=sigma_y, observed=log_radon, dims="obs_id")
pm.model_to_graphviz(partial_pooling)
with partial_pooling:
partial_pooling_trace = pm.sample(tune=2000, random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu_a, sigma_a, alpha, sigma_y]
Sampling 4 chains for 2_000 tune and 1_000 draw iterations (8_000 + 4_000 draws total) took 10 seconds.
N_county = srrs_mn.groupby("county")["idnum"].count().values
fig, axes = plt.subplots(1, 2, figsize=(10, 4), sharex=True, sharey=True)
for ax, trace, level in zip(
axes,
(unpooled_trace, partial_pooling_trace),
("no pooling", "partial pooling"),
):
# add variable with x values to xarray dataset
trace.posterior = trace.posterior.assign_coords({"N_county": ("county", N_county)})
# plot means
trace.posterior.mean(dim=("chain", "draw")).plot.scatter(
x="N_county", y="alpha", ax=ax, alpha=0.9
)
ax.hlines(
partial_pooling_trace.posterior.alpha.mean(),
0.9,
max(N_county) + 1,
alpha=0.4,
ls="--",
label="Est. population mean",
)
# plot hdi
hdi = az.hdi(trace).alpha
ax.vlines(N_county, hdi.sel(hdi="lower"), hdi.sel(hdi="higher"), color="orange", alpha=0.5)
ax.set(
title=f"{level.title()} Estimates",
xlabel="Nbr obs in county (log scale)",
xscale="log",
ylabel="Log radon",
)
ax.legend(fontsize=10)
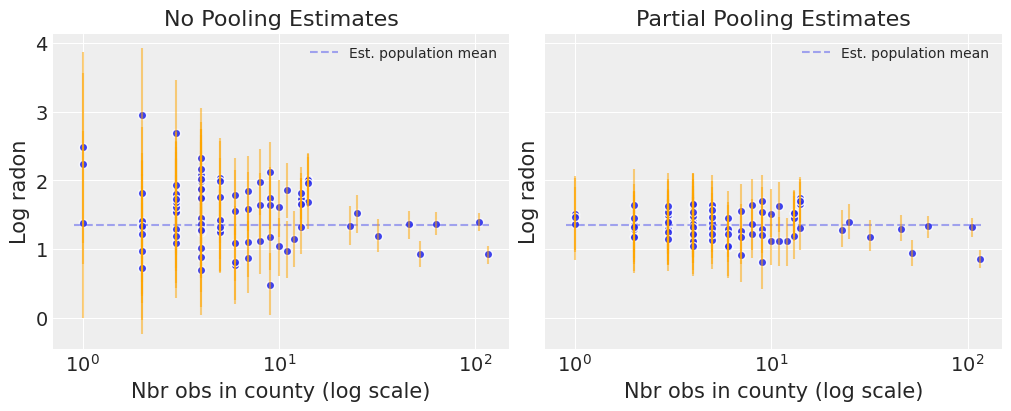
注意未合并和部分合并估计之间的差异,特别是在较小的样本量下:正如预期的那样,前者更为极端且更不精确。实际上,在部分合并模型中,小样本量县的估计值受到总体参数的影响——因此估计更为精确。此外,样本量越小,回归到总体均值(虚线灰色线)的程度越大——因此估计值越不极端。换句话说,模型对数据稀疏的县中与总体均值的极端偏差持怀疑态度。这就是所谓的收缩。
现在让我们回到前面,将floor预测器整合进来,但允许截距因县而异。
变化截距模型#
该模型允许截距在各县之间根据随机效应而变化。
哪里
和截距随机效应:
与“无池化”模型一样,我们为每个县设置了一个单独的截距,但与为每个县拟合单独的最小二乘回归模型不同,多层次建模在各县之间共享力量,使得在数据较少的县中也能进行更合理的推断。
with pm.Model(coords=coords) as varying_intercept:
floor_idx = pm.MutableData("floor_idx", floor_measure, dims="obs_id")
county_idx = pm.MutableData("county_idx", county, dims="obs_id")
# Priors
mu_a = pm.Normal("mu_a", mu=0.0, sigma=10.0)
sigma_a = pm.Exponential("sigma_a", 1)
# Random intercepts
alpha = pm.Normal("alpha", mu=mu_a, sigma=sigma_a, dims="county")
# Common slope
beta = pm.Normal("beta", mu=0.0, sigma=10.0)
# Model error
sd_y = pm.Exponential("sd_y", 1)
# Expected value
y_hat = alpha[county_idx] + beta * floor_idx
# Data likelihood
y_like = pm.Normal("y_like", mu=y_hat, sigma=sd_y, observed=log_radon, dims="obs_id")
pm.model_to_graphviz(varying_intercept)
with varying_intercept:
varying_intercept_trace = pm.sample(tune=2000, random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu_a, sigma_a, alpha, beta, sd_y]
Sampling 4 chains for 2_000 tune and 1_000 draw iterations (8_000 + 4_000 draws total) took 10 seconds.
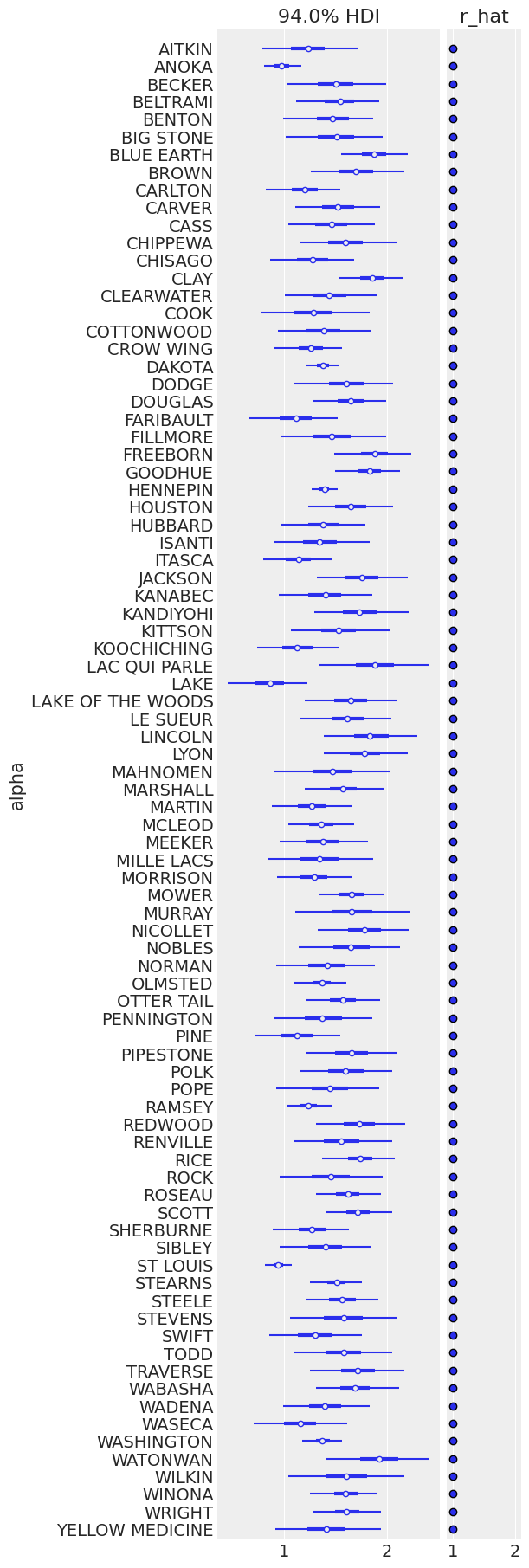
ax = pm.plot_forest(
varying_intercept_trace,
var_names=["alpha"],
figsize=(6, 18),
combined=True,
r_hat=True,
labeller=az.labels.NoVarLabeller(),
)
ax[0].set_ylabel("alpha")
Text(0, 0.5, 'alpha')
pm.plot_posterior(varying_intercept_trace, var_names=["sigma_a", "beta"]);
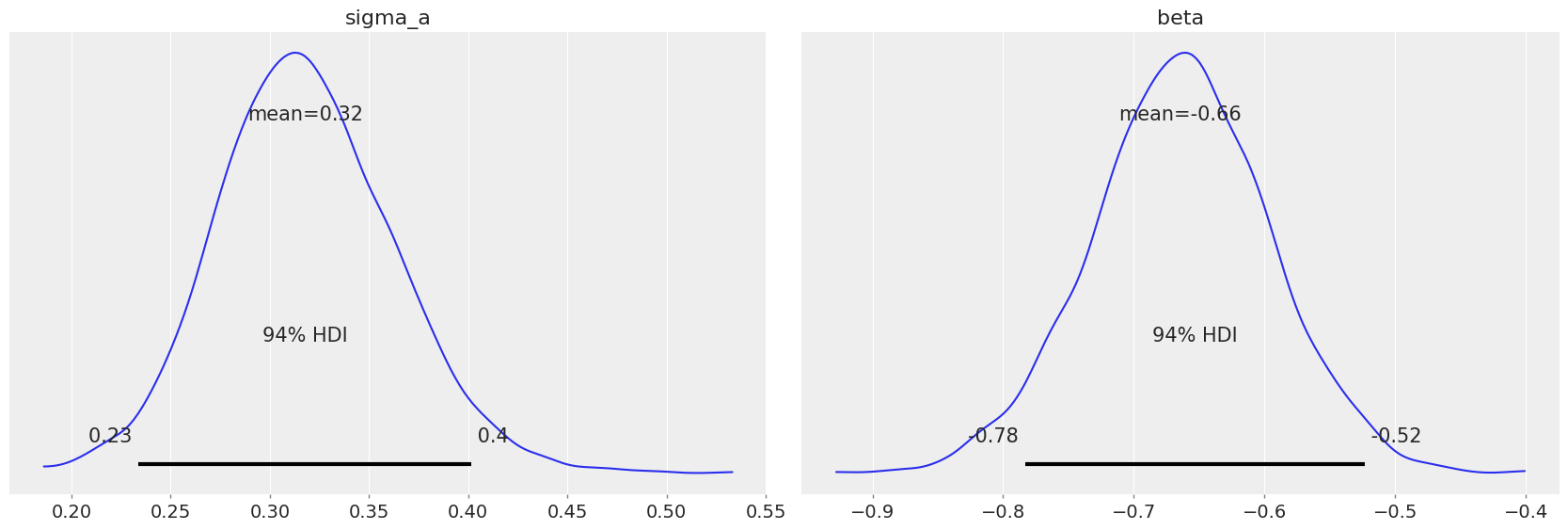
估计的floor系数约为-0.66,可以解释为没有地下室的房屋的氡水平约为有地下室的房屋的一半(\(\exp(-0.66) = 0.52\)),在考虑县的情况下。
az.summary(varying_intercept_trace, var_names=["beta"])
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| beta | -0.664 | 0.068 | -0.783 | -0.523 | 0.001 | 0.001 | 2624.0 | 2422.0 | 1.0 |
xvals = xr.DataArray([0, 1], dims="Level", coords={"Level": ["Basement", "Floor"]})
post = varying_intercept_trace.posterior # alias for readability
theta = (
(post.alpha + post.beta * xvals).mean(dim=("chain", "draw")).to_dataset(name="Mean log radon")
)
_, ax = plt.subplots()
theta.plot.scatter(x="Level", y="Mean log radon", alpha=0.2, color="k", ax=ax) # scatter
ax.plot(xvals, theta["Mean log radon"].T, "k-", alpha=0.2)
# add lines too
ax.set_title("MEAN LOG RADON BY COUNTY");
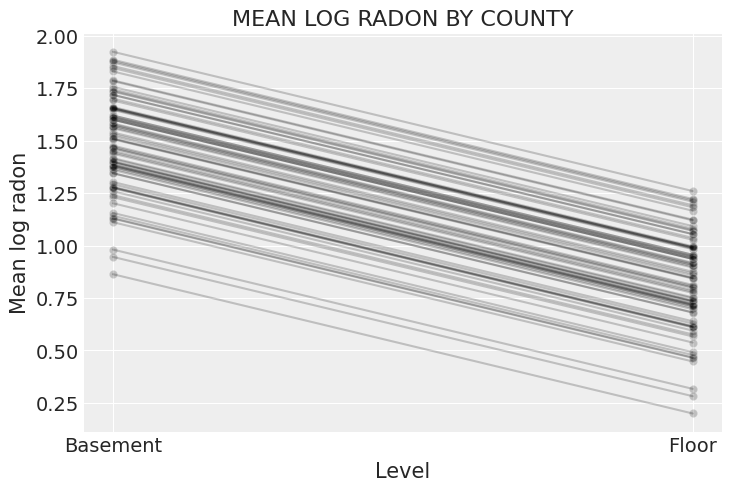
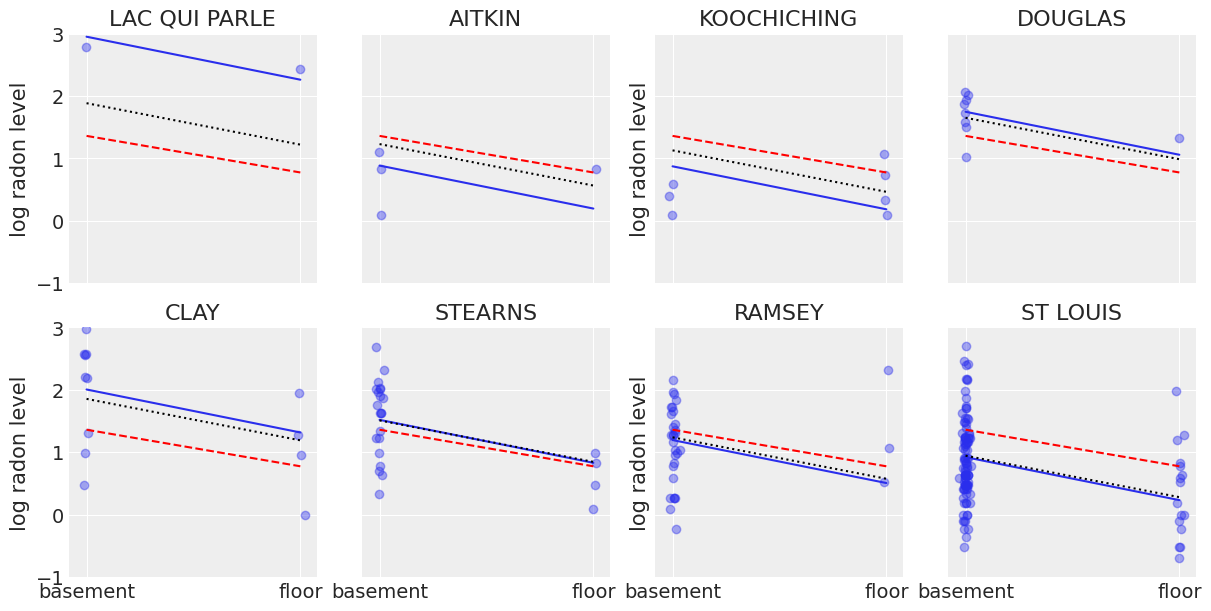
很容易证明,部分池化模型比池化或非池化模型提供了更客观合理的估计,至少对于样本量较小的县来说是如此。
sample_counties = (
"LAC QUI PARLE",
"AITKIN",
"KOOCHICHING",
"DOUGLAS",
"CLAY",
"STEARNS",
"RAMSEY",
"ST LOUIS",
)
fig, axes = plt.subplots(2, 4, figsize=(12, 6), sharey=True, sharex=True)
axes = axes.ravel()
m = unpooled_means["beta"]
for i, c in enumerate(sample_counties):
y = srrs_mn.log_radon[srrs_mn.county == c]
x = srrs_mn.floor[srrs_mn.county == c]
axes[i].scatter(x + np.random.randn(len(x)) * 0.01, y, alpha=0.4)
# No pooling model
b = unpooled_means["alpha"].sel(county=c)
# Plot both models and data
xvals = xr.DataArray(np.linspace(0, 1))
axes[i].plot(xvals, m.values * xvals + b.values)
axes[i].plot(xvals, post_mean["beta"] * xvals + post_mean["alpha"], "r--")
varying_intercept_trace.posterior.sel(county=c).beta
post = varying_intercept_trace.posterior.sel(county=c).mean(dim=("chain", "draw"))
theta = post.alpha.values + post.beta.values * xvals
axes[i].plot(xvals, theta, "k:")
axes[i].set_xticks([0, 1])
axes[i].set_xticklabels(["basement", "floor"])
axes[i].set_ylim(-1, 3)
axes[i].set_title(c)
if not i % 2:
axes[i].set_ylabel("log radon level")
变化截距和斜率模型#
最一般的模型允许截距和斜率都按县变化:
with pm.Model(coords=coords) as varying_intercept_slope:
floor_idx = pm.MutableData("floor_idx", floor_measure, dims="obs_id")
county_idx = pm.MutableData("county_idx", county, dims="obs_id")
# Priors
mu_a = pm.Normal("mu_a", mu=0.0, sigma=10.0)
sigma_a = pm.Exponential("sigma_a", 1)
mu_b = pm.Normal("mu_b", mu=0.0, sigma=10.0)
sigma_b = pm.Exponential("sigma_b", 1)
# Random intercepts
alpha = pm.Normal("alpha", mu=mu_a, sigma=sigma_a, dims="county")
# Random slopes
beta = pm.Normal("beta", mu=mu_b, sigma=sigma_b, dims="county")
# Model error
sigma_y = pm.Exponential("sigma_y", 1)
# Expected value
y_hat = alpha[county_idx] + beta[county_idx] * floor_idx
# Data likelihood
y_like = pm.Normal("y_like", mu=y_hat, sigma=sigma_y, observed=log_radon, dims="obs_id")
pm.model_to_graphviz(varying_intercept_slope)
with varying_intercept_slope:
varying_intercept_slope_trace = pm.sample(tune=2000, random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu_a, sigma_a, mu_b, sigma_b, alpha, beta, sigma_y]
Sampling 4 chains for 2_000 tune and 1_000 draw iterations (8_000 + 4_000 draws total) took 38 seconds.
请注意,此模型的跟踪中包含分歧,这可能会根据其发生的位置和频率而产生问题。这些分歧可能出现在某些层次模型中,并且可以通过使用非中心参数化来避免。
非中心化参数化#
上述部分池化模型使用了斜率随机效应的中心化参数化。也就是说,各个县的效应围绕县均值分布,其扩散程度由层次标准差参数控制。正如前面的图表所示,这一约束使得县的估计值向总体均值收缩,收缩程度与县的样本量成比例。这正是我们想要的,模型似乎拟合得很好——Gelman-Rubin统计量正好为1。
但是,仔细检查后,会发现一些问题的迹象。具体来说,让我们看看随机效应的轨迹,以及它们对应的标准差:
fig, axs = plt.subplots(nrows=2)
axs[0].plot(varying_intercept_slope_trace.posterior.sel(chain=0)["sigma_b"], alpha=0.5)
axs[0].set(ylabel="sigma_b")
axs[1].plot(varying_intercept_slope_trace.posterior.sel(chain=0)["beta"], alpha=0.05)
axs[1].set(ylabel="beta");
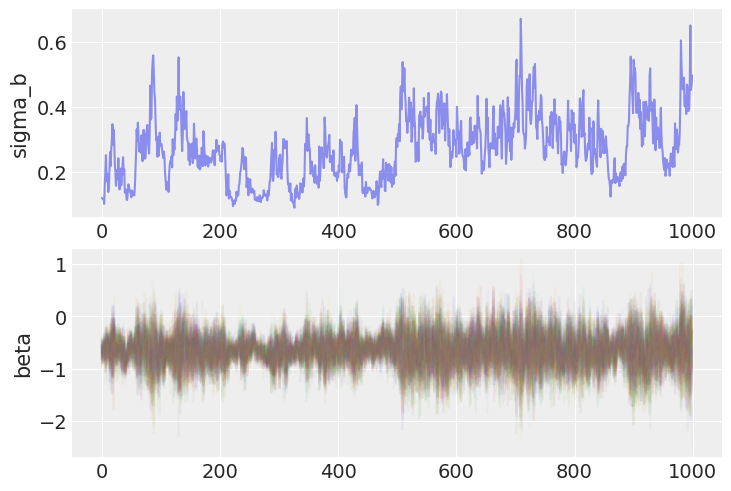
请注意,当链到达参数空间中\(\sigma_b\)的较低端时,它似乎“卡住”了,整个采样器,包括随机斜率b,混合效果不佳。
联合绘制随机效应方差和其中一个个体随机斜率展示了正在发生的情况。
ax = az.plot_pair(
varying_intercept_slope_trace,
var_names=["beta", "sigma_b"],
coords=dict(county="AITKIN"),
marginals=True,
# marginal_kwargs={"kind": "hist"},
)
ax[1, 0].set_ylim(0, 0.7);
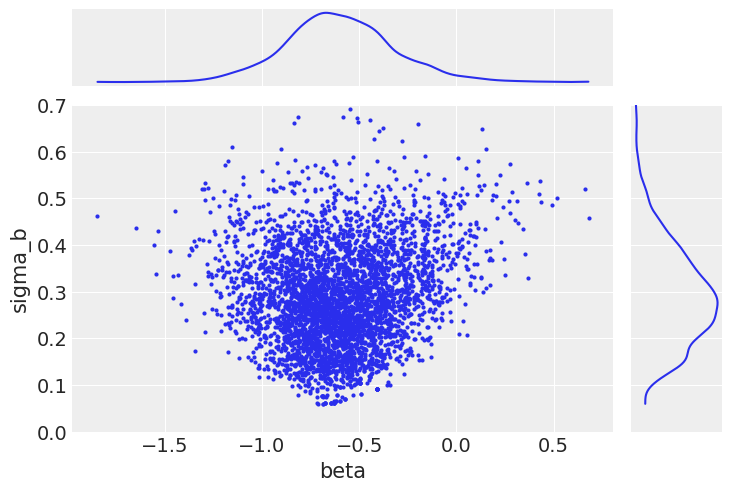
当组内方差较小时,这意味着个体随机斜率本身接近组均值。这导致组内方差样本与任意斜率(特别是那些样本量较小的斜率)之间呈现出漏斗形关系。
本身,这并不是一个问题,因为这是我们期望的行为。然而,如果采样器是为参数空间的较宽(无约束)部分调整的,它在高曲率区域会遇到困难。其结果是,接近\(\sigma_b\)下限的邻域采样不佳;实际上,在我们的链中,低于0.1的部分根本没有被采样。这将导致推断结果出现偏差。
既然我们已经发现了问题,我们能做些什么呢?解决这个问题的最佳方法是重新参数化我们的模型。请注意这个版本中的随机斜率:
with pm.Model(coords=coords) as varying_intercept_slope_noncentered:
floor_idx = pm.MutableData("floor_idx", floor_measure, dims="obs_id")
county_idx = pm.MutableData("county_idx", county, dims="obs_id")
# Priors
mu_a = pm.Normal("mu_a", mu=0.0, sigma=10.0)
sigma_a = pm.Exponential("sigma_a", 5)
# Non-centered random intercepts
# Centered: a = pm.Normal('a', mu_a, sigma=sigma_a, shape=counties)
z_a = pm.Normal("z_a", mu=0, sigma=1, dims="county")
alpha = pm.Deterministic("alpha", mu_a + z_a * sigma_a, dims="county")
mu_b = pm.Normal("mu_b", mu=0.0, sigma=10.0)
sigma_b = pm.Exponential("sigma_b", 5)
# Non-centered random slopes
z_b = pm.Normal("z_b", mu=0, sigma=1, dims="county")
beta = pm.Deterministic("beta", mu_b + z_b * sigma_b, dims="county")
# Model error
sigma_y = pm.Exponential("sigma_y", 5)
# Expected value
y_hat = alpha[county_idx] + beta[county_idx] * floor_idx
# Data likelihood
y_like = pm.Normal("y_like", mu=y_hat, sigma=sigma_y, observed=log_radon, dims="obs_id")
pm.model_to_graphviz(varying_intercept_slope_noncentered)
这是一个非中心化参数化。通过这种方式,我们指的是随机偏差不再被显式地建模为以\(\mu_b\)为中心。相反,它们是独立的标准正态分布\(\upsilon\),然后通过适当的\(\sigma_b\)值进行缩放,再通过均值进行位置变换。
这个模型采样效果更好。
with varying_intercept_slope_noncentered:
noncentered_trace = pm.sample(tune=3000, target_accept=0.95, random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [mu_a, sigma_a, z_a, mu_b, sigma_b, z_b, sigma_y]
Sampling 4 chains for 3_000 tune and 1_000 draw iterations (12_000 + 4_000 draws total) took 56 seconds.
请注意,跟踪中的瓶颈已经消失了。|
fig, axs = plt.subplots(nrows=2)
axs[0].plot(noncentered_trace.posterior.sel(chain=0)["sigma_b"], alpha=0.5)
axs[0].set(ylabel="sigma_b")
axs[1].plot(noncentered_trace.posterior.sel(chain=0)["beta"], alpha=0.05)
axs[1].set(ylabel="beta");
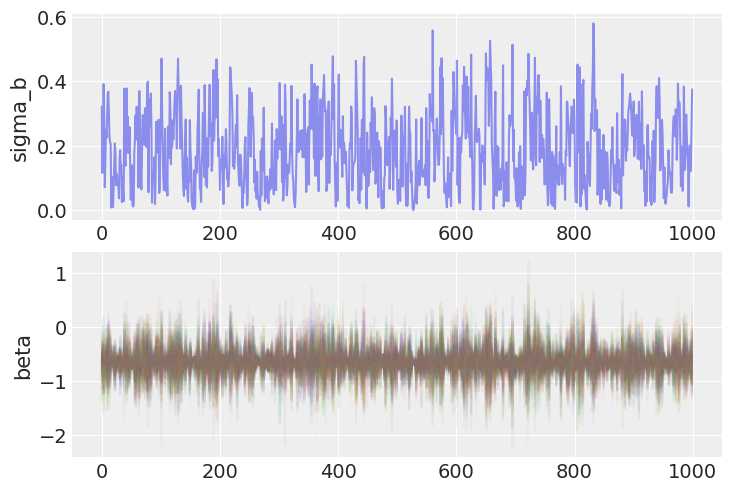
并且相应地,现在可以有效地对斜率随机效应方差的后期分布的低端进行采样。
ax = az.plot_pair(
noncentered_trace,
var_names=["beta", "sigma_b"],
coords=dict(county="AITKIN"),
marginals=True,
# marginal_kwargs={"kind": "hist"},
)
ax[1, 0].set_ylim(0, 0.7);
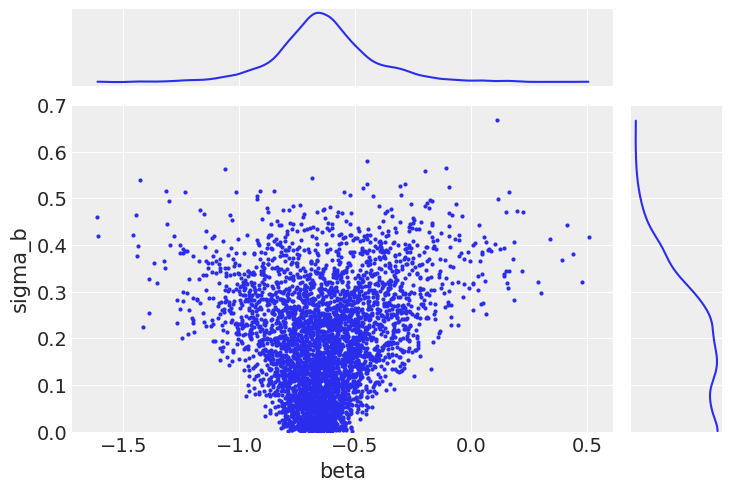
因此,我们现在正在全面探索后验支持。这导致这些参数中的偏差更小。
fig, (ax1, ax2) = plt.subplots(2, 1, sharex=True, constrained_layout=True)
az.plot_posterior(varying_intercept_slope_trace, var_names=["sigma_b"], ax=ax1)
az.plot_posterior(noncentered_trace, var_names=["sigma_b"], ax=ax2)
ax1.set_title("Centered (top) and non-centered (bottom)");
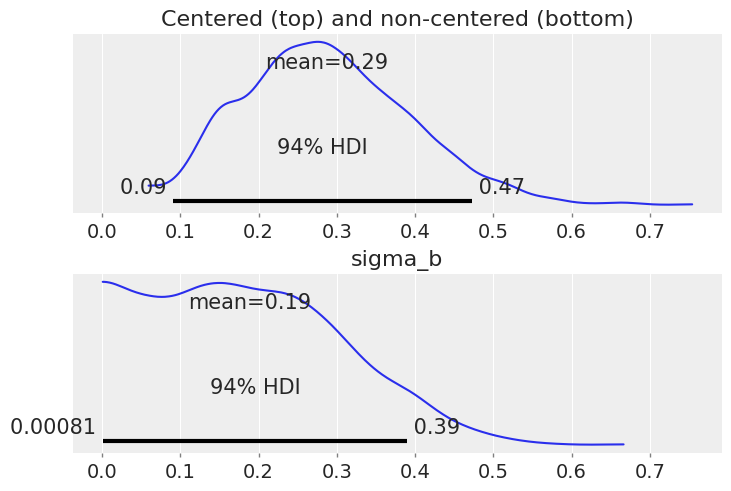
请注意,sigma_b 现在在零附近有大量的密度,这表明各县在回答 floor “处理”时并没有太大差异。
这是原始参数化的问题:当斜率随机效应的标准差非常接近零时,与它们为正值时相比,采样器在处理后验分布的几何形状时遇到了困难。然而,即使在使用非中心化模型的情况下,采样器对sigma_b也不太适应:事实上,如果你使用az.summary查看估计值,你会发现sigma_b的有效样本数量相当低。
另请注意,sigma_a 也不是那么大——即各县的基线氡水平确实有所不同,但差异不大。然而,我们从这个分布中采样并没有太大问题,因为它比 sigma_b 窄得多,并且不会危险地接近 0。
az.summary(varying_intercept_slope_trace, var_names=["sigma_a", "sigma_b"])
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| sigma_a | 0.323 | 0.045 | 0.241 | 0.408 | 0.001 | 0.001 | 1191.0 | 1807.0 | 1.01 |
| sigma_b | 0.287 | 0.106 | 0.090 | 0.473 | 0.010 | 0.007 | 105.0 | 155.0 | 1.05 |
总结这个模型,让我们绘制每个县氡气与楼层之间的关系:
xvals = xr.DataArray([0, 1], dims="Level", coords={"Level": ["Basement", "Floor"]})
post = noncentered_trace.posterior # alias for readability
theta = (
(post.alpha + post.beta * xvals).mean(dim=("chain", "draw")).to_dataset(name="Mean log radon")
)
_, ax = plt.subplots()
theta.plot.scatter(x="Level", y="Mean log radon", alpha=0.2, color="k", ax=ax) # scatter
ax.plot(xvals, theta["Mean log radon"].T, "k-", alpha=0.2)
# add lines too
ax.set_title("MEAN LOG RADON BY COUNTY");
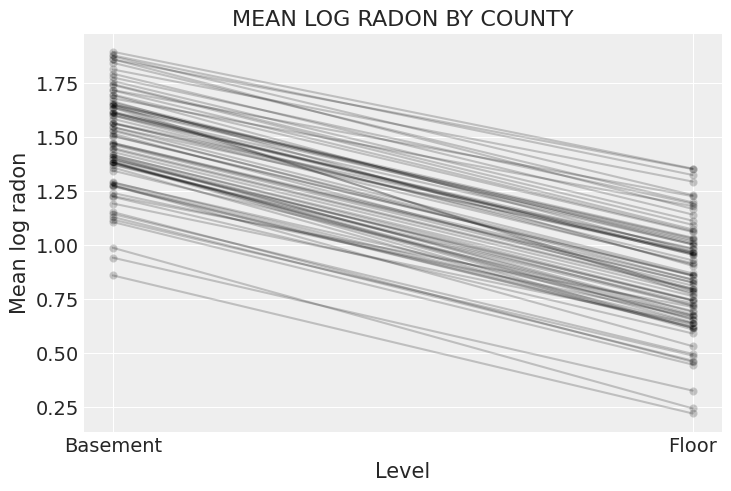
这表明,虽然截距和斜率都因县而异,但斜率的变异程度要小得多。
但等等,还有更多!我们可以(也许应该)考虑截距和斜率之间的协变:当某个县的基线氡气较低时,可能意味着楼层和地下室测量值之间的差异会减小——因为无论如何氡气都不多。这将转化为alpha和beta之间的正相关,将这一点加入我们的模型中将更有效地利用可用数据。
为了建模这种相关性,我们将使用多元正态分布,而不是为alpha和beta分别使用两个不同的正态分布。这仅仅意味着每个县的参数来自一个具有均值mu_alpha(截距)和mu_beta(斜率)的共同分布,并且斜率和截距根据协方差矩阵S共同变化。在数学形式上:
其中 \(\alpha\) 和 \(\beta\) 分别是平均截距和斜率,\(\sigma_{\alpha}\) 和 \(\sigma_{\beta}\) 分别表示截距和斜率的变化,\(P\) 是截距和斜率的协方差矩阵。在这种情况下,由于只有一个斜率,\(P\) 只包含一个相关数字:\(\alpha\) 和 \(\beta\) 之间的相关性。
这在 PyMC 中很容易实现:
coords["param"] = ["alpha", "beta"]
coords["param_bis"] = ["alpha", "beta"]
with pm.Model(coords=coords) as covariation_intercept_slope:
floor_idx = pm.MutableData("floor_idx", floor_measure, dims="obs_id")
county_idx = pm.MutableData("county_idx", county, dims="obs_id")
# prior stddev in intercepts & slopes (variation across counties):
sd_dist = pm.Exponential.dist(0.5, shape=(2,))
# get back standard deviations and rho:
chol, corr, stds = pm.LKJCholeskyCov("chol", n=2, eta=2.0, sd_dist=sd_dist)
# prior for average intercept:
mu_alpha_beta = pm.Normal("mu_alpha", mu=0.0, sigma=5.0, shape=2)
# prior for average slope:
mu_beta = pm.Normal("mu_beta", mu=0.0, sigma=1.0)
# population of varying effects:
alpha_beta_county = pm.MvNormal(
"alpha_beta_county", mu=mu_alpha_beta, chol=chol, dims=("county", "param")
)
# Expected value per county:
theta = alpha_beta_county[county_idx, 0] + alpha_beta_county[county_idx, 1] * floor_idx
# Model error:
sigma = pm.Exponential("sigma", 1.0)
y = pm.Normal("y", theta, sigma=sigma, observed=log_radon, dims="obs_id")
这是迄今为止我们做过的最复杂的模型,因此模型代码也相应地复杂。主要复杂之处在于使用了LKJCholeskyCov分布来表示协方差矩阵。这是一种协方差矩阵的Cholesky分解,使得采样更加容易。
正如你可能预料的那样,我们也希望在这里去中心化随机效应。这再次导致了一个确定性操作,这里将协方差与独立的标准正态分布相乘。
coords["param"] = ["alpha", "beta"]
coords["param_bis"] = ["alpha", "beta"]
with pm.Model(coords=coords) as covariation_intercept_slope:
floor_idx = pm.MutableData("floor_idx", floor_measure, dims="obs_id")
county_idx = pm.MutableData("county_idx", county, dims="obs_id")
# prior stddev in intercepts & slopes (variation across counties):
sd_dist = pm.Exponential.dist(0.5, shape=(2,))
# get back standard deviations and rho:
chol, corr, stds = pm.LKJCholeskyCov("chol", n=2, eta=2.0, sd_dist=sd_dist)
# priors for average intercept and slope:
mu_alpha_beta = pm.Normal("mu_alpha_beta", mu=0.0, sigma=5.0, shape=2)
# population of varying effects:
z = pm.Normal("z", 0.0, 1.0, dims=("param", "county"))
alpha_beta_county = pm.Deterministic(
"alpha_beta_county", pt.dot(chol, z).T, dims=("county", "param")
)
# Expected value per county:
theta = (
mu_alpha_beta[0]
+ alpha_beta_county[county_idx, 0]
+ (mu_alpha_beta[1] + alpha_beta_county[county_idx, 1]) * floor_idx
)
# Model error:
sigma = pm.Exponential("sigma", 1.0)
y = pm.Normal("y", theta, sigma=sigma, observed=log_radon, dims="obs_id")
covariation_intercept_slope_trace = pm.sample(
1000,
tune=3000,
target_accept=0.95,
idata_kwargs={"dims": {"chol_stds": ["param"], "chol_corr": ["param", "param_bis"]}},
)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [chol, mu_alpha_beta, z, sigma]
Sampling 4 chains for 3_000 tune and 1_000 draw iterations (12_000 + 4_000 draws total) took 111 seconds.
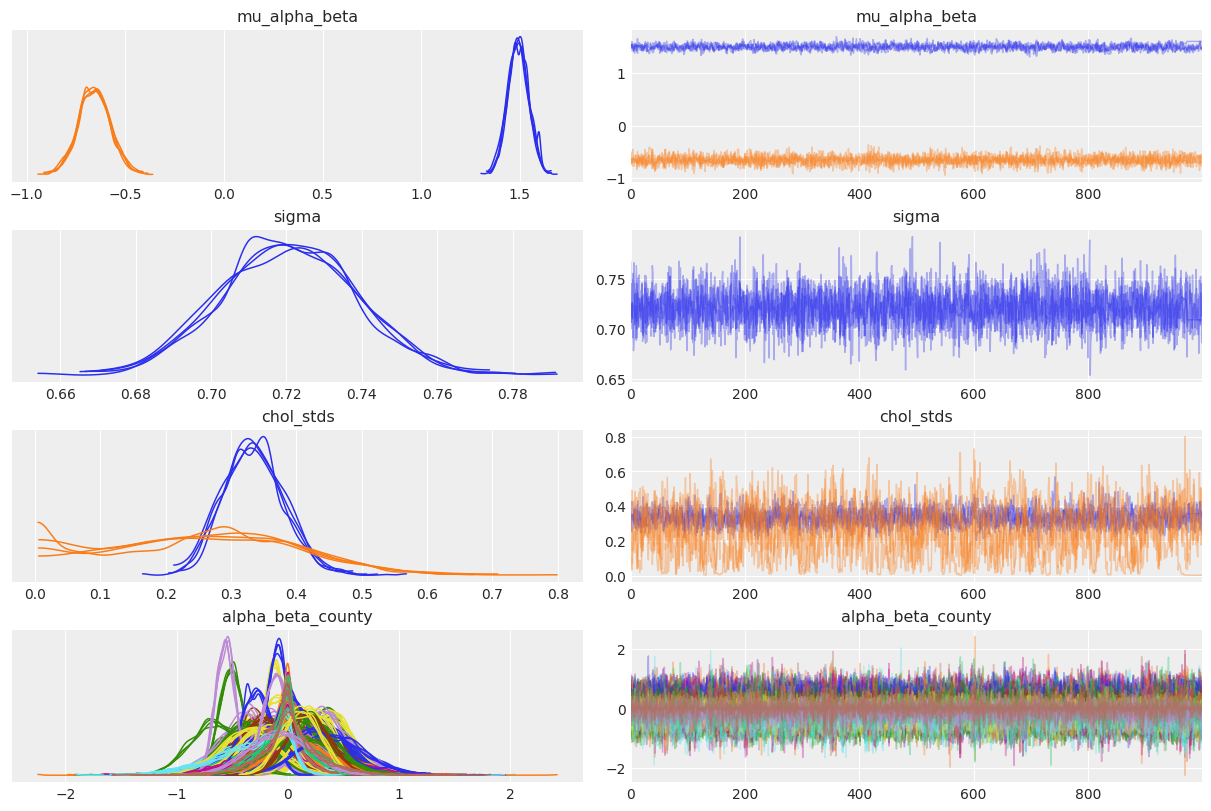
az.plot_trace(
covariation_intercept_slope_trace,
var_names=["~z", "~chol", "~chol_corr"],
compact=True,
chain_prop={"ls": "-"},
);
az.plot_trace(
covariation_intercept_slope_trace,
var_names="chol_corr",
lines=[("chol_corr", {}, 0.0)],
compact=True,
chain_prop={"ls": "-"},
coords={
"param": xr.DataArray(["alpha"], dims=["pointwise_sel"]),
"param_bis": xr.DataArray(["beta"], dims=["pointwise_sel"]),
},
);
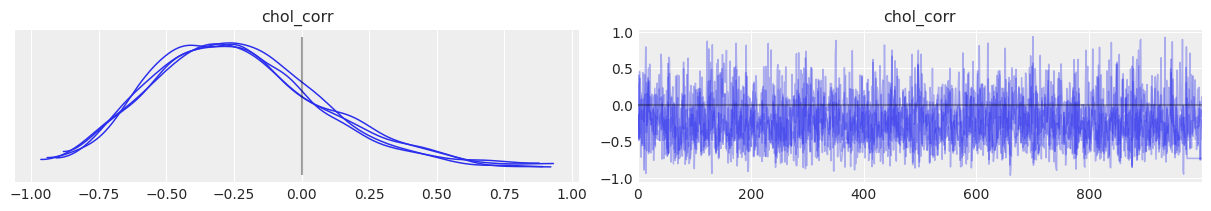
因此,斜率与截距之间的相关性似乎是负的:当县截距增加时,县斜率往往会减少。换句话说,当一个县的地下室氡气增加时,地下室氡气与地板氡气之间的差异往往会变大(因为地板读数变小,而地下室读数变大)。但同样,由于不确定性很大,相关性可能朝相反的方向发展,或者可能接近于零。
各县之间的差异有多大?直接阅读 sigma_ab 并不容易,因此让我们绘制一个森林图,并将估计值与不包括斜率和截距之间协变量的模型进行比较:
az.plot_forest(
[varying_intercept_slope_trace, covariation_intercept_slope_trace],
model_names=["No covariation", "With covariation"],
var_names=["mu_a", "mu_b", "mu_alpha_beta", "sigma_a", "sigma_b", "chol_stds", "chol_corr"],
combined=True,
figsize=(8, 6),
);
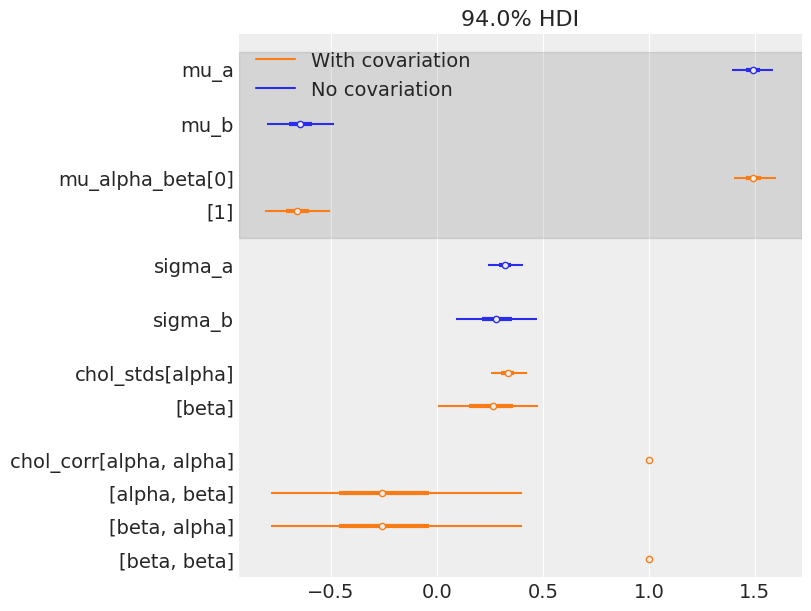
估计值非常接近,无论是均值还是标准差。但请记住,相关性提供的信息仅在县级别可见:理论上,它利用了数据中更多的信息,以获得所有县参数的更明智的信息汇总。因此,让我们在县级别上直观地比较两种模型的估计值:
# posterior means of covariation model:
a_county_cov = (
covariation_intercept_slope_trace.posterior["mu_alpha_beta"][..., 0]
+ covariation_intercept_slope_trace.posterior["alpha_beta_county"].sel(param="alpha")
).mean(dim=("chain", "draw"))
b_county_cov = (
covariation_intercept_slope_trace.posterior["mu_alpha_beta"][..., 1]
+ covariation_intercept_slope_trace.posterior["alpha_beta_county"].sel(param="beta")
).mean(dim=("chain", "draw"))
# plot both and connect with lines
avg_a_county = noncentered_trace.posterior["alpha"].mean(dim=("chain", "draw"))
avg_b_county = noncentered_trace.posterior["beta"].mean(dim=("chain", "draw"))
plt.scatter(avg_a_county, avg_b_county, label="No cov estimates", alpha=0.6)
plt.scatter(
a_county_cov,
b_county_cov,
facecolors="none",
edgecolors="k",
lw=1,
label="With cov estimates",
alpha=0.8,
)
plt.plot([avg_a_county, a_county_cov], [avg_b_county, b_county_cov], "k-", alpha=0.5)
plt.xlabel("Intercept")
plt.ylabel("Slope")
plt.legend();
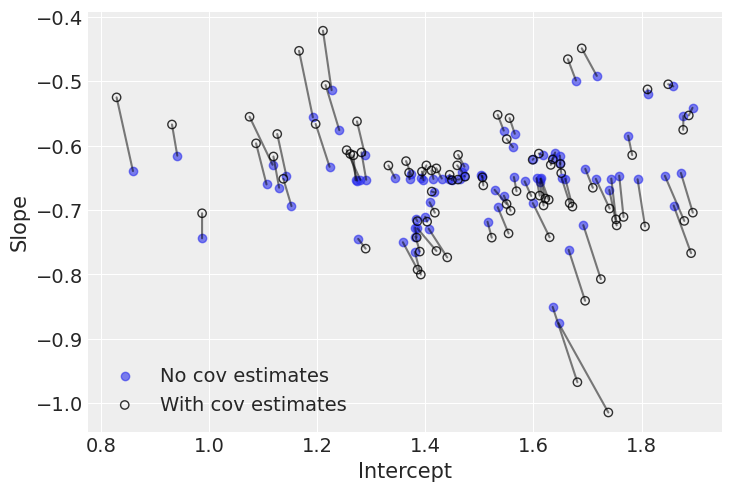
这里的负相关关系有些明显:当截距增加时,斜率减小。因此,我们理解了为什么模型将大部分后验权重放在了负相关区域。尽管如此,模型还是给出了一个非零的后验概率,认为相关性实际上可能为零或为正。
有趣的是,两个模型之间的差异出现在极端的斜率和截距值上。这是因为第二个模型利用了截距和斜率之间的轻微负相关来调整它们的估计:当截距大于(小于)平均值时,模型会降低(提高)相关斜率的值。
在全球范围内,这里有很多共识:建模相关性并没有对推断产生太大影响。我们已经看到,氡水平往往在楼层比地下室低,当我们检查平均效应(alpha 和 beta)和标准差的后验分布时,我们注意到它们几乎相同。但平均而言,具有协变量的模型会更准确——因为它从数据中挤出了额外的信息,以在两个维度上缩小估计值。
添加组级别的预测因子#
多层次模型的主要优势之一是能够同时处理多个层次的预测变量。如果我们考虑上述的变截距模型:
我们可能不使用简单的随机效应来描述预期氡值的变化,而是指定另一个带有县级别协变量的回归模型。这里,我们使用县铀读数 \(u_j\),据认为这与氡水平有关:
因此,我们现在加入了房屋级别的预测因子(楼层或地下室)以及县级别的预测因子(铀)。
请注意,该模型为每个县都设置了指示变量,并包含一个县级协变量。在经典回归中,这会导致共线性问题。在多层次模型中,通过将截距部分池化至组级线性模型的期望值,可以避免这一问题。
组级预测变量也有助于减少组级变异,\(\sigma_{\alpha}\)(这里指的是各县之间的变异,sigma_a)。这一重要含义是,组级估计会引发更强的池化效应——根据定义,较小的\(\sigma_{\alpha}\)意味着各县参数向总体州平均值的收缩更强。
这在 PyMC 中实现起来相当简单——我们只需再添加一个层次:
with pm.Model(coords=coords) as hierarchical_intercept:
# Priors
sigma_a = pm.HalfCauchy("sigma_a", 5)
# County uranium model
gamma_0 = pm.Normal("gamma_0", mu=0.0, sigma=10.0)
gamma_1 = pm.Normal("gamma_1", mu=0.0, sigma=10.0)
# Uranium model for intercept
mu_a = pm.Deterministic("mu_a", gamma_0 + gamma_1 * u)
# County variation not explained by uranium
epsilon_a = pm.Normal("epsilon_a", mu=0, sigma=1, dims="county")
alpha = pm.Deterministic("alpha", mu_a + sigma_a * epsilon_a, dims="county")
# Common slope
beta = pm.Normal("beta", mu=0.0, sigma=10.0)
# Model error
sigma_y = pm.Uniform("sigma_y", lower=0, upper=100)
# Expected value
y_hat = alpha[county] + beta * floor_measure
# Data likelihood
y_like = pm.Normal("y_like", mu=y_hat, sigma=sigma_y, observed=log_radon)
pm.model_to_graphviz(hierarchical_intercept)
你看到新的层次了吗,包括sigma_a和gamma,这是一个二维的层次,因为它包含了a_county的线性模型?
with hierarchical_intercept:
hierarchical_intercept_trace = pm.sample(tune=2000, random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [sigma_a, gamma_0, gamma_1, epsilon_a, beta, sigma_y]
Sampling 4 chains for 2_000 tune and 1_000 draw iterations (8_000 + 4_000 draws total) took 22 seconds.
uranium = u
post = hierarchical_intercept_trace.posterior.assign_coords(uranium=uranium)
avg_a = post["mu_a"].mean(dim=("chain", "draw")).values[np.argsort(uranium)]
avg_a_county = post["alpha"].mean(dim=("chain", "draw"))
avg_a_county_hdi = az.hdi(post, var_names="alpha")["alpha"]
_, ax = plt.subplots()
ax.plot(uranium[np.argsort(uranium)], avg_a, "k--", alpha=0.6, label="Mean intercept")
az.plot_hdi(
uranium,
post["alpha"],
fill_kwargs={"alpha": 0.1, "color": "k", "label": "Mean intercept HPD"},
ax=ax,
)
ax.scatter(uranium, avg_a_county, alpha=0.8, label="Mean county-intercept")
ax.vlines(
uranium,
avg_a_county_hdi.sel(hdi="lower"),
avg_a_county_hdi.sel(hdi="higher"),
alpha=0.5,
color="orange",
)
plt.xlabel("County-level uranium")
plt.ylabel("Intercept estimate")
plt.legend(fontsize=9);
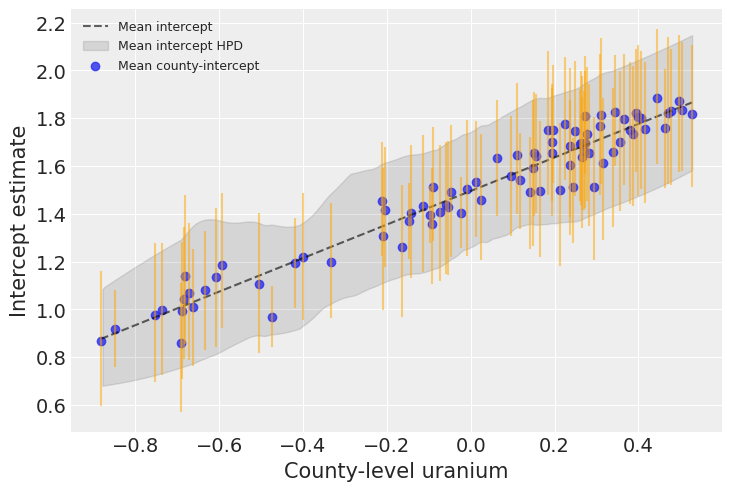
铀确实与每个县的基线氡水平密切相关。上图显示了平均关系及其不确定性:一个平均县的基线氡水平作为铀的函数,以及该氡水平的94% HPD(虚线和包络线)。蓝色点和橙色条表示基线氡与铀之间的关系,但现在针对每个县。正如你所见,现在的不确定性更大了,因为它叠加在平均不确定性之上——毕竟每个县都有其独特性。
如果我们比较这个模型的县截距与没有县级别协变量的部分池化模型的截距:截距的标准误差比没有县级别协变量的部分池化模型更窄。
labeller = az.labels.mix_labellers((az.labels.NoVarLabeller, az.labels.NoModelLabeller))
ax = az.plot_forest(
[varying_intercept_trace, hierarchical_intercept_trace],
model_names=["W/t. county pred.", "With county pred."],
var_names=["alpha"],
combined=True,
figsize=(6, 40),
textsize=9,
labeller=labeller(),
)
ax[0].set_ylabel("alpha");
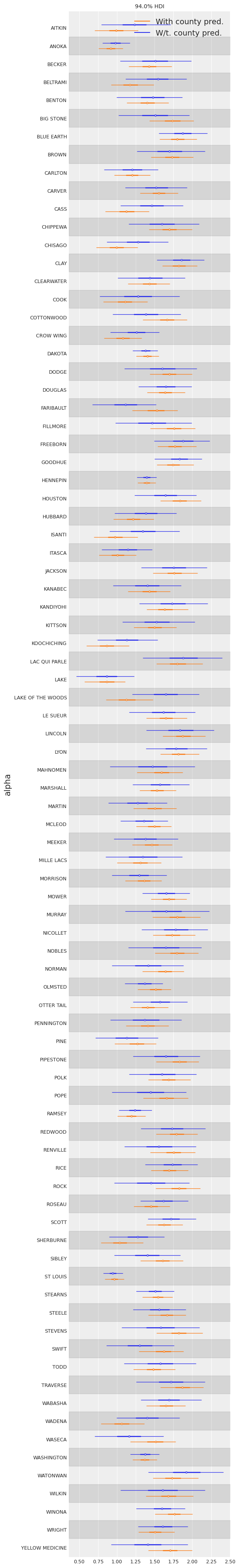
我们看到,包含县级协变量的模型的兼容区间更窄。这是预期的,因为协变量的作用是减少结果变量的变异——前提是协变量具有预测价值。更重要的是,通过这个模型,我们能够从数据中提取更多的信息。
各级别之间的相关性#
在某些情况下,拥有多层次的预测变量可以揭示个体层面变量与组残差之间的相关性。我们可以通过在组截距模型中包含个体预测变量的平均值作为协变量来解释这一点。
这些通常被称为情境效应。
要将这些效果添加到我们的模型中,让我们创建一个包含每个县floor均值的新变量,并将其添加到我们之前的模型中:
# Create new variable for mean of floor across counties
avg_floor_data = srrs_mn.groupby("county")["floor"].mean().values
with pm.Model(coords=coords) as contextual_effect:
floor_idx = pm.Data("floor_idx", floor_measure, mutable=True)
county_idx = pm.Data("county_idx", county, mutable=True)
y = pm.Data("y", log_radon, mutable=True)
# Priors
sigma_a = pm.HalfCauchy("sigma_a", 5)
# County uranium model for slope
gamma = pm.Normal("gamma", mu=0.0, sigma=10, shape=3)
# Uranium model for intercept
mu_a = pm.Deterministic("mu_a", gamma[0] + gamma[1] * u + gamma[2] * avg_floor_data)
# County variation not explained by uranium
epsilon_a = pm.Normal("epsilon_a", mu=0, sigma=1, dims="county")
alpha = pm.Deterministic("alpha", mu_a + sigma_a * epsilon_a)
# Common slope
beta = pm.Normal("beta", mu=0.0, sigma=10)
# Model error
sigma_y = pm.Uniform("sigma_y", lower=0, upper=100)
# Expected value
y_hat = alpha[county_idx] + beta * floor_idx
# Data likelihood
y_like = pm.Normal("y_like", mu=y_hat, sigma=sigma_y, observed=y)
with contextual_effect:
contextual_effect_trace = pm.sample(tune=2000, random_seed=RANDOM_SEED)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [sigma_a, gamma, epsilon_a, beta, sigma_y]
Sampling 4 chains for 2_000 tune and 1_000 draw iterations (8_000 + 4_000 draws total) took 23 seconds.
az.summary(contextual_effect_trace, var_names="gamma", round_to=2)
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| gamma[0] | 1.43 | 0.05 | 1.33 | 1.51 | 0.0 | 0.0 | 2614.08 | 2072.20 | 1.0 |
| gamma[1] | 0.70 | 0.09 | 0.53 | 0.86 | 0.0 | 0.0 | 2935.94 | 1793.38 | 1.0 |
| gamma[2] | 0.39 | 0.19 | 0.04 | 0.77 | 0.0 | 0.0 | 3077.64 | 2550.77 | 1.0 |
因此,我们可以从这一点推断出,没有地下室的房屋比例较高的县往往具有较高的氡基线水平。这似乎是新的,因为到目前为止我们看到floor与氡水平呈负相关。但请记住,这是在家庭层面:氡往往在有地下室的房屋中较高。但在县层面,似乎平均而言,县内地下室越少,氡越多。所以这并不矛盾。更重要的是,\(\gamma_2\)的估计相当不确定,并且与零重叠,因此关系可能并不那么强。最后,让我们注意到\(\gamma_2\)估计的是与铀效应不同的东西,因为这已经由\(\gamma_1\)考虑了——它回答了“一旦我们知道县内的铀水平,了解没有地下室的房屋比例是否有任何价值?”的问题。
所有这些都是为了说明我们不应该因果性地解释这一点:没有可信的机制表明地下室(或其缺失)导致氡排放。更有可能的是,我们的因果图遗漏了某些东西:一个混杂变量,它同时影响地下室建设和氡水平,正潜伏在某个角落……也许它是土壤类型,它可能影响建筑结构的类型和氡的水平?也许将这一点加入我们的模型会有助于因果推断。
预测#
Gelman [2006] 使用交叉验证测试来检查未合并、合并和部分合并模型的预测误差
交叉验证预测误差的均方根:
未合并 = 0.86
pooled = 0.84
多级 = 0.79
在多层次模型中,可以进行两种类型的预测:
现有组中的一个新个体
新群体中的新个体
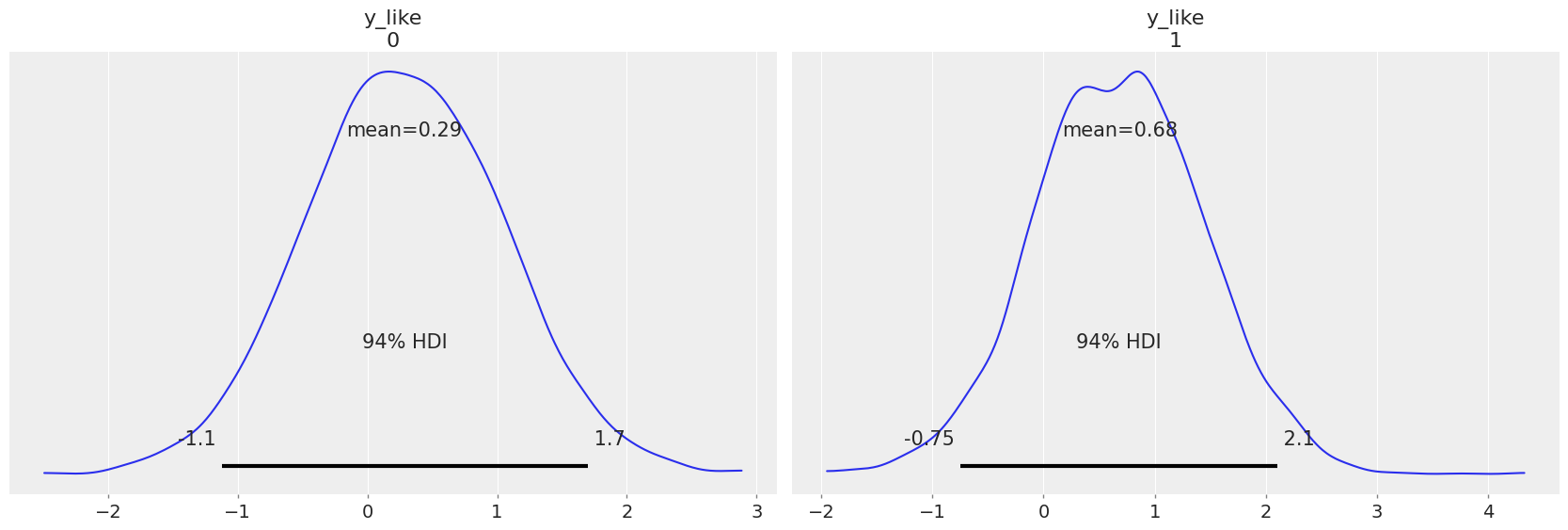
例如,如果我们想为圣路易斯和卡纳贝克县没有地下室的新房子做一个预测,我们只需要从具有适当截距的氡模型中进行采样。
也就是说,
因为我们之前明智地将县指数和楼层值设置为共享变量,所以我们可以直接将它们修改为所需的值(分别为69和1),并采样相应的后验预测,而不需要重新定义和重新编译我们的模型。使用上面刚刚提到的模型:
prediction_coords = {"obs_id": ["ST LOUIS", "KANABEC"]}
with contextual_effect:
pm.set_data({"county_idx": np.array([69, 31]), "floor_idx": np.array([1, 1]), "y": np.ones(2)})
stl_pred = pm.sample_posterior_predictive(contextual_effect_trace.posterior)
contextual_effect_trace.extend(stl_pred)
Sampling: [y_like]
az.plot_posterior(contextual_effect_trace, group="posterior_predictive");
多层次模型的优势#
考虑观测数据的自然层次结构。
(代表性不足的)群体的系数估计。
在估计组级别系数时结合个体和组级别的信息。
允许在不同组之间个体水平系数的变异。
作为解决此问题的层次建模的替代方法,请查看使用地理空间方法来建模氡水平。
参考资料#
水印#
%load_ext watermark
%watermark -n -u -v -iv -w
Last updated: Wed Dec 07 2022
Python implementation: CPython
Python version : 3.9.10
IPython version : 8.7.0
arviz : 0.14.0
xarray : 2022.12.0
pymc : 4.4.0
matplotlib: 3.6.2
pandas : 1.5.2
seaborn : 0.12.1
numpy : 1.21.6
pytensor : 2.8.7
Watermark: 2.3.0