经验近似概述#
对于大多数模型,我们使用Metropolis或NUTS等采样MCMC算法。在PyMC中,我们习惯于存储MCMC样本的轨迹,然后使用它们进行分析。PyMC中的变分推断子模块也有类似的概念:Empirical。这种近似类型为SVGD采样器存储粒子。独立SVGD粒子与MCMC样本之间没有区别。Empirical充当MCMC采样输出与apply_replacements或sample_node等全功能VI工具之间的桥梁。有关接口描述,请参见变分API快速入门。这里我们将只关注Emprical,并概述Empirical近似的特定内容。
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pymc as pm
import pytensor
import seaborn as sns
from pandas import DataFrame
print(f"Running on PyMC v{pm.__version__}")
Running on PyMC v5.0.1
%config InlineBackend.figure_format = 'retina'
az.style.use("arviz-darkgrid")
np.random.seed(42)
多模态密度#
让我们回顾一下变分API快速入门中的问题,我们在那里首次获得了NUTS轨迹
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [x]
Sampling 4 chains for 1_000 tune and 50_000 draw iterations (4_000 + 200_000 draws total) took 87 seconds.
with model:
idata = pm.to_inference_data(trace)
az.plot_trace(idata);
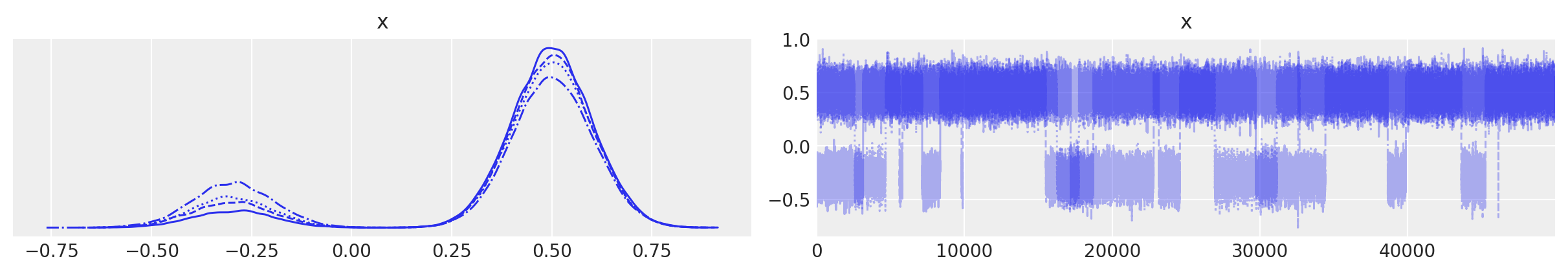
太好了。首先,有了轨迹,我们可以创建 Empirical 近似
print(pm.Empirical.__doc__)
**Single Group Full Rank Approximation**
Builds Approximation instance from a given trace,
it has the same interface as variational approximation
with model:
approx = pm.Empirical(trace)
<pymc.variational.approximations.Empirical at 0x7f64b15d15b0>
这种近似方法有其自己的底层存储用于样本,即 pytensor.shared 本身
approx.histogram
histogram
approx.histogram.get_value()[:10]
array([[-0.27366748],
[-0.32806332],
[-0.56953621],
[-0.2994719 ],
[-0.18962334],
[-0.24262214],
[-0.36759098],
[-0.23522732],
[-0.37741766],
[-0.3298074 ]])
approx.histogram.get_value().shape
(200000, 1)
它与你之前在trace中的样本数量完全相同。在我们的特定情况下,它是50k。另一个需要注意的事情是,如果你有一个包含多个链的multitrace,你将一次性存储更多的样本。我们展平所有的trace以创建Empirical。
这个直方图是关于我们如何存储样本的。结构非常简单:(n_samples, n_dim) 这些变量的顺序在类内部存储,在大多数情况下终端用户不需要使用
approx.ordering
OrderedDict([('x', ('x', slice(0, 1, None), (), dtype('float64')))])
从后验分布中进行采样是带有替换的均匀采样。调用 approx.sample(1000) 你会再次得到轨迹,但顺序是不确定的。现在无法通过 approx.sample 重建原始轨迹。
new_trace = approx.sample(50000)
采样函数编译后,采样变得非常快
az.plot_trace(new_trace);
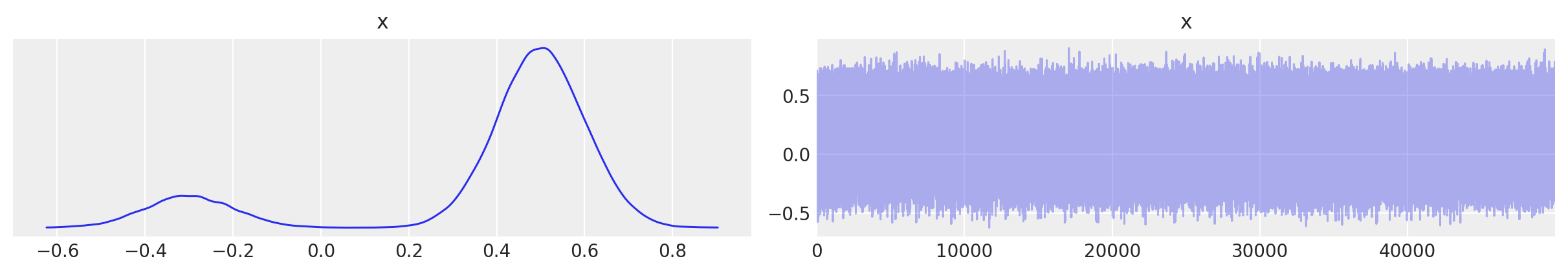
你看到这里已经没有顺序了,但重建的密度是相同的。
2D 密度#
mu = pm.floatX([0.0, 0.0])
cov = pm.floatX([[1, 0.5], [0.5, 1.0]])
with pm.Model() as model:
pm.MvNormal("x", mu=mu, cov=cov, shape=2)
trace = pm.sample(1000, return_inferencedata=False)
idata = pm.to_inference_data(trace)
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [x]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 7 seconds.
with model:
approx = pm.Empirical(trace)
az.plot_trace(approx.sample(10000));
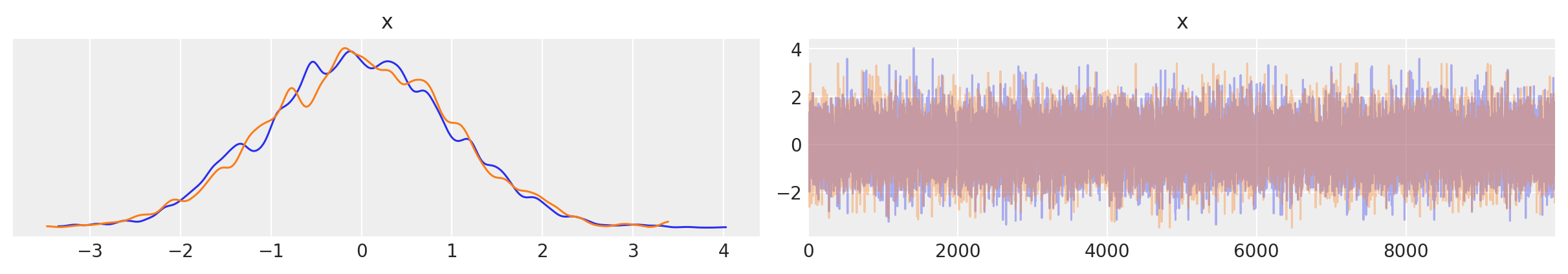
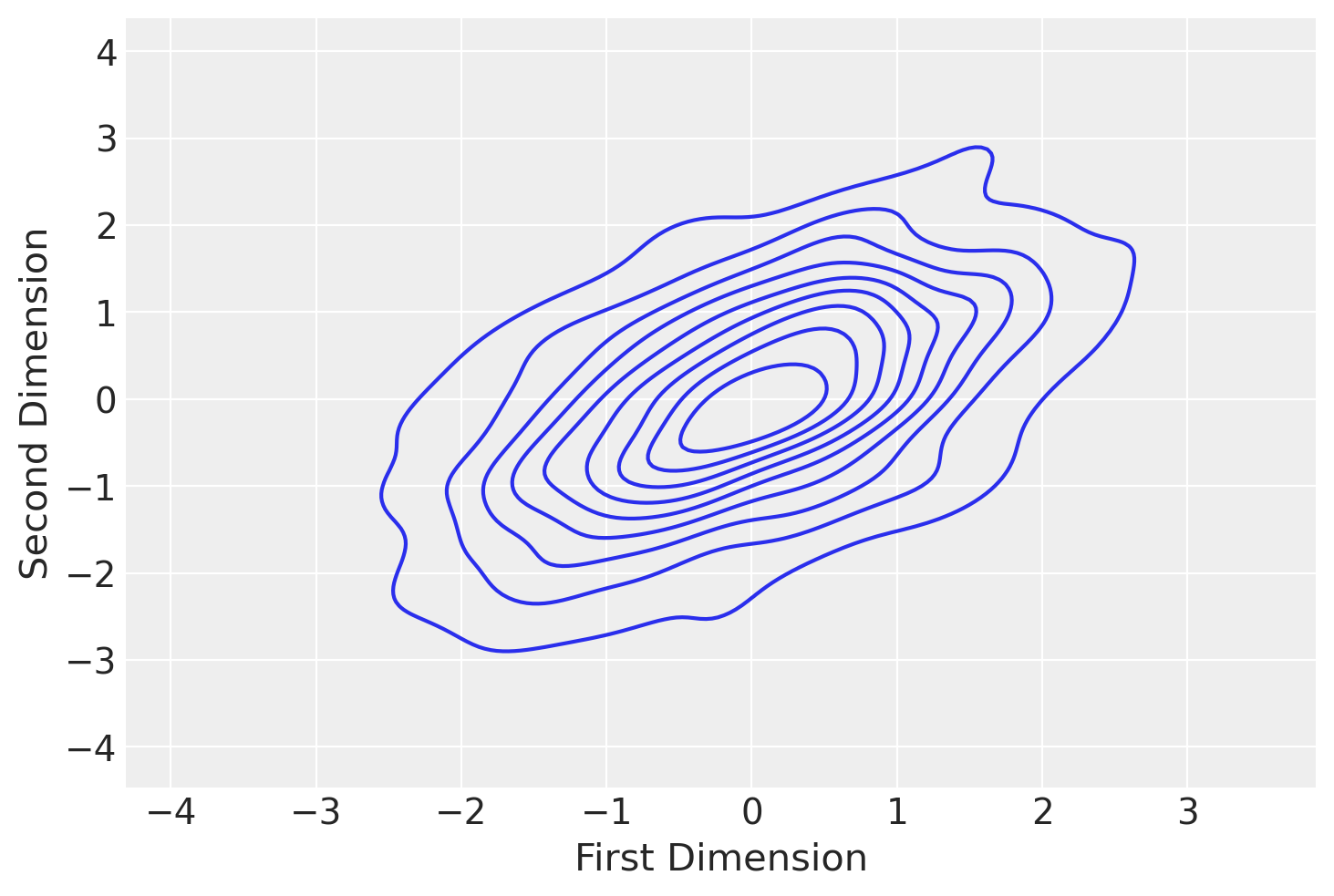
之前我们有一个 trace_cov 函数
with model:
print(pm.trace_cov(trace))
[[1.04134257 0.53158646]
[0.53158646 1.02179671]]
现在我们可以使用Empirical来估计相同的协方差
print(approx.cov)
Elemwise{true_div,no_inplace}.0
这是一个张量对象,我们需要对其进行评估。
print(approx.cov.eval())
[[1.04108223 0.53145356]
[0.53145356 1.02154126]]
估计值非常接近,差异是由于精度误差造成的。我们可以用同样的方式得到平均值
print(approx.mean.eval())
[-0.03548692 -0.03420244]
水印#
%load_ext watermark
%watermark -n -u -v -iv -w
Last updated: Fri Jan 13 2023
Python implementation: CPython
Python version : 3.9.0
IPython version : 8.8.0
pymc : 5.0.1
pytensor : 2.8.11
arviz : 0.14.0
sys : 3.9.0 | packaged by conda-forge | (default, Nov 26 2020, 07:57:39)
[GCC 9.3.0]
numpy : 1.23.5
seaborn : 0.12.2
matplotlib: 3.6.2
Watermark: 2.3.1