GLM: 负二项回归#
注意
本笔记本使用了不是 PyMC 依赖项的库,因此需要专门安装这些库才能运行此笔记本。打开下面的下拉菜单以获取更多指导。
Extra dependencies install instructions
为了在本地或binder上运行此笔记本,您不仅需要一个安装了所有可选依赖项的PyMC工作环境,还需要安装一些额外的依赖项。有关安装PyMC本身的建议,请参阅安装
您可以使用您喜欢的包管理器安装这些依赖项,我们提供了以下作为示例的pip和conda命令。
$ pip install seaborn
请注意,如果您想(或需要)从笔记本内部而不是命令行安装软件包,您可以通过运行pip命令的变体来安装软件包:
导入系统
!{sys.executable} -m pip install seaborn
您不应运行!pip install,因为它可能会在不同的环境中安装包,即使安装了也无法从Jupyter笔记本中使用。
另一个替代方案是使用conda:
$ conda install seaborn
在安装科学计算Python包时,我们推荐使用conda forge
RANDOM_SEED = 8927
rng = np.random.default_rng(RANDOM_SEED)
%config InlineBackend.figure_format = "retina"
az.style.use("arviz-darkgrid")
本笔记本紧密跟随Jonathan Sedar的GLM泊松回归示例(该示例又受到Ian Osvald的项目的启发),除了这里的数据是负二项分布而不是泊松分布。
负二项回归用于对计数数据进行建模,这些数据的方差大于均值。负二项分布可以被视为泊松分布,其速率参数是伽马分布的,因此速率参数可以调整以解释增加的方差。
生成数据#
与泊松回归示例一样,我们假设打喷嚏的发生率在某个基线水平,并且饮酒、不服用抗组胺药或两者都做会增加其频率。
泊松数据#
首先,让我们看一下泊松回归示例中的一些泊松分布数据。
# Mean Poisson values
theta_noalcohol_meds = 1 # no alcohol, took an antihist
theta_alcohol_meds = 3 # alcohol, took an antihist
theta_noalcohol_nomeds = 6 # no alcohol, no antihist
theta_alcohol_nomeds = 36 # alcohol, no antihist
# Create samples
q = 1000
df_pois = pd.DataFrame(
{
"nsneeze": np.concatenate(
(
rng.poisson(theta_noalcohol_meds, q),
rng.poisson(theta_alcohol_meds, q),
rng.poisson(theta_noalcohol_nomeds, q),
rng.poisson(theta_alcohol_nomeds, q),
)
),
"alcohol": np.concatenate(
(
np.repeat(False, q),
np.repeat(True, q),
np.repeat(False, q),
np.repeat(True, q),
)
),
"nomeds": np.concatenate(
(
np.repeat(False, q),
np.repeat(False, q),
np.repeat(True, q),
np.repeat(True, q),
)
),
}
)
df_pois.groupby(["nomeds", "alcohol"])["nsneeze"].agg(["mean", "var"])
| mean | var | ||
|---|---|---|---|
| nomeds | alcohol | ||
| False | False | 1.047 | 1.127919 |
| True | 2.986 | 2.960765 | |
| True | False | 5.981 | 6.218858 |
| True | 35.929 | 36.064023 |
由于泊松分布的随机变量的均值和方差相等,样本均值和方差非常接近。
负二项分布数据#
现在,假设数据集中的每个对象都患有流感,增加了他们打喷嚏的方差(并导致一些不幸的人每天打喷嚏超过70次)。如果打喷嚏的平均次数保持不变但方差增加,数据可能遵循负二项分布。
# Gamma shape parameter
alpha = 10
def get_nb_vals(mu, alpha, size):
"""Generate negative binomially distributed samples by
drawing a sample from a gamma distribution with mean `mu` and
shape parameter `alpha', then drawing from a Poisson
distribution whose rate parameter is given by the sampled
gamma variable.
"""
g = stats.gamma.rvs(alpha, scale=mu / alpha, size=size)
return stats.poisson.rvs(g)
# Create samples
n = 1000
df = pd.DataFrame(
{
"nsneeze": np.concatenate(
(
get_nb_vals(theta_noalcohol_meds, alpha, n),
get_nb_vals(theta_alcohol_meds, alpha, n),
get_nb_vals(theta_noalcohol_nomeds, alpha, n),
get_nb_vals(theta_alcohol_nomeds, alpha, n),
)
),
"alcohol": np.concatenate(
(
np.repeat(False, n),
np.repeat(True, n),
np.repeat(False, n),
np.repeat(True, n),
)
),
"nomeds": np.concatenate(
(
np.repeat(False, n),
np.repeat(False, n),
np.repeat(True, n),
np.repeat(True, n),
)
),
}
)
df
| nsneeze | alcohol | nomeds | |
|---|---|---|---|
| 0 | 2 | False | False |
| 1 | 1 | False | False |
| 2 | 2 | False | False |
| 3 | 2 | False | False |
| 4 | 3 | False | False |
| ... | ... | ... | ... |
| 3995 | 28 | True | True |
| 3996 | 63 | True | True |
| 3997 | 19 | True | True |
| 3998 | 34 | True | True |
| 3999 | 40 | True | True |
4000行 × 3列
df.groupby(["nomeds", "alcohol"])["nsneeze"].agg(["mean", "var"])
| mean | var | ||
|---|---|---|---|
| nomeds | alcohol | ||
| False | False | 0.990 | 1.137037 |
| True | 2.983 | 4.030742 | |
| True | False | 6.004 | 8.658643 |
| True | 35.918 | 169.018294 |
与泊松回归示例一样,我们看到饮酒和/或不服用抗组胺药会增加打喷嚏率,程度各不相同。与该示例不同的是,对于每种alcohol和nomeds的组合,nsneeze的方差都大于均值。这表明泊松分布不适合这些数据,因为泊松分布的均值和方差是相等的。
可视化数据#
g = sns.catplot(x="nsneeze", row="nomeds", col="alcohol", data=df, kind="count", aspect=1.5)
# Make x-axis ticklabels less crowded
ax = g.axes[1, 0]
labels = range(len(ax.get_xticklabels(which="both")))
ax.set_xticks(labels[::5])
ax.set_xticklabels(labels[::5]);
/opt/homebrew/Caskroom/miniconda/base/envs/pymc-dev/lib/python3.11/site-packages/seaborn/axisgrid.py:123: UserWarning: The figure layout has changed to tight
self._figure.tight_layout(*args, **kwargs)
/opt/homebrew/Caskroom/miniconda/base/envs/pymc-dev/lib/python3.11/site-packages/seaborn/axisgrid.py:123: UserWarning: The figure layout has changed to tight
self._figure.tight_layout(*args, **kwargs)
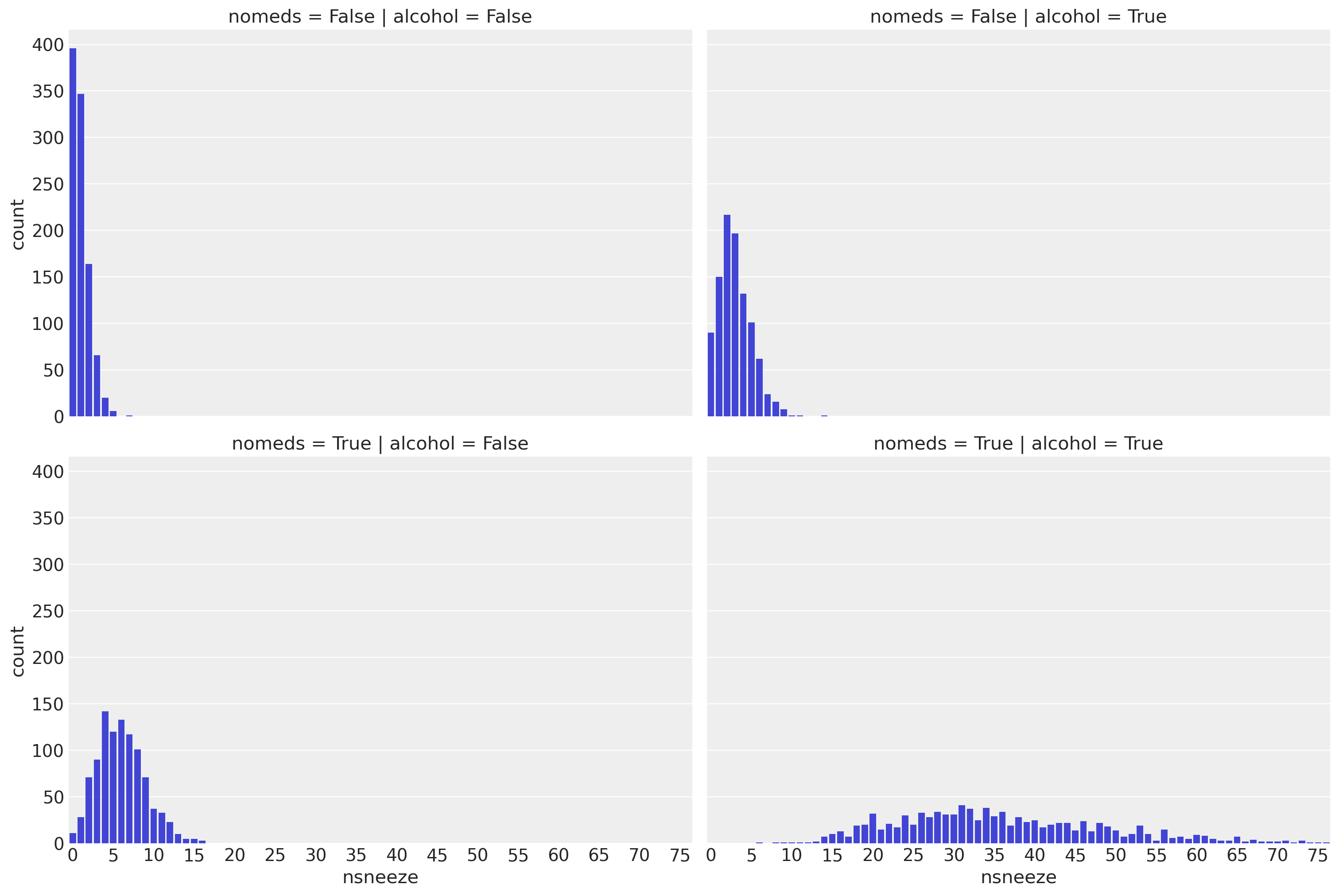
负二项回归#
创建GLM模型#
COORDS = {"regressor": ["nomeds", "alcohol", "nomeds:alcohol"], "obs_idx": df.index}
with pm.Model(coords=COORDS) as m_sneeze_inter:
a = pm.Normal("intercept", mu=0, sigma=5)
b = pm.Normal("slopes", mu=0, sigma=1, dims="regressor")
alpha = pm.Exponential("alpha", 0.5)
M = pm.ConstantData("nomeds", df.nomeds.to_numpy(), dims="obs_idx")
A = pm.ConstantData("alcohol", df.alcohol.to_numpy(), dims="obs_idx")
S = pm.ConstantData("nsneeze", df.nsneeze.to_numpy(), dims="obs_idx")
λ = pm.math.exp(a + b[0] * M + b[1] * A + b[2] * M * A)
y = pm.NegativeBinomial("y", mu=λ, alpha=alpha, observed=S, dims="obs_idx")
idata = pm.sample()
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (4 chains in 4 jobs)
NUTS: [intercept, slopes, alpha]
Sampling 4 chains for 1_000 tune and 1_000 draw iterations (4_000 + 4_000 draws total) took 11 seconds.
查看结果#
az.plot_trace(idata, compact=False);
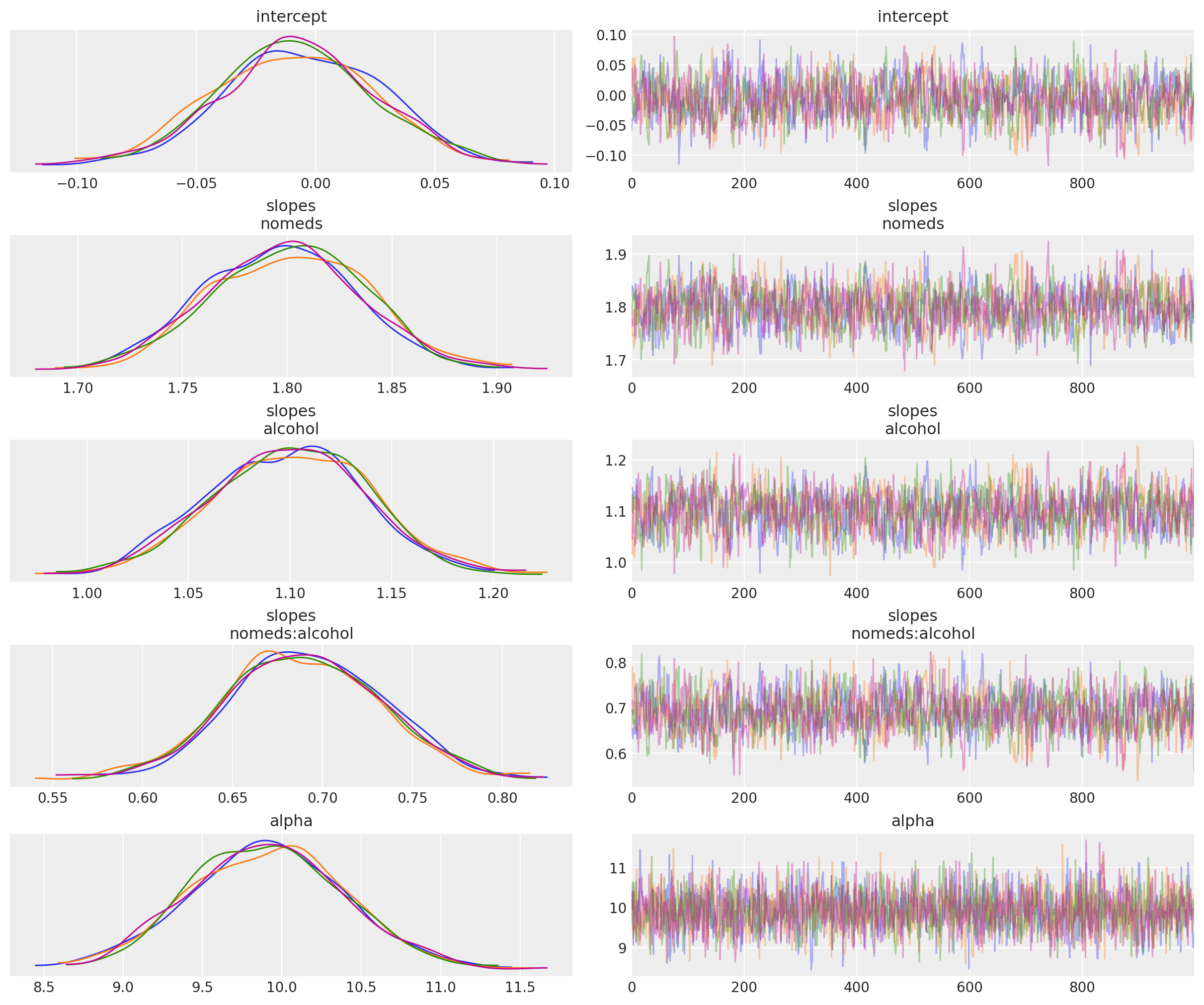
# Transform coefficients to recover parameter values
az.summary(np.exp(idata.posterior), kind="stats", var_names=["intercept", "slopes"])
| mean | sd | hdi_3% | hdi_97% | |
|---|---|---|---|---|
| intercept | 0.993 | 0.033 | 0.935 | 1.056 |
| slopes[nomeds] | 6.048 | 0.221 | 5.635 | 6.438 |
| slopes[alcohol] | 3.006 | 0.115 | 2.789 | 3.215 |
| slopes[nomeds:alcohol] | 1.995 | 0.086 | 1.842 | 2.163 |
az.summary(idata.posterior, kind="stats", var_names="alpha")
| mean | sd | hdi_3% | hdi_97% | |
|---|---|---|---|---|
| alpha | 9.909 | 0.487 | 9.05 | 10.887 |
均值接近我们在生成数据时指定的值:
基础利率是一个常数 1。
饮酒使基础率增加了两倍。
不服用抗组胺药会使基础率增加6倍。
饮酒且不服用抗组胺药会使预期发生率翻倍。如果它们是独立的,那么同时进行这两项会使基础发生率增加3*6=18倍,但实际上基础发生率增加了3*6*2=36倍。
最后,nsneeze_alpha 的平均值也非常接近其真实值 10。
另请参阅,bambi的负二项式示例以供进一步参考。
许可证声明#
本示例库中的所有笔记本均在MIT许可证下提供,该许可证允许修改和重新分发,前提是保留版权和许可证声明。
引用 PyMC 示例#
要引用此笔记本,请使用Zenodo为pymc-examples仓库提供的DOI。
重要
许多笔记本是从其他来源改编的:博客、书籍……在这种情况下,您应该引用原始来源。
同时记得引用代码中使用的相关库。
这是一个BibTeX的引用模板:
@incollection{citekey,
author = "<notebook authors, see above>",
title = "<notebook title>",
editor = "PyMC Team",
booktitle = "PyMC examples",
doi = "10.5281/zenodo.5654871"
}
渲染后可能看起来像: