import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pymc3 as pm
import theano
from scipy.integrate import odeint
from theano import *
THEANO_FLAGS = "optimizer=fast_compile"
print(f"PyMC3 Version: {pm.__version__}")
PyMC3 Version: 3.11.0
Lotka-Volterra 手动梯度#
数学模型广泛应用于各种科学和工程领域,用于模拟物理变量的时间演变。这些数学模型通常被描述为常微分方程(ODE),其特征包括模型结构——动力学变量的函数——和模型参数。然而,对于绝大多数实际感兴趣的系统来说,必须从实验观察中推断出模型参数和适当的模型结构。这些实验数据通常显得稀缺且不完整。此外,大量被描述为动力系统的模型显示出松散性特征(参见Gutenkunst等人,2007),并且具有不可识别的参数组合。从实验数据中推断模型参数和结构的这一任务对于可靠地分析动力系统的行为并在此类系统的复杂性所带来的困难面前做出可信的预测至关重要。此外,任何未来的模型预测都应该包含并传播模型参数和/或结构中的变异性和不确定性。因此,推断方法还必须能够量化并传播上述不确定性,从模型描述到模型预测。作为一种自然选择来处理不确定性,至少在参数方面,贝叶斯推断越来越多地被用于将ODE模型拟合到实验数据(Mark Girolami,2008)。然而,由于我上面指出的一些困难,使用贝叶斯推断拟合ODE模型是一项具有挑战性的任务。在本教程中,我将接受这一挑战,并展示如何潜在地使用PyMC3来实现这一目的。
我必须指出,模型拟合(推断未知参数)只是建模者为了更深入地理解物理过程而必须完成的众多关键任务之一。然而,成功完成这项任务至关重要,而这正是PyMC3以及概率编程(ppl)在一般情况下极其有用的地方。建模者可以充分利用PyMC3提供的各种采样器和分布来自动化推断过程。
在本教程中,我将重点放在拟合练习上,即根据一些带有噪声的实验时间序列估计参数的后验分布。
ODE参数的贝叶斯推断#
我首先介绍用于推断耦合非线性ODE的贝叶斯框架,定义为
考虑一组在\(T\)个实验时间点上观测到的\(K\)个状态的噪声实验观测值\(\boldsymbol{Y} \in \mathbb{R}^{T\times K}\)。我们可以得到似然\(p(\boldsymbol{Y}|\boldsymbol{X})\),其中我使用符号\(\boldsymbol{X}:=\boldsymbol{X}(\boldsymbol{\theta}, \mathbf{x_0})\),并结合参数的先验分布\(p(\boldsymbol{\theta})\),使用贝叶斯定理,得到后验分布为
在本教程中,我选择了两个常微分方程:
我将展示两种独特的方法(NUTS 和 SMC 步方法),这些方法由 PyMC3 支持,用于估计这些模型中的未知参数。
Lotka-Volterra 捕食者-猎物模型#
Lotka Volterra模型描述了一个生态系统,用于描述捕食者和猎物种群之间的相互作用。这个由以下ODE给出的模型展示了极限环行为,并且经常被用于基准测试贝叶斯推理方法。\(\boldsymbol{\theta}=(\alpha,\beta,\gamma,\delta, x(0),y(0))\)是我们希望从状态向量\(X(t)=(x(t),y(t))\)的实验观察中推断出的未知参数集合,分别表示猎物和捕食者的浓度。\(x(0), y(0)\)是求解ODE所需的初始状态值,也被视为未知量。捕食者-猎物模型最近被用来展示NUTS采样器以及Stan ppl在ODE模型推理中的适用性。我将紧密跟随这个Stan教程,因此我将完全按照Stan教程中的方式设置这个模型及其相关的推理问题(包括数据)。让我首先写下使用SciPy的odeint来求解这个ODE的代码。请注意,本教程中的方法并不局限于或绑定于odeint。这里我选择odeint只是为了保持在PyMC3的依赖项(在这种情况下是SciPy)范围内。
class LotkaVolterraModel:
def __init__(self, y0=None):
self._y0 = y0
def simulate(self, parameters, times):
alpha, beta, gamma, delta, Xt0, Yt0 = [x for x in parameters]
def rhs(y, t, p):
X, Y = y
dX_dt = alpha * X - beta * X * Y
dY_dt = -gamma * Y + delta * X * Y
return dX_dt, dY_dt
values = odeint(rhs, [Xt0, Yt0], times, (parameters,))
return values
ode_model = LotkaVolterraModel()
处理ODE梯度#
NUTS 需要目标密度对未知参数的对数梯度,\(\nabla_{\boldsymbol{\theta}}p(\boldsymbol{\theta}|\boldsymbol{Y})\),可以使用微分的链式法则进行计算,即
关于其参数的ODE的梯度,即术语 \(\frac{\partial \boldsymbol{X}}{\partial \boldsymbol{\theta}}\),可以通过局部敏感性分析获得,尽管这不是获得梯度的唯一方法。然而,就像求解一个ODE(准确地说是非线性ODE)一样,梯度的评估也只能通过某种数值方法来实现,比如说著名的用于非刚性ODE的Runge-Kutta方法。PyMC3使用Theano作为自动微分引擎,因此所有模型都是通过拼接Theano支持的基本操作(Ops)来实现的。即使要扩展PyMC3,我们也需要构建可以表示为Theano的Ops的符号组合的模型。然而,如果我们退后一步思考Theano,显然无论是ODE的解还是其关于参数的梯度都不能表示为Theano基本Ops的符号组合。因此,从Theano的角度来看,ODE(以及任何其他形式的非线性微分方程)是一个不可微分的黑箱函数。然而,有人可能会争辩说,如果一个数值方法在Theano中被编码(比如说使用 scan Op),那么就有可能符号化地表示评估ODE状态的数值方法,然后我们可以轻松地使用Theano的自动微分引擎通过数值求解器本身来获得梯度。我想指出的是,前者,即获得解,确实可以通过这种方式实现,但获得的梯度可能会有误差。此外,这涉及到完全“重新发明轮子”,因为人们将不得不从头开始在Theano中再次实现已有几十年历史的复杂数值算法。
因此,在本教程中,我将介绍一种替代方法,即定义新的自定义 Theano Ops,扩展 Theano,这将同时封装数值解和向量-矩阵乘积,\( \frac{\partial p(\boldsymbol{\theta}|\boldsymbol{Y})}{\partial \boldsymbol{X}}^T \frac{\partial \boldsymbol{X}}{\partial \boldsymbol{\theta}}\),在自动微分文献中通常称为向量-雅可比积 (VJP)。我想在这里指出,在非线性 ODE 的背景下,术语雅可比矩阵用于表示 ODE 动力学 \(\boldsymbol{f}\) 相对于 ODE 状态 \(X(t)\) 的梯度。因此,为了避免混淆,从现在开始我将使用术语向量-敏感度积 (VSP) 来表示与术语 VJP 相同的量。
我将首先介绍前向灵敏度分析。
ODE敏感性分析#
对于一个耦合的ODE系统 \(\frac{d X(t)}{dt} = \boldsymbol{f}(X(t),\boldsymbol{\theta})\),解对参数的局部敏感性定义为解随参数变化而变化的程度,即第 \(k\) 个状态的敏感性简单来说就是其相对于第 \(d\) 个参数的梯度的时间演化。这个量,记为 \(Z_{kd}(t)\),由以下公式给出
使用前向敏感性分析,我们可以在每个时间点同时获得状态 \(X(t)\) 及其关于参数的导数,作为初始值问题的解,通过将原始ODE系统与敏感性方程 \(Z_{kd}\) 相结合。然后可以使用所选的数值方法一起求解增广的ODE系统 \(\big(X(t), Z(t)\big)\)。增广的ODE系统需要敏感性方程的初始值。除了那些寻求状态对其自身初始值的敏感性的情况,即 \( \frac{\partial X_k(t)}{\partial X_k (0)} =1 \),所有这些都应该设置为零。请注意,为了求解这个增广系统,我们必须进行繁琐的过程来推导 \( \frac{\partial f_k}{\partial X_i (t)}\),也称为ODE的雅可比矩阵,以及 \(\frac{\partial f_k}{\partial \theta_d}\) 项。幸运的是,许多ODE求解器在用户请求时会计算这些项并求解增广系统。一个例子是 SUNDIAL CVODES求解器套件。可以在 这里 找到CVODES的Python包装器。
然而,在本教程中,我将手动推导上述术语,并在以下代码块中解决Lotka-Volterra ODEs及其敏感性。下面的函数jac和dfdp分别计算Lotka-Volterra模型中的\( \frac{\partial f_k}{\partial X_i (t)}\)和\(\frac{\partial f_k}{\partial \theta_d}\)。为了方便起见,我将敏感性方程转换为矩阵形式。在这里,我扩展了上面的求解器代码片段,以在需要时包含敏感性。
n_states = 2
n_odeparams = 4
n_ivs = 2
class LotkaVolterraModel:
def __init__(self, n_states, n_odeparams, n_ivs, y0=None):
self._n_states = n_states
self._n_odeparams = n_odeparams
self._n_ivs = n_ivs
self._y0 = y0
def simulate(self, parameters, times):
return self._simulate(parameters, times, False)
def simulate_with_sensitivities(self, parameters, times):
return self._simulate(parameters, times, True)
def _simulate(self, parameters, times, sensitivities):
alpha, beta, gamma, delta, Xt0, Yt0 = [x for x in parameters]
def r(y, t, p):
X, Y = y
dX_dt = alpha * X - beta * X * Y
dY_dt = -gamma * Y + delta * X * Y
return dX_dt, dY_dt
if sensitivities:
def jac(y):
X, Y = y
ret = np.zeros((self._n_states, self._n_states))
ret[0, 0] = alpha - beta * Y
ret[0, 1] = -beta * X
ret[1, 0] = delta * Y
ret[1, 1] = -gamma + delta * X
return ret
def dfdp(y):
X, Y = y
ret = np.zeros(
(self._n_states, self._n_odeparams + self._n_ivs)
) # except the following entries
ret[0, 0] = (
X # \frac{\partial [\alpha X - \beta XY]}{\partial \alpha}, and so on...
)
ret[0, 1] = -X * Y
ret[1, 2] = -Y
ret[1, 3] = X * Y
return ret
def rhs(y_and_dydp, t, p):
y = y_and_dydp[0 : self._n_states]
dydp = y_and_dydp[self._n_states :].reshape(
(self._n_states, self._n_odeparams + self._n_ivs)
)
dydt = r(y, t, p)
d_dydp_dt = np.matmul(jac(y), dydp) + dfdp(y)
return np.concatenate((dydt, d_dydp_dt.reshape(-1)))
y0 = np.zeros((2 * (n_odeparams + n_ivs)) + n_states)
y0[6] = 1.0 # \frac{\partial [X]}{\partial Xt0} at t==0, and same below for Y
y0[13] = 1.0
y0[0:n_states] = [Xt0, Yt0]
result = odeint(rhs, y0, times, (parameters,), rtol=1e-6, atol=1e-5)
values = result[:, 0 : self._n_states]
dvalues_dp = result[:, self._n_states :].reshape(
(len(times), self._n_states, self._n_odeparams + self._n_ivs)
)
return values, dvalues_dp
else:
values = odeint(r, [Xt0, Yt0], times, (parameters,), rtol=1e-6, atol=1e-5)
return values
ode_model = LotkaVolterraModel(n_states, n_odeparams, n_ivs)
对于这个模型,我分别将相对容差和绝对容差设置为 \(10^{-6}\) 和 \(10^{-5}\),这是根据Stan教程中的建议。这将产生足够准确的解。进一步减小容差将提高准确性,但代价是增加计算时间。关于选择和使用数值方法来求解ODE的深入讨论超出了本教程的范围。然而,我必须指出,ODE求解器的不准确性确实会影响似然性,从而影响推断。对于刚性系统,这种情况更为明显。我建议感兴趣的读者阅读这篇精彩的博客文章,其中详细讨论了这种影响,适用于心脏ODE模型。还有一个新兴的不确定性量化领域,它解决了由于数值算法的不精确性而产生的噪声问题,概率数值。这确实是一个优雅的框架,可以在考虑数值ODE求解器产生的误差的同时进行推断。
自定义 ODE Op#
为了定义自定义的 Op,我编写了两个 theano.Op 类 ODEGradop 和 ODEop。ODEop 本质上封装了 ODE 解决方案,并将被 PyMC3 调用。ODEGradop 封装了数值 VSP,然后这个操作在 ODEop 的 grad 方法中使用,以返回 VSP。请注意,我们传入了两个函数:state 和 numpy_vsp 作为相应操作的参数。我将在后面定义这些函数。这些函数作为连接器,使用它们我们将状态和 VSP 的数值解的 Python 代码连接到 Theano 以及 PyMC3。
class ODEGradop(theano.tensor.Op):
def __init__(self, numpy_vsp):
self._numpy_vsp = numpy_vsp
def make_node(self, x, g):
x = theano.tensor.as_tensor_variable(x)
g = theano.tensor.as_tensor_variable(g)
node = theano.Apply(self, [x, g], [g.type()])
return node
def perform(self, node, inputs_storage, output_storage):
x = inputs_storage[0]
g = inputs_storage[1]
out = output_storage[0]
out[0] = self._numpy_vsp(x, g) # get the numerical VSP
class ODEop(theano.tensor.Op):
def __init__(self, state, numpy_vsp):
self._state = state
self._numpy_vsp = numpy_vsp
def make_node(self, x):
x = theano.tensor.as_tensor_variable(x)
return theano.tensor.Apply(self, [x], [x.type()])
def perform(self, node, inputs_storage, output_storage):
x = inputs_storage[0]
out = output_storage[0]
out[0] = self._state(x) # get the numerical solution of ODE states
def grad(self, inputs, output_grads):
x = inputs[0]
g = output_grads[0]
grad_op = ODEGradop(self._numpy_vsp) # pass the VSP when asked for gradient
grad_op_apply = grad_op(x, g)
return [grad_op_apply]
我必须指出,按照我上面定义自定义ODE Ops的方式,可能会导致ODE对相同的参数值求解两次,一次用于状态,另一次用于VSP。为了避免这种行为,我编写了一个辅助类来阻止这种双重求解。
class solveCached:
def __init__(self, times, n_params, n_outputs):
self._times = times
self._n_params = n_params
self._n_outputs = n_outputs
self._cachedParam = np.zeros(n_params)
self._cachedSens = np.zeros((len(times), n_outputs, n_params))
self._cachedState = np.zeros((len(times), n_outputs))
def __call__(self, x):
if np.all(x == self._cachedParam):
state, sens = self._cachedState, self._cachedSens
else:
state, sens = ode_model.simulate_with_sensitivities(x, times)
return state, sens
times = np.arange(0, 21) # number of measurement points (see below)
cached_solver = solveCached(times, n_odeparams + n_ivs, n_states)
ODE状态与VSP评估#
大多数实际感兴趣的ODE系统将具有多个状态,因此求解器的输出,我迄今为止表示为\(\boldsymbol{X}\),对于具有\(K\)个状态并在\(T\)个时间点上求解的系统,将是一个\(T \times K\)维矩阵。对于Lotka-Volterra模型,该矩阵的列表示各个物种浓度随时间的变化。我将此矩阵展平为一个\(TK\)维向量\(vec(\boldsymbol{X})\),并相应地重新排列敏感性以获得所需的向量-矩阵乘积。此时,按照这里的描述测试自定义Op是有益的。
def state(x):
State, Sens = cached_solver(np.array(x, dtype=np.float64))
cached_solver._cachedState, cached_solver._cachedSens, cached_solver._cachedParam = (
State,
Sens,
x,
)
return State.reshape((2 * len(State),))
def numpy_vsp(x, g):
numpy_sens = cached_solver(np.array(x, dtype=np.float64))[1].reshape(
(n_states * len(times), len(x))
)
return numpy_sens.T.dot(g)
哈德逊湾公司数据#
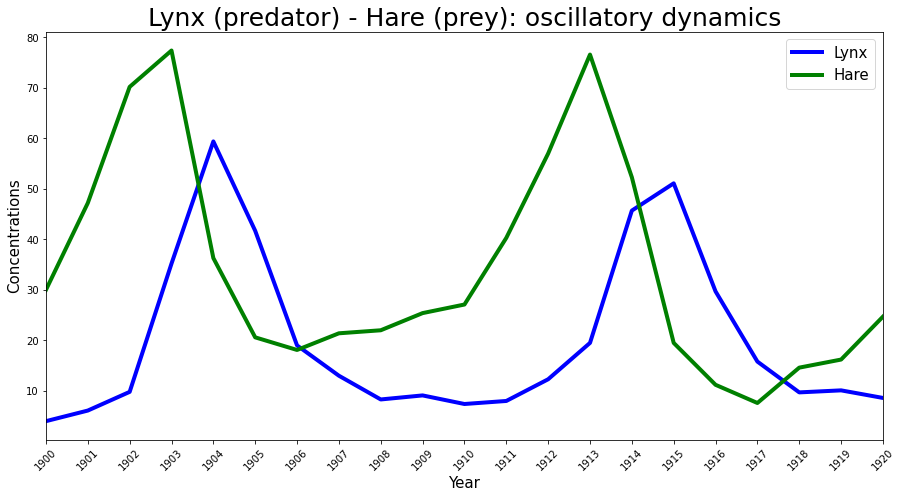
Lotka-Volterra捕食者-猎物模型之前已被成功用于解释捕食者和猎物的自然种群动态,例如哈德逊湾公司的猞猁和雪鞋兔数据。这是与Stan示例中使用的相同数据(已在此处共享),因此我将使用此数据集作为实验观察值\(\boldsymbol{Y}(t)\)来推断参数。
Year = np.arange(1900, 1921, 1)
# fmt: off
Lynx = np.array([4.0, 6.1, 9.8, 35.2, 59.4, 41.7, 19.0, 13.0, 8.3, 9.1, 7.4,
8.0, 12.3, 19.5, 45.7, 51.1, 29.7, 15.8, 9.7, 10.1, 8.6])
Hare = np.array([30.0, 47.2, 70.2, 77.4, 36.3, 20.6, 18.1, 21.4, 22.0, 25.4,
27.1, 40.3, 57.0, 76.6, 52.3, 19.5, 11.2, 7.6, 14.6, 16.2, 24.7])
# fmt: on
plt.figure(figsize=(15, 7.5))
plt.plot(Year, Lynx, color="b", lw=4, label="Lynx")
plt.plot(Year, Hare, color="g", lw=4, label="Hare")
plt.legend(fontsize=15)
plt.xlim([1900, 1920])
plt.xlabel("Year", fontsize=15)
plt.ylabel("Concentrations", fontsize=15)
plt.xticks(Year, rotation=45)
plt.title("Lynx (predator) - Hare (prey): oscillatory dynamics", fontsize=25);
概率模型#
我现在已经获得了在PyMC3中定义概率模型所需的所有成分。正如我之前提到的,我将使用与Stan示例中完全相同的似然函数和先验来设置概率模型。观测数据定义如下:
其中 \(\eta(t)\) 被假设为具有未知标准差 \(\sigma\) 的零均值独立同分布高斯噪声,需要进行估计。上述乘法(在自然尺度上)噪声模型编码了对数正态分布作为似然:
然后在参数上放置以下先验:
为了直观地解释(为了简洁起见,我省略了这一点)关于先验选择以及似然模型的内容,我建议参考上面提到的Stan示例。上述概率模型在PyMC3中定义如下。请注意,展平的状态向量被重塑以匹配数据的维度。
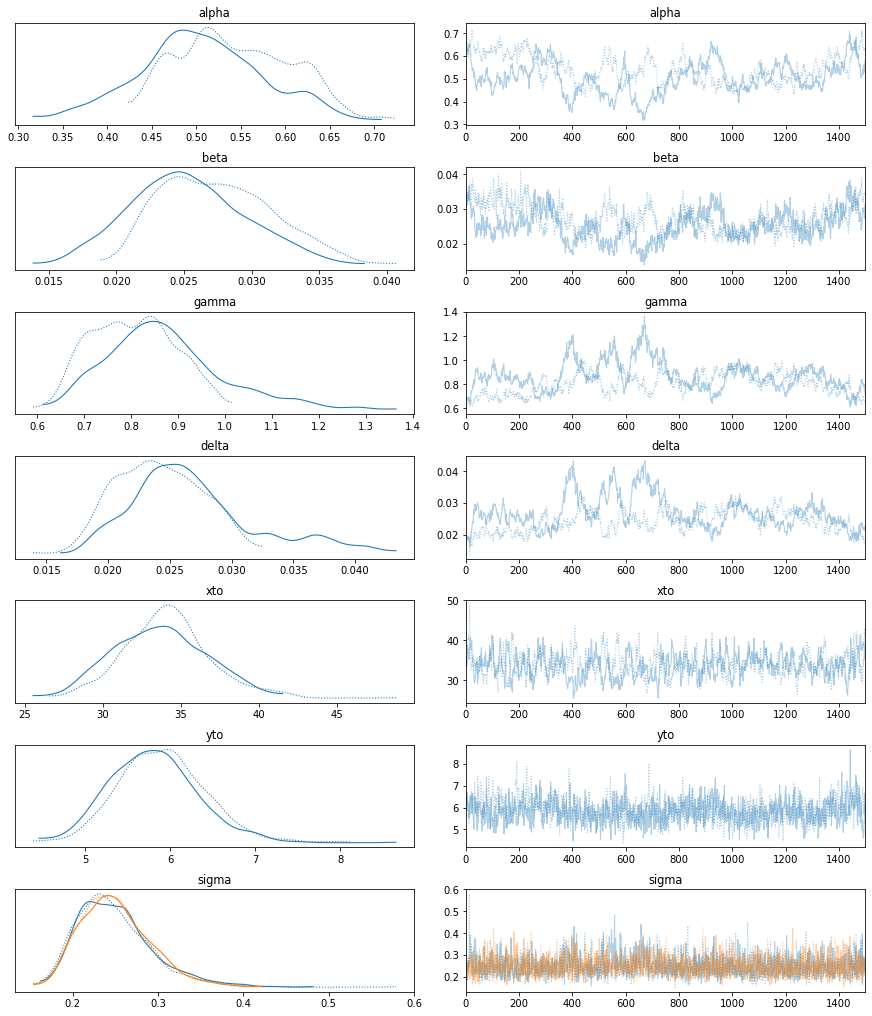
最后,我使用 pm.sample 方法默认运行 NUTS,并从后验中获得 \(1500\) 个预热后的样本。
theano.config.exception_verbosity = "high"
theano.config.floatX = "float64"
# Define the data matrix
Y = np.vstack((Hare, Lynx)).T
# Now instantiate the theano custom ODE op
my_ODEop = ODEop(state, numpy_vsp)
# The probabilistic model
with pm.Model() as LV_model:
# Priors for unknown model parameters
alpha = pm.Normal("alpha", mu=1, sigma=0.5)
beta = pm.Normal("beta", mu=0.05, sigma=0.05)
gamma = pm.Normal("gamma", mu=1, sigma=0.5)
delta = pm.Normal("delta", mu=0.05, sigma=0.05)
xt0 = pm.Lognormal("xto", mu=np.log(10), sigma=1)
yt0 = pm.Lognormal("yto", mu=np.log(10), sigma=1)
sigma = pm.Lognormal("sigma", mu=-1, sigma=1, shape=2)
# Forward model
all_params = pm.math.stack([alpha, beta, gamma, delta, xt0, yt0], axis=0)
ode_sol = my_ODEop(all_params)
forward = ode_sol.reshape(Y.shape)
# Likelihood
Y_obs = pm.Lognormal("Y_obs", mu=pm.math.log(forward), sigma=sigma, observed=Y)
trace = pm.sample(1500, init="jitter+adapt_diag", cores=1)
trace["diverging"].sum()
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Initializing NUTS failed. Falling back to elementwise auto-assignment.
Sequential sampling (2 chains in 1 job)
CompoundStep
>CompoundStep
>>Slice: [yto]
>>Slice: [xto]
>>Slice: [delta]
>>Slice: [gamma]
>>Slice: [beta]
>>Slice: [alpha]
>NUTS: [sigma]
/Users/CloudChaoszero/opt/anaconda3/envs/pymc3-dev-py38/lib/python3.8/site-packages/scipy/integrate/odepack.py:247: ODEintWarning: Excess work done on this call (perhaps wrong Dfun type). Run with full_output = 1 to get quantitative information.
warnings.warn(warning_msg, ODEintWarning)
Sampling 2 chains for 1_000 tune and 1_500 draw iterations (2_000 + 3_000 draws total) took 1327 seconds.
The rhat statistic is larger than 1.05 for some parameters. This indicates slight problems during sampling.
The estimated number of effective samples is smaller than 200 for some parameters.
0
with LV_model:
az.plot_trace(trace);
import pandas as pd
summary = az.summary(trace)
STAN_mus = [0.549, 0.028, 0.797, 0.024, 33.960, 5.949, 0.248, 0.252]
STAN_sds = [0.065, 0.004, 0.091, 0.004, 2.909, 0.533, 0.045, 0.044]
summary["STAN_mus"] = pd.Series(np.array(STAN_mus), index=summary.index)
summary["STAN_sds"] = pd.Series(np.array(STAN_sds), index=summary.index)
summary
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_mean | ess_sd | ess_bulk | ess_tail | r_hat | STAN_mus | STAN_sds | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| alpha | 0.524 | 0.069 | 0.403 | 0.653 | 0.017 | 0.012 | 16.0 | 16.0 | 16.0 | 15.0 | 1.12 | 0.549 | 0.065 |
| beta | 0.026 | 0.004 | 0.019 | 0.035 | 0.001 | 0.001 | 17.0 | 17.0 | 17.0 | 17.0 | 1.11 | 0.028 | 0.004 |
| gamma | 0.837 | 0.111 | 0.652 | 1.045 | 0.029 | 0.022 | 14.0 | 14.0 | 15.0 | 15.0 | 1.12 | 0.797 | 0.091 |
| delta | 0.026 | 0.005 | 0.017 | 0.034 | 0.001 | 0.001 | 12.0 | 11.0 | 16.0 | 17.0 | 1.12 | 0.024 | 0.004 |
| xto | 33.921 | 2.912 | 28.246 | 39.126 | 0.179 | 0.127 | 264.0 | 264.0 | 257.0 | 539.0 | 1.02 | 33.960 | 2.909 |
| yto | 5.857 | 0.515 | 4.873 | 6.783 | 0.039 | 0.028 | 174.0 | 174.0 | 175.0 | 1060.0 | 1.01 | 5.949 | 0.533 |
| sigma[0] | 0.249 | 0.044 | 0.173 | 0.330 | 0.002 | 0.001 | 683.0 | 661.0 | 789.0 | 896.0 | 1.00 | 0.248 | 0.045 |
| sigma[1] | 0.250 | 0.041 | 0.178 | 0.327 | 0.001 | 0.001 | 1793.0 | 1756.0 | 1864.0 | 1949.0 | 1.00 | 0.252 | 0.044 |
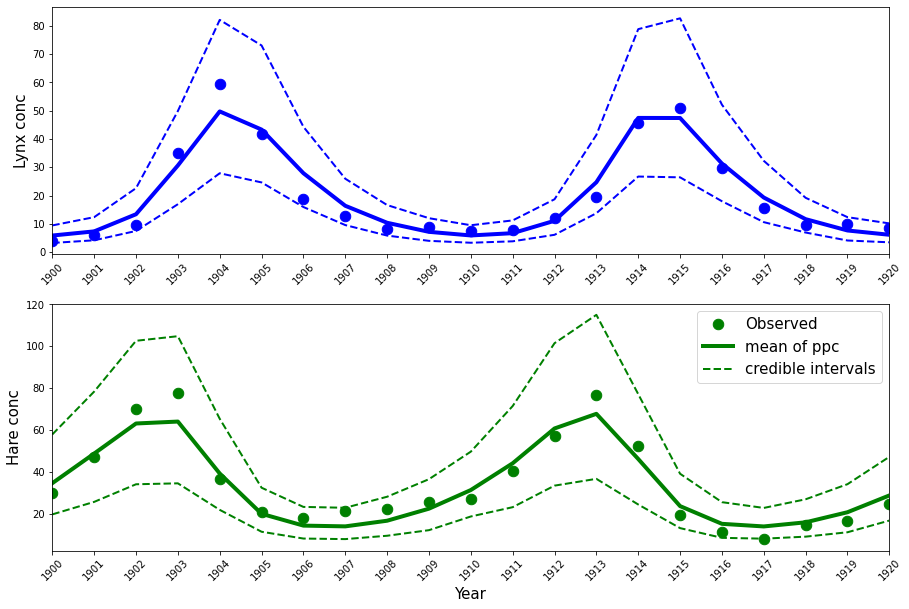
这些估计值与Stan教程中得到的估计值几乎相同(见上表最后两列),这也是我们可以预期的结果。后验预测可以通过以下方式绘制。
ppc_samples = pm.sample_posterior_predictive(trace, samples=1000, model=LV_model)["Y_obs"]
mean_ppc = ppc_samples.mean(axis=0)
CriL_ppc = np.percentile(ppc_samples, q=2.5, axis=0)
CriU_ppc = np.percentile(ppc_samples, q=97.5, axis=0)
/Users/CloudChaoszero/Documents/Projects-Dev/pymc3/pymc3/sampling.py:1687: UserWarning: samples parameter is smaller than nchains times ndraws, some draws and/or chains may not be represented in the returned posterior predictive sample
warnings.warn(
plt.figure(figsize=(15, 2 * (5)))
plt.subplot(2, 1, 1)
plt.plot(Year, Lynx, "o", color="b", lw=4, ms=10.5)
plt.plot(Year, mean_ppc[:, 1], color="b", lw=4)
plt.plot(Year, CriL_ppc[:, 1], "--", color="b", lw=2)
plt.plot(Year, CriU_ppc[:, 1], "--", color="b", lw=2)
plt.xlim([1900, 1920])
plt.ylabel("Lynx conc", fontsize=15)
plt.xticks(Year, rotation=45)
plt.subplot(2, 1, 2)
plt.plot(Year, Hare, "o", color="g", lw=4, ms=10.5, label="Observed")
plt.plot(Year, mean_ppc[:, 0], color="g", lw=4, label="mean of ppc")
plt.plot(Year, CriL_ppc[:, 0], "--", color="g", lw=2, label="credible intervals")
plt.plot(Year, CriU_ppc[:, 0], "--", color="g", lw=2)
plt.legend(fontsize=15)
plt.xlim([1900, 1920])
plt.xlabel("Year", fontsize=15)
plt.ylabel("Hare conc", fontsize=15)
plt.xticks(Year, rotation=45);
使用SMC高效探索后验分布景观#
在几篇论文中已经指出,ODE的复杂非线性动力学导致的后验景观对于许多MCMC采样器来说极难高效导航。因此,最近利用后验曲面曲率信息来构建强大的几何感知采样器(Mark Girolami 和 Ben Calderhead, 2011),这些采样器在ODE推断问题中表现非常出色。另一组想法建议将复杂的推断任务分解为一系列更简单的任务。本质上,这个想法是使用顺序重要性采样从一个人工的、逐渐复杂的分布序列中进行采样,其中序列中的第一个分布是容易采样的先验分布,而序列中的最后一个分布是实际的复杂目标分布。相关的重要性分布是通过使用马尔可夫核(例如MH核)移动前一步采样的粒子集来构建的。
构建分布序列的一个简单方法是使用一个温度 \(\beta\),它从 \(0\) 缓慢上升到 \(1\)。使用这个温度变量 \(\beta\),我们可以写下退火中间分布为
执行这些人工序列分布的顺序重要性采样的采样器,以避免直接从 \(p(\boldsymbol{\theta}|\boldsymbol{y})\) 采样的困难任务,被称为顺序蒙特卡罗(SMC)采样器(P Del Moral 等,2006)。这些采样器的性能对温度计划的选择非常敏感,即用户定义的 \(\beta\) 在 \(0\) 和 \(1\) 之间的递增值集合。幸运的是,PyMC3 提供了一个 SMC 采样器版本(Jianye Ching 和 Yi-Chu Chen,2007),它可以自动确定这个温度计划。此外,PyMC3 的 SMC 采样器不需要对数目标密度的梯度。因此,使用这个采样器进行 ODE 模型推断非常容易。在下一个示例中,我将应用这个 SMC 采样器来估计 Fitzhugh-Nagumo 模型的参数。
Fitzhugh-Nagumo 模型#
由以下公式给出的Fitzhugh-Nagumo模型:
class FitzhughNagumoModel:
def __init__(self, times, y0=None):
self._y0 = np.array([-1, 1], dtype=np.float64)
self._times = times
def _simulate(self, parameters, times):
a, b, c = [float(x) for x in parameters]
def rhs(y, t, p):
V, R = y
dV_dt = (V - V**3 / 3 + R) * c
dR_dt = (V - a + b * R) / -c
return dV_dt, dR_dt
values = odeint(rhs, self._y0, times, (parameters,), rtol=1e-6, atol=1e-6)
return values
def simulate(self, x):
return self._simulate(x, self._times)
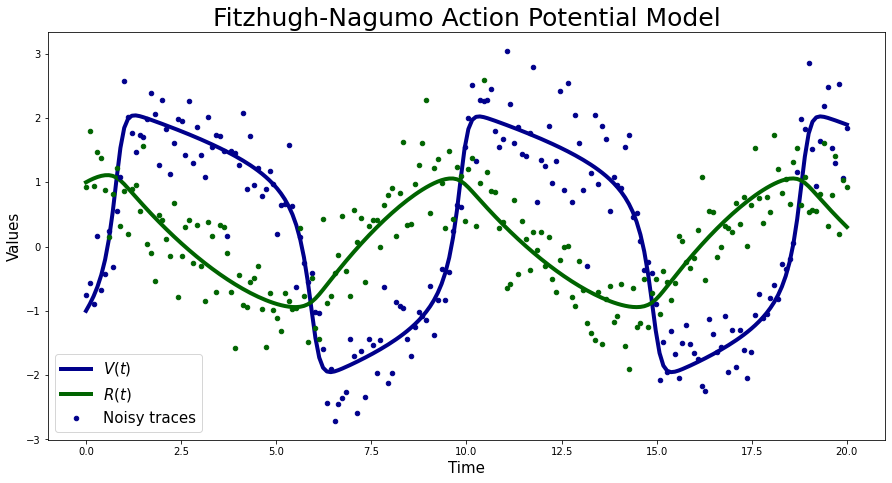
模拟数据#
对于这个例子,我将使用模拟数据,即我将从上述定义的前向模型生成带有噪声的轨迹,参数 \(\theta\) 分别设置为 \((0.2,0.2,3)\),并受到独立同分布的高斯噪声的干扰,标准差为 \(\sigma=0.5\)。初始值分别设置为 \(V(0)=-1\) 和 \(R(0)=1\)。再次参考 Mark Girolami 和 Ben Calderhead, 2011,我将假设初始值是已知的。这些参数值将模型推向振荡状态。
n_states = 2
n_times = 200
true_params = [0.2, 0.2, 3.0]
noise_sigma = 0.5
FN_solver_times = np.linspace(0, 20, n_times)
ode_model = FitzhughNagumoModel(FN_solver_times)
sim_data = ode_model.simulate(true_params)
np.random.seed(42)
Y_sim = sim_data + np.random.randn(n_times, n_states) * noise_sigma
plt.figure(figsize=(15, 7.5))
plt.plot(FN_solver_times, sim_data[:, 0], color="darkblue", lw=4, label=r"$V(t)$")
plt.plot(FN_solver_times, sim_data[:, 1], color="darkgreen", lw=4, label=r"$R(t)$")
plt.plot(FN_solver_times, Y_sim[:, 0], "o", color="darkblue", ms=4.5, label="Noisy traces")
plt.plot(FN_solver_times, Y_sim[:, 1], "o", color="darkgreen", ms=4.5)
plt.legend(fontsize=15)
plt.xlabel("Time", fontsize=15)
plt.ylabel("Values", fontsize=15)
plt.title("Fitzhugh-Nagumo Action Potential Model", fontsize=25);
使用 Theano @as_op 定义一个不可微的黑色盒子操作#
记住,我提到过SMC采样器不需要梯度,顺便说一下,Metropolis-Hastings和Slice采样器也是如此,这些采样器在PyMC3中也得到了支持。对于所有这些不需要梯度的采样器,我将展示一种简单快捷的方法来包装正向模型,即在Theano中的ODE解。我们所要做的就是简单地使用装饰器as_op,它将一个python函数转换为一个基本的Theano Op。我们还通过使用as_op装饰器告诉Theano我们有三个参数,每个参数都是一个Theano标量。输出则是一个Theano矩阵,其列是状态向量。
import theano.tensor as tt
from theano.compile.ops import as_op
@as_op(itypes=[tt.dscalar, tt.dscalar, tt.dscalar], otypes=[tt.dmatrix])
def th_forward_model(param1, param2, param3):
param = [param1, param2, param3]
th_states = ode_model.simulate(param)
return th_states
生成模型#
由于我使用独立同分布的高斯噪声破坏了原始轨迹,因此似然函数由以下公式给出:
注意在这个例子中我是如何使用start参数的。就像pm.sample一样,pm.sample_smc也有许多设置,但我发现对于像这样的简单模型,默认设置已经足够好了。
draws = 1000
with pm.Model() as FN_model:
a = pm.Gamma("a", alpha=2, beta=1)
b = pm.Normal("b", mu=0, sigma=1)
c = pm.Uniform("c", lower=0.1, upper=10)
sigma = pm.HalfNormal("sigma", sigma=1)
forward = th_forward_model(a, b, c)
cov = np.eye(2) * sigma**2
Y_obs = pm.MvNormal("Y_obs", mu=forward, cov=cov, observed=Y_sim)
startsmc = {v.name: np.random.uniform(1e-3, 2, size=draws) for v in FN_model.free_RVs}
trace_FN = pm.sample_smc(draws, start=startsmc)
Initializing SMC sampler...
Sampling 2 chains in 2 jobs
Stage: 0 Beta: 0.009
Stage: 1 Beta: 0.029
Stage: 2 Beta: 0.043
Stage: 3 Beta: 0.054
Stage: 4 Beta: 0.065
Stage: 5 Beta: 0.087
Stage: 6 Beta: 0.118
Stage: 7 Beta: 0.163
Stage: 8 Beta: 0.229
Stage: 9 Beta: 0.317
Stage: 10 Beta: 0.456
Stage: 11 Beta: 0.645
Stage: 12 Beta: 0.914
Stage: 13 Beta: 1.000
Stage: 0 Beta: 0.009
Stage: 1 Beta: 0.031
Stage: 2 Beta: 0.046
Stage: 3 Beta: 0.056
Stage: 4 Beta: 0.067
Stage: 5 Beta: 0.088
Stage: 6 Beta: 0.122
Stage: 7 Beta: 0.169
Stage: 8 Beta: 0.235
Stage: 9 Beta: 0.339
Stage: 10 Beta: 0.494
Stage: 11 Beta: 0.695
Stage: 12 Beta: 0.999
Stage: 13 Beta: 1.000
az.plot_posterior(trace_FN, kind="hist", bins=30, color="seagreen");
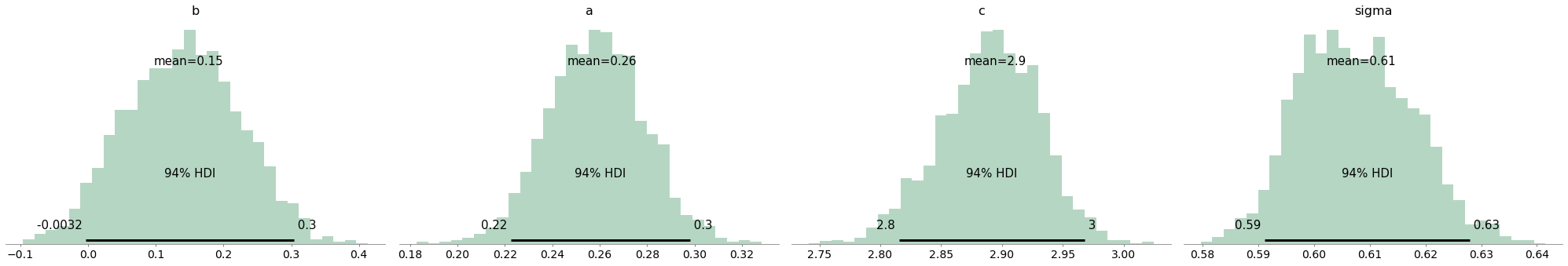
推理总结#
使用 pm.SMC,我能获得与几何MCMC采样器相似的性能吗(参见 Mark Girolami 和 Ben Calderhead, 2011)?我认为可以!
results = [
az.summary(trace_FN, ["a"]),
az.summary(trace_FN, ["b"]),
az.summary(trace_FN, ["c"]),
az.summary(trace_FN, ["sigma"]),
]
results = pd.concat(results)
true_params.append(noise_sigma)
results["True values"] = pd.Series(np.array(true_params), index=results.index)
true_params.pop()
results
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_mean | ess_sd | ess_bulk | ess_tail | r_hat | True values | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| a | 0.261 | 0.020 | 0.223 | 0.298 | 0.007 | 0.005 | 8.0 | 8.0 | 8.0 | 108.0 | 1.18 | 0.2 |
| b | 0.149 | 0.084 | -0.003 | 0.304 | 0.003 | 0.002 | 1014.0 | 654.0 | 1009.0 | 1772.0 | 1.01 | 0.2 |
| c | 2.895 | 0.041 | 2.815 | 2.968 | 0.016 | 0.012 | 7.0 | 7.0 | 7.0 | 98.0 | 1.22 | 3.0 |
| sigma | 0.609 | 0.010 | 0.591 | 0.628 | 0.005 | 0.004 | 4.0 | 4.0 | 4.0 | 75.0 | 1.38 | 0.5 |
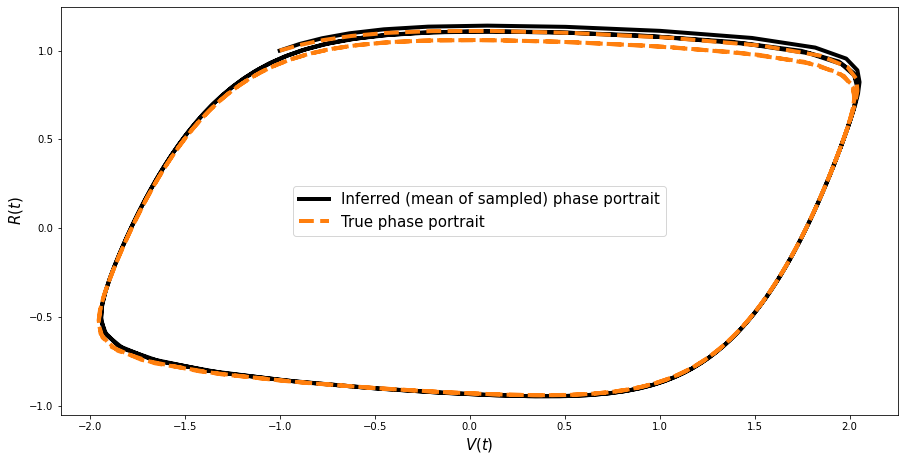
相图的重构#
检查我们能否基于获得的样本重构该模型的(著名)相图是很有益的。
params = np.array([trace_FN.get_values("a"), trace_FN.get_values("b"), trace_FN.get_values("c")]).T
params.shape
new_values = []
for ind in range(len(params)):
ppc_sol = ode_model.simulate(params[ind])
new_values.append(ppc_sol)
new_values = np.array(new_values)
mean_values = np.mean(new_values, axis=0)
plt.figure(figsize=(15, 7.5))
plt.plot(
mean_values[:, 0],
mean_values[:, 1],
color="black",
lw=4,
label="Inferred (mean of sampled) phase portrait",
)
plt.plot(
sim_data[:, 0], sim_data[:, 1], "--", color="#ff7f0e", lw=4, ms=6, label="True phase portrait"
)
plt.legend(fontsize=15)
plt.xlabel(r"$V(t)$", fontsize=15)
plt.ylabel(r"$R(t)$", fontsize=15);
观点#
使用其他一些ODE模型#
我已经尽量保持一切尽可能通用。因此,我的自定义ODE Op、状态和VSP评估器以及缓存的求解器并不与特定的ODE模型绑定。因此,要使用任何其他ODE模型,只需根据其特定的ODE模型实现一个simulate_with_sensitivities方法。
其他形式的微分方程(DDE、DAE、PDE)#
我希望这两个例子已经阐明了PyMC3在拟合ODE模型方面的适用性。尽管ODE是最基本的数学模型组成部分,但确实还存在其他形式的动态系统,如延迟微分方程(DDE)、微分代数方程(DAE)和偏微分方程(PDE),它们的参数估计同样重要。SMC以及PyMC3支持的任何其他非梯度采样器都可以用于拟合所有这些形式的微分方程,当然使用as_op。然而,就像ODE一样,我们可以求解DDE/DAE的增广系统及其敏感性方程。DDE和DAE的敏感性方程可以在最近的一篇论文中找到,C Rackauckas等,2018(方程9和10)。因此,我们可以轻松地将NUTS采样器应用于这些模型。
Stan 已经支持 ODEs#
嗯,有很多问题我认为SMC采样器比NUTS更合适,因此有这个选项是很好的。
模型选择#
自Vladislav Vyshemirsky 和 Mark Girolami, 2008以来的大多数ODE推断文献都推荐使用贝叶斯因子进行模型选择/比较。这涉及到边际似然的计算,这是一个更为复杂的话题,我在此不做任何讨论。幸运的是,SMC采样器作为副产品计算边际似然,因此可以用于获得贝叶斯因子。有关如何在运行SMC采样器后获得边际似然的进一步信息,请参阅PyMC3的其他教程。
由于我们通常将ODE推断框架化为一个回归问题(在大多数情况下,伴随着独立同分布测量噪声假设),我们可以直接使用任何支持的信息准则,例如广泛可用的信息准则(WAIC),而不论用于推断的采样器是什么。有关WAIC的更多信息,请参阅PyMC3的API。
其他AD包#
尽管这是一个小小的偏离主题,但我仍然想指出我对这个问题的观察。我在这里提出的将ODE(也扩展到DDE/DAE)嵌入为自定义Op的方法可以轻松地应用于其他AD包,如TensorFlow和PyTorch。我曾使用TensorFlow的py_func构建了一个自定义的TensorFlow ODE Op,然后在Edward ppl中使用它。我推荐这个教程,给那些对使用Pyro ppl感兴趣的人,用于编写PyTorch扩展。
%load_ext watermark
%watermark -n -u -v -iv -w
Last updated: Sat Mar 13 2021
Python implementation: CPython
Python version : 3.8.6
IPython version : 7.20.0
logging : 0.5.1.2
pandas : 1.2.1
theano : 1.1.2
numpy : 1.20.0
sys : 3.8.6 | packaged by conda-forge | (default, Jan 25 2021, 23:22:12)
[Clang 11.0.1 ]
matplotlib: None
pymc3 : 3.11.0
arviz : 0.11.0
Watermark: 2.1.0