基本多目标功能#
在本教程中,我们将学习如何使用pygmo来解决多目标优化问题。我们假设您已经知道如何编写自己的多目标问题(UDP),因此这里将重点介绍一些旨在简化问题分析的附加功能。
让我们开始定义我们的总体:
>>> from pygmo import *
>>> udp = zdt(prob_id = 1)
>>> pop = population(prob = udp, size = 10, seed = 3453412)
我们在这里使用了ZDT基准测试套件中的第一个问题,该问题在zdt中实现,并且我们创建了一个population,其中包含在边界内随机创建的10个个体。哪些个体属于哪个非支配前沿?我们可以通过运行快速非支配排序算法fast_non_dominated_sorting()立即看到这一点:
>>> ndf, dl, dc, ndl = fast_non_dominated_sorting(pop.get_f())
[array([3, 4, 7, 8, 9], dtype=uint64), array([0, 5, 1, 6], dtype=uint64), array([2], dtype=uint64)]
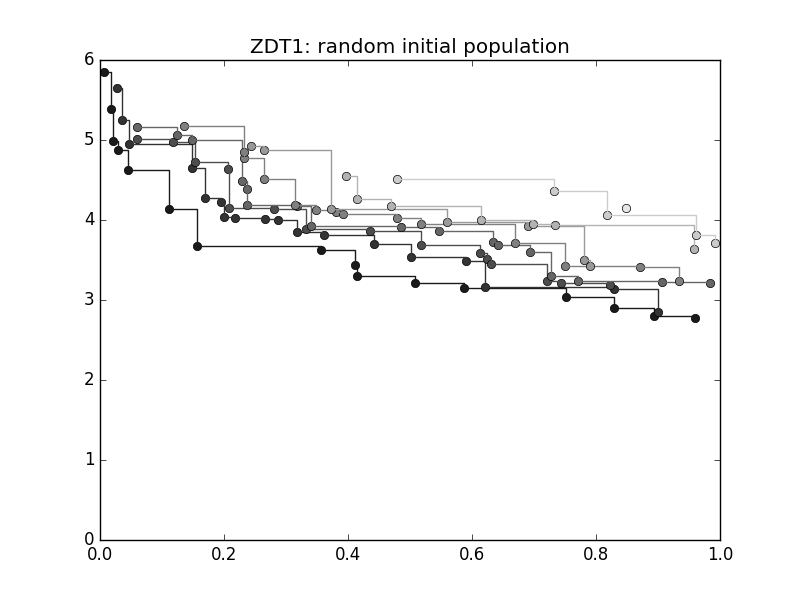
不同非支配前沿的可视化也可以轻松获得。
例如,生成一个包含100个个体的新population:
>>> from matplotlib import pyplot as plt
>>> pop = population(udp, 100)
>>> ax = plot_non_dominated_fronts(pop.get_f())
>>> plt.ylim([0,6])
>>> plt.title("ZDT1: random initial population")
其中每个连续的帕累托前沿以更深的颜色绘制。如果我们现在输入:
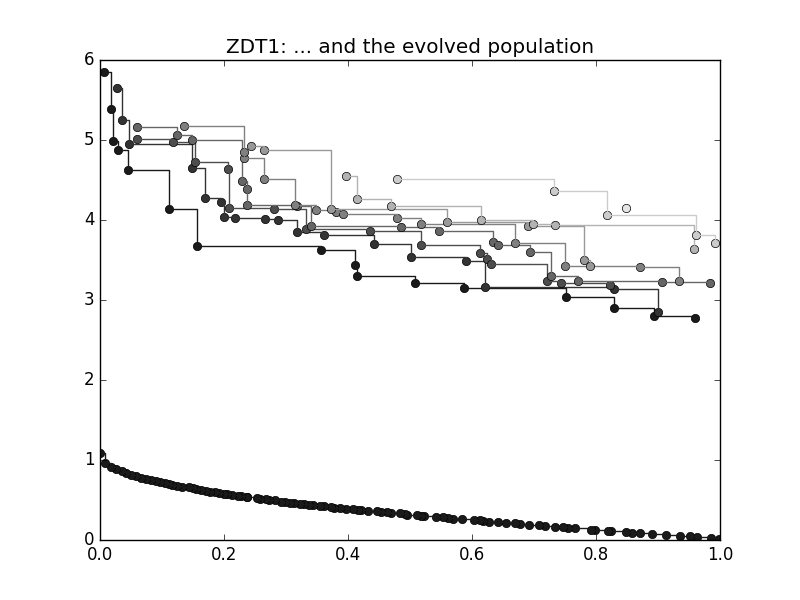
>>> algo = algorithm(moead(gen = 250))
>>> pop = algo.evolve(pop)
>>> ax = plot_non_dominated_fronts(pop.get_f())
>>> plt.title("ZDT1: ... and the evolved population")
我们已经实例化了算法 moead,能够处理多目标问题,并将代数固定为250。在下一行中,我们直接使用算法的方法 pygmo.moead.evolve() 来进化 population
整个种群现在处于一个非支配前沿,可以通过输入以下内容轻松验证:
>>> ndf, dl, dc, ndl = fast_non_dominated_sorting(pop.get_f())
>>> print(ndf)
[array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16,
17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33,
34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50,
51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67,
68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84,
85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99], dtype=uint64)]
pygmo.zdt 问题套件中的问题(以及 pygmo.dtlz 中的问题)实现了一个称为 p_distance 的良好收敛度量。我们可以检查非支配前沿覆盖已知帕累托前沿的程度。
>>> udp.p_distance(pop)
0.03926512747685471
如果我们对某个指标的值不满意,我们可以让种群进化更多代以提高这个数值:
>>> pop = algo.evolve(pop)
>>> udp.p_distance(pop)
0.010346571321103046