使用pygmo的moead#
在本教程中,我们将看到如何使用pygmo提供的用户定义算法moead。特别是我们将使用pygmo中实现的DTLZ套件中的问题作为测试案例,这些问题是作为用户定义问题dtlz实现的。
首先,快速的想法是实例化问题(例如DTLZ1)并使用默认设置运行moead。我们可以使用p_distance()指标来监控整个种群向帕累托前沿的收敛情况。

>>> from pygmo import *
>>> udp = dtlz(prob_id = 1)
>>> pop = population(prob = udp, size = 105)
>>> algo = algorithm(moead(gen = 100))
>>> for i in range(10):
... pop = algo.evolve(pop)
>>> print(udp.p_distance(pop))
11.906892367806368
5.7957743802958595
5.6155823329927355
5.227825963470699
3.3244186681980863
1.6876728522762465
1.2704673513592113
1.01938844212957
0.9181813093367411
0.6759127264898211
由于p_distance()没有捕捉到解决方案的分布信息,我们还使用pygmo类hypervolume计算了超体积指标:
>>> hv = hypervolume(pop)
>>> hv.compute(ref_point = [1.,1.,1.])
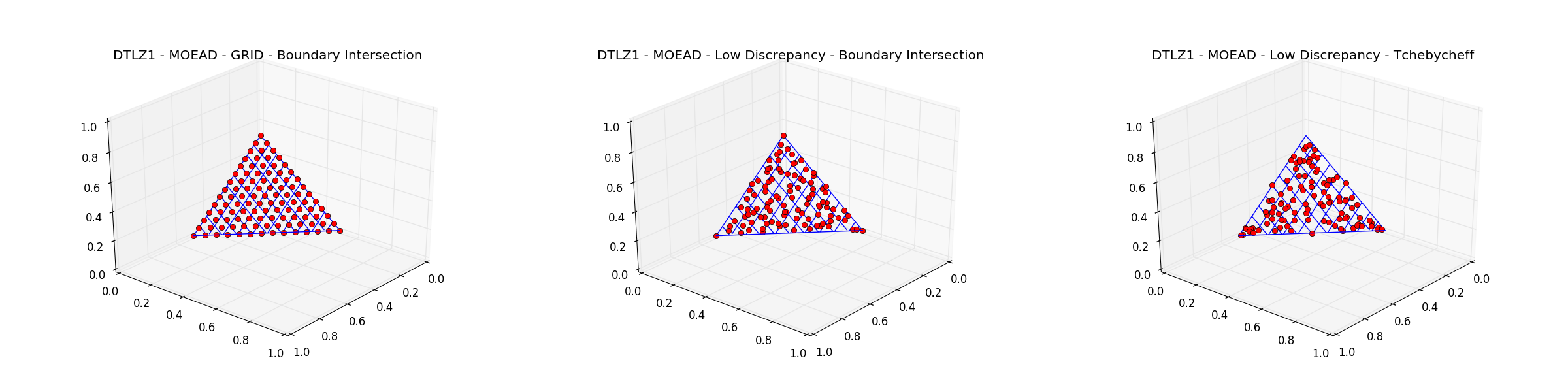
在这种情况下,参考点可以手动设置,因为dtlz1问题是众所周知的。我们还可以将整个种群可视化,因为用户定义的问题dtlz实现了绘图功能:
>>> from matplotlib import pyplot as plt
>>> udp.plot(pop)
>>> plt.title("DTLZ1 - MOEAD - GRID - TCHEBYCHEFF")
我们在获得上述结果时使用了moead的默认参数。特别是weight_generation参数被设置为grid,而decomposition参数被设置为tchebycheff,如下所示:
>>> print(algo)
Algorithm name: MOEAD: MOEA/D - DE [stochastic]
C++ class name: ...
Thread safety: basic
Extra info:
Generations: 100
Weight generation: grid
Decomposition method: tchebycheff
Neighbourhood size: 20
Parameter CR: 1
Parameter F: 0.5
Distribution index: 20
Chance for diversity preservation: 0.9
Seed: ...
Verbosity: 0
weight_generation 方法 grid 提供了分解权重的均匀分布,但它限制了种群大小,因为它只允许根据目标数量确定某些大小。这在使用 moead 进行比较或其他高级设置时可能会显得有限制。在这些情况下,pygmo 提供了替代的权重生成方法。特别是原始的 low discrepancy 方法确保可以生成任意数量的权重,同时确保在目标空间上的低差异分布。
分解方法tchebycheff也可以更改为pygmo实现的边界交叉方法,当适用时,这会导致最终种群在Pareto前沿上的更好分布。使用不同的moead实例重复上述优化,结果如下所示。