通用 API 快速入门#
import arviz as az
import matplotlib.pyplot as plt
import numpy as np
import pymc as pm
import pytensor.tensor as pt
/home/xian/anaconda3/envs/pymc-dev-py39/lib/python3.9/site-packages/pkg_resources/__init__.py:123: PkgResourcesDeprecationWarning: main is an invalid version and will not be supported in a future release
warnings.warn(
RANDOM_SEED = 8927
rng = np.random.default_rng(RANDOM_SEED)
az.style.use("arviz-darkgrid")
1. 模型创建#
PyMC 中的模型围绕 Model 类展开。它包含所有随机变量 (RVs) 的引用,并计算模型的对数概率及其梯度。通常,您会将其作为 with 上下文的一部分进行实例化:
with pm.Model() as model:
# Model definition
pass
我们在下面进一步讨论RVs,但让我们创建一个简单的模型来探索Model类。
model.basic_RVs
[mu ~ N(0, 1), obs ~ N(mu, 1)]
model.free_RVs
[mu ~ N(0, 1)]
model.observed_RVs
[obs ~ N(mu, 1)]
model.compile_logp()({"mu": 0})
array(-143.03962875)
值得强调的是我们在logp中做出的设计选择。如上所示,logp正在被调用并传递参数,因此它是模型实例的一个方法。更准确地说,它根据模型的当前状态或作为logp参数给出的状态组合一个函数(见下面的示例)。
由于多种原因,我们假设一个Model实例不是静态的。如果你需要在内部循环中使用logp并且它需要是静态的,只需使用类似logp = model.logp的方式。下面是一个示例——注意缓存效果和速度提升:
%timeit model.compile_logp()({"mu": 0.1})
logp = model.compile_logp()
%timeit logp({"mu": 0.1})
83 ms ± 7.1 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
18 µs ± 276 ns per loop (mean ± std. dev. of 7 runs, 100,000 loops each)
2. 概率分布#
每个概率程序都由观测到的和未观测到的随机变量(RVs)组成。观测到的随机变量通过似然分布定义,而未观测到的随机变量通过先验分布定义。在PyMC模块中,概率分布的结构如下所示:
pymc:api_distributions_continuous
pymc:api_distributions_discrete
pymc:api_distributions_multivariate
pymc:api_distributions_mixture
pymc:api_distributions_timeseries
pymc:api_distributions_censored
pymc:api_distributions_simulator
未观测的随机变量#
每个未观察到的随机变量都有以下调用签名:名称(字符串),参数关键字参数。因此,可以在模型上下文中这样定义一个正态先验:
与模型一样,我们可以评估其对数概率:
观察到的随机变量#
观测到的RVs与未观测到的RVs定义方式相同,但需要将数据传递到observed关键字参数中:
observed 支持列表、numpy.ndarray 和 pytensor 数据结构。
确定性转换#
PyMC 允许你以各种方式自由地对随机变量进行代数运算:
尽管这些变换可以无缝工作,但它们的结果并不会自动存储。因此,如果你想跟踪一个变换后的变量,你必须使用pm.Deterministic:
with pm.Model():
x = pm.Normal("x", mu=0, sigma=1)
plus_2 = pm.Deterministic("x plus 2", x + 2)
请注意,plus_2 可以以与上述相同的方式使用,我们只是告诉 PyMC 为我们跟踪这个随机变量。
随机变量列表 / 高维随机变量#
上面我们已经看到了如何创建标量随机变量。在许多模型中,我们希望有多个随机变量。用户有时会尝试创建随机变量列表,如下所示:
这可以工作,但它很慢且不推荐使用。相反,我们可以使用坐标:
x 现在是一个长度为3的数组,并且这个数组中的每个变量都与一个标签相关联。这将使我们在查看结果时非常容易区分这3个不同的变量。我们可以对这个数组进行索引或进行线性代数运算:
初始化随机变量#
尽管PyMC会自动初始化模型,但有时为随机变量(RVs)定义初始值是有帮助的。这可以通过initval关键字参数来实现:
{'x': array([0., 0., 0., 0., 0.])}
{'x': array([-0.36012097, -0.16168135, 1.07485641, -0.08855632, -0.03857412])}
当尝试识别模型规范或初始化中的问题时,这种技术有时很有用。
3. 推理#
一旦我们定义了模型,我们必须进行推理以近似后验分布。PyMC 支持两种广泛的推理方法:采样和变分推理。
3.1 采样#
MCMC采样算法的主要入口是通过pm.sample()函数。默认情况下,此函数会尝试自动分配正确的采样器。pm.sample()返回一个arviz.InferenceData对象。InferenceData对象可以轻松地从文件中保存/加载,并且可以携带额外的(元)数据,如日期/版本和后验预测样本。查看ArviZ快速入门以了解更多信息。
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (2 chains in 2 jobs)
NUTS: [mu]
Sampling 2 chains for 1_000 tune and 2_000 draw iterations (2_000 + 4_000 draws total) took 4 seconds.
如您所见,对于仅包含连续变量的模型,PyMC 会分配 NUTS 采样器,即使在复杂模型中也非常高效。PyMC 还会运行初始调整以找到采样器的好起点参数。这里我们从后验中抽取 2000 个样本,并允许采样器在额外的 1500 次迭代中调整其参数。
如果没有通过 chains 参数设置,链的数量将根据可用 CPU 核心的数量来确定。
idata.posterior.dims
Frozen({'chain': 2, 'draw': 2000})
默认情况下,调优样本会被丢弃。通过设置discard_tuned_samples=False,它们可以被保留,并最终出现在InferenceData对象中的一个单独的组中(即,idata.warmup_posterior)。
您可以使用 chains 和 cores 参数来控制链的并行运行方式:
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (6 chains in 4 jobs)
NUTS: [mu]
Sampling 6 chains for 1_000 tune and 1_000 draw iterations (6_000 + 6_000 draws total) took 7 seconds.
idata.posterior["mu"].shape
(6, 1000)
# get values of a single chain
idata.posterior["mu"].sel(chain=2).shape
(1000,)
3.2 分析采样结果#
分析采样结果时最常用的图是所谓的轨迹图:
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (2 chains in 2 jobs)
NUTS: [mu, sd]
Sampling 2 chains for 1_000 tune and 1_000 draw iterations (2_000 + 2_000 draws total) took 4 seconds.
az.plot_trace(idata);
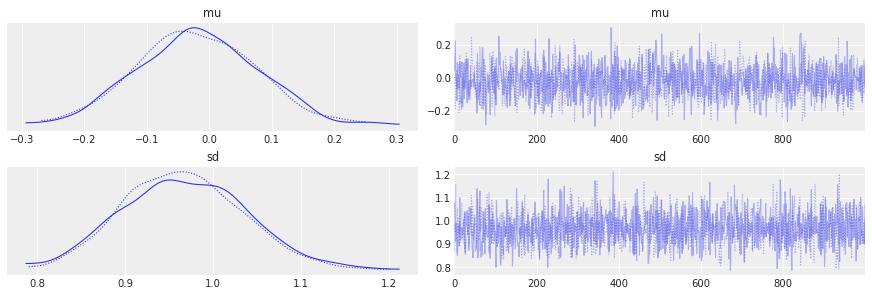
另一个常见的指标是Gelman-Rubin统计量,或R-hat:
az.summary(idata)
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| mu | -0.019 | 0.096 | -0.193 | 0.163 | 0.002 | 0.002 | 1608.0 | 1343.0 | 1.0 |
| sd | 0.967 | 0.069 | 0.835 | 1.089 | 0.002 | 0.001 | 1836.0 | 1406.0 | 1.0 |
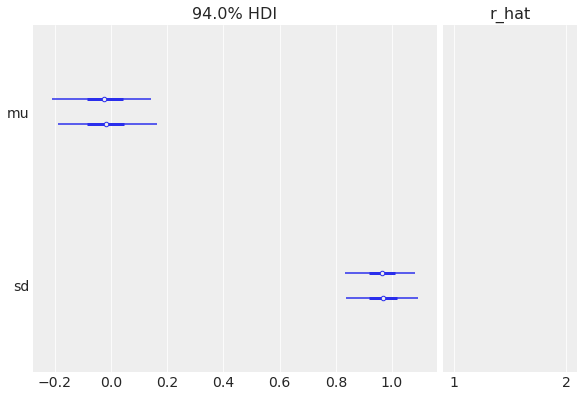
R-hat 也作为 az.plot_forest 的一部分呈现:
az.plot_forest(idata, r_hat=True);
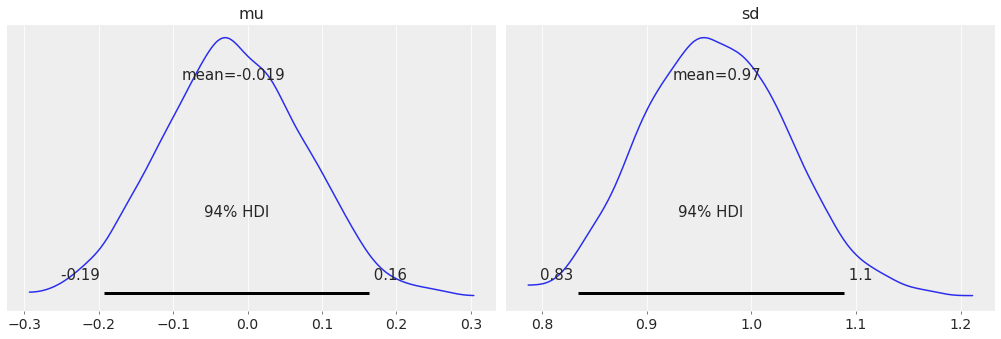
最后,对于受[Kruschke, 2014]启发的后验图,您可以使用:
az.plot_posterior(idata);
对于高维模型,查看所有参数的轨迹变得繁琐。当使用NUTS时,我们可以查看能量图来评估收敛问题:
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (2 chains in 2 jobs)
NUTS: [x]
Sampling 2 chains for 1_000 tune and 1_000 draw iterations (2_000 + 2_000 draws total) took 5 seconds.
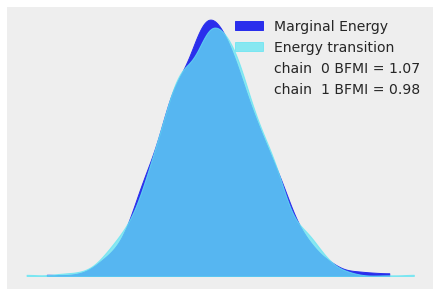
有关采样器统计信息和能量图的更多信息,请参阅采样器统计信息。有关识别采样问题及其解决方法的更多信息,请参阅使用分歧诊断偏差推理。
3.3 变分推断#
PyMC 支持各种变分推断技术。虽然这些方法速度更快,但它们通常也准确性较低,并且可能导致有偏的推断。主要的入口点是 pymc.fit()。
Finished [100%]: Average Loss = 142
返回的 Approximation 对象具有多种功能,例如从近似后验中抽取样本,我们可以像常规采样运行一样对其进行分析:
idata = approx.sample(1000)
az.summary(idata)
arviz - WARNING - Shape validation failed: input_shape: (1, 1000), minimum_shape: (chains=2, draws=4)
| mean | sd | hdi_3% | hdi_97% | mcse_mean | mcse_sd | ess_bulk | ess_tail | r_hat | |
|---|---|---|---|---|---|---|---|---|---|
| mu | -0.023 | 0.169 | -0.338 | 0.296 | 0.005 | 0.004 | 973.0 | 880.0 | NaN |
| sd | 0.989 | 0.158 | 0.694 | 1.262 | 0.005 | 0.004 | 972.0 | 1026.0 | NaN |
The variational 子模块提供了很大的灵活性,可以选择使用哪种变分推断(VI),并遵循面向对象的设计。例如,全秩ADVI估计一个完整的协方差矩阵:
Finished [100%]: Average Loss = 0.012772
使用面向对象接口的等效表达式是:
with pm.Model(coords={"idx": np.arange(2)}) as model:
pm.MvNormal("x", mu=mu, cov=cov, dims="idx")
approx = pm.FullRankADVI().fit()
Finished [100%]: Average Loss = 0.020531
with pm.Model(coords={"idx": np.arange(2)}) as model:
pm.MvNormal("x", mu=mu, cov=cov, dims="idx")
approx = pm.FullRankADVI().fit()
Finished [100%]: Average Loss = 0.014143
plt.figure()
idata = approx.sample(10000)
az.plot_pair(idata, var_names="x", coords={"idx": [0, 1]});
<Figure size 432x288 with 0 Axes>
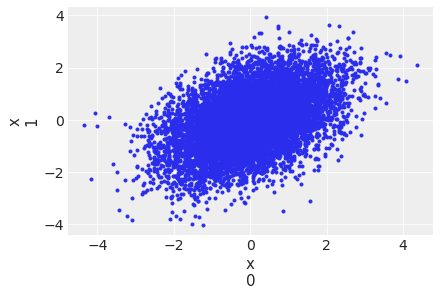
Stein变分梯度下降(SVGD)使用粒子来估计后验分布:
with pm.Model() as model:
pm.NormalMixture("x", w=[0.2, 0.8], mu=[-0.3, 0.5], sigma=[0.1, 0.1])
plt.figure()
idata = approx.sample(10000)
az.plot_dist(idata.posterior["x"]);
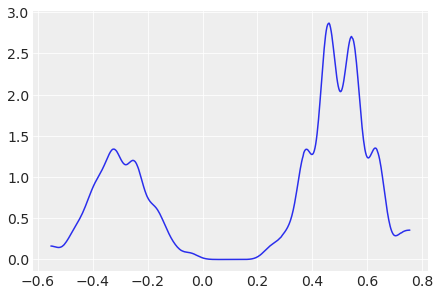
有关变分推断的更多信息,请参阅变分推断。
4. 后验预测采样#
函数 sample_posterior_predictive() 对保留数据进行预测并执行后验预测检查。
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (2 chains in 2 jobs)
NUTS: [mu, sd]
Sampling 2 chains for 1_000 tune and 1_000 draw iterations (2_000 + 2_000 draws total) took 4 seconds.
with model:
idata.extend(pm.sample_posterior_predictive(idata))
fig, ax = plt.subplots()
az.plot_ppc(idata, ax=ax)
ax.axvline(data.mean(), ls="--", color="r", label="True mean")
ax.legend(fontsize=10);
/home/xian/anaconda3/envs/pymc-dev-py39/lib/python3.9/site-packages/IPython/core/pylabtools.py:151: UserWarning: Creating legend with loc="best" can be slow with large amounts of data.
fig.canvas.print_figure(bytes_io, **kw)
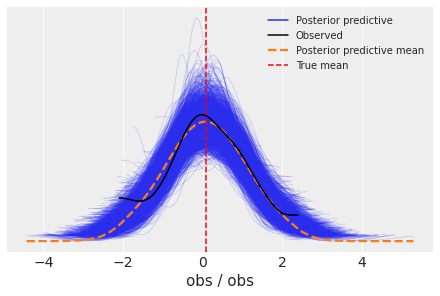
4.1 对保留数据的预测#
在许多情况下,您希望对未见过的/保留的数据进行预测。这在概率机器学习和贝叶斯深度学习中尤为重要。PyMC 包含一个 pm.MutableData 容器,以帮助实现这些用途。它是一个围绕 pytensor.shared 变量的包装器,并允许稍后更改数据的值。否则,pm.MutableData 对象可以像任何其他 numpy 数组或张量一样使用。
这一区别非常重要,因为PyMC中的所有模型在内部都是巨大的符号表达式。当你直接将原始数据传递到模型中时,你实际上是允许PyTensor将这些数据视为常量,并在这样做有意义的情况下对其进行优化。如果你以后需要更改这些数据,你可能无法在更大的符号表达式中指向它。使用pm.MutableData提供了一种在符号表达式中指向特定位置并更改其内容的方法。
x = rng.standard_normal(100)
y = x > 0
coords = {"idx": np.arange(100)}
with pm.Model() as model:
# create shared variables that can be changed later on
x_obs = pm.MutableData("x_obs", x, dims="idx")
y_obs = pm.MutableData("y_obs", y, dims="idx")
coeff = pm.Normal("x", mu=0, sigma=1)
logistic = pm.math.sigmoid(coeff * x_obs)
pm.Bernoulli("obs", p=logistic, observed=y_obs, dims="idx")
idata = pm.sample()
Auto-assigning NUTS sampler...
Initializing NUTS using jitter+adapt_diag...
Multiprocess sampling (2 chains in 2 jobs)
NUTS: [x]
Sampling 2 chains for 1_000 tune and 1_000 draw iterations (2_000 + 2_000 draws total) took 3 seconds.
现在假设我们要对未见过的数据进行预测。为此,我们需要更改 x_obs 和 y_obs 的值。理论上我们不需要设置 y_obs,因为我们想要预测它,但它必须与 x_obs 的形状相匹配。
with model:
# change the value and shape of the data
pm.set_data(
{
"x_obs": [-1, 0, 1.0],
# use dummy values with the same shape:
"y_obs": [0, 0, 0],
},
coords={"idx": [1001, 1002, 1003]},
)
idata.extend(pm.sample_posterior_predictive(idata))
idata.posterior_predictive["obs"].mean(dim=["draw", "chain"])
<xarray.DataArray 'obs' (idx: 3)> array([0.0215, 0.488 , 0.982 ]) Coordinates: * idx (idx) int64 1001 1002 1003
参考资料#
约翰·克鲁施克。贝叶斯数据分析:R、JAGS和Stan的教程。学术出版社,2014年。
%load_ext watermark
%watermark -n -u -v -iv -w -p pytensor,aeppl,xarray
Last updated: Fri Jun 03 2022
Python implementation: CPython
Python version : 3.9.13
IPython version : 8.4.0
pytensor: 2.6.2
aeppl : 0.0.31
xarray: 2022.3.0
arviz : 0.12.1
numpy : 1.22.4
pymc : 4.0.0b6
pytensor : 2.6.2
matplotlib: 3.5.2
Watermark: 2.3.1