Note
Go to the end to download the full example code. or to run this example in your browser via Binder
特征缩放的重要性#
通过标准化进行特征缩放,也称为Z分数归一化,是许多机器学习算法的重要预处理步骤。它涉及重新缩放每个特征,使其具有标准差为1且均值为0。
即使基于树的模型(几乎)不受缩放的影响,许多其他算法也需要对特征进行归一化,原因各不相同:为了加速收敛(如非惩罚的逻辑回归),为了创建与未缩放数据相比完全不同的模型拟合(如K近邻模型)。后者在本示例的第一部分中进行了演示。
在示例的第二部分中,我们展示了特征归一化如何影响主成分分析(PCA)。为此,我们比较了在未缩放数据上使用:class:~sklearn.decomposition.PCA 找到的主成分与使用:class:~sklearn.preprocessing.StandardScaler 先缩放数据后找到的主成分。
在示例的最后一部分中,我们展示了归一化对在PCA降维数据上训练的模型准确性的影响。
# 作者:scikit-learn 开发者
# SPDX-License-Identifier: BSD-3-Clause
加载和准备数据#
所使用的数据集是UCI提供的 Wine recognition dataset 。该数据集具有连续特征,由于所测量的属性不同(例如酒精含量和苹果酸),这些特征在尺度上是异质的。
from sklearn.datasets import load_wine
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
X, y = load_wine(return_X_y=True, as_frame=True)
scaler = StandardScaler().set_output(transform="pandas")
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.30, random_state=42
)
scaled_X_train = scaler.fit_transform(X_train)
k-邻近模型的重新缩放效果#
为了可视化 KNeighborsClassifier 的决策边界,在本节中我们选择了一个具有不同数量级值的2个特征的子集。
请注意,使用特征子集来训练模型可能会遗漏具有高预测影响的特征,从而导致决策边界相比于使用全特征集训练的模型要差得多。
import matplotlib.pyplot as plt
from sklearn.inspection import DecisionBoundaryDisplay
from sklearn.neighbors import KNeighborsClassifier
X_plot = X[["proline", "hue"]]
X_plot_scaled = scaler.fit_transform(X_plot)
clf = KNeighborsClassifier(n_neighbors=20)
def fit_and_plot_model(X_plot, y, clf, ax):
clf.fit(X_plot, y)
disp = DecisionBoundaryDisplay.from_estimator(
clf,
X_plot,
response_method="predict",
alpha=0.5,
ax=ax,
)
disp.ax_.scatter(X_plot["proline"], X_plot["hue"], c=y, s=20, edgecolor="k")
disp.ax_.set_xlim((X_plot["proline"].min(), X_plot["proline"].max()))
disp.ax_.set_ylim((X_plot["hue"].min(), X_plot["hue"].max()))
return disp.ax_
fig, (ax1, ax2) = plt.subplots(ncols=2, figsize=(12, 6))
fit_and_plot_model(X_plot, y, clf, ax1)
ax1.set_title("KNN without scaling")
fit_and_plot_model(X_plot_scaled, y, clf, ax2)
ax2.set_xlabel("scaled proline")
ax2.set_ylabel("scaled hue")
_ = ax2.set_title("KNN with scaling")
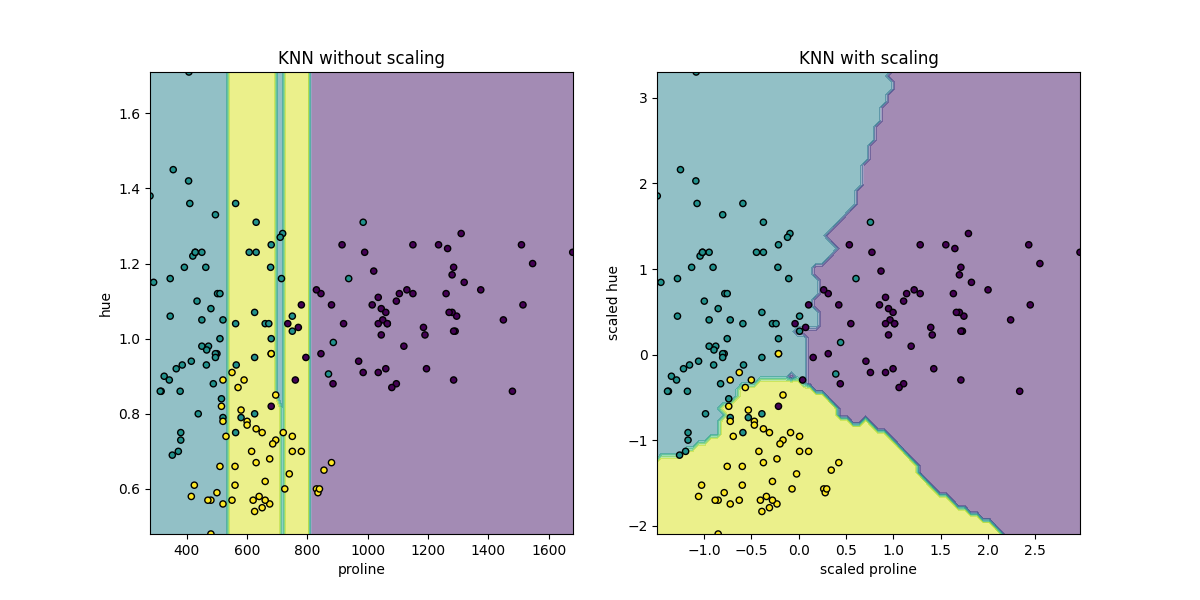
这里的决策边界显示了拟合经过缩放和未缩放的数据会导致完全不同的模型。原因在于变量“proline”的取值范围在0到1000之间,而变量“hue”的取值范围在1到10之间。因此,样本之间的距离主要受“proline”值的差异影响,而“hue”值则相对被忽略。如果使用:class:~sklearn.preprocessing.StandardScaler 来标准化这个数据库,两个缩放后的值大约在-3到3之间,邻居结构将会受到两个变量的等量影响。
重新缩放对PCA降维的影响#
使用 PCA 进行降维包括找到使方差最大的特征。如果一个特征仅因为其尺度不同而比其他特征变化更大,PCA 会认为该特征主导了主成分的方向。
我们可以使用所有原始特征来检查第一个主成分:
import pandas as pd
from sklearn.decomposition import PCA
pca = PCA(n_components=2).fit(X_train)
scaled_pca = PCA(n_components=2).fit(scaled_X_train)
X_train_transformed = pca.transform(X_train)
X_train_std_transformed = scaled_pca.transform(scaled_X_train)
first_pca_component = pd.DataFrame(
pca.components_[0], index=X.columns, columns=["without scaling"]
)
first_pca_component["with scaling"] = scaled_pca.components_[0]
first_pca_component.plot.bar(
title="Weights of the first principal component", figsize=(6, 8)
)
_ = plt.tight_layout()
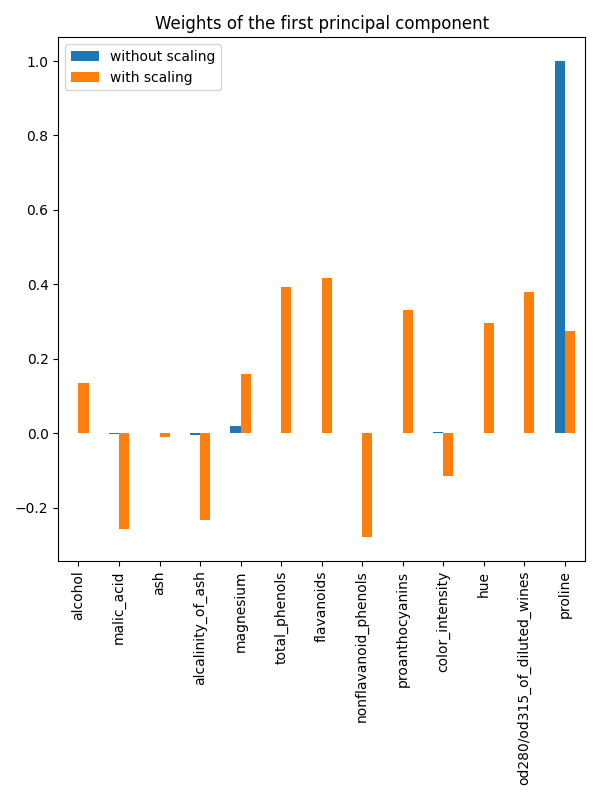
确实,我们发现“脯氨酸”特征在未缩放的情况下主导了第一个主成分的方向,其量级大约比其他特征高两个数量级。相比之下,观察缩放版本数据的第一个主成分时,所有特征的数量级大致相同。
我们可以在两种情况下可视化主成分的分布:
fig, (ax1, ax2) = plt.subplots(nrows=1, ncols=2, figsize=(10, 5))
target_classes = range(0, 3)
colors = ("blue", "red", "green")
markers = ("^", "s", "o")
for target_class, color, marker in zip(target_classes, colors, markers):
ax1.scatter(
x=X_train_transformed[y_train == target_class, 0],
y=X_train_transformed[y_train == target_class, 1],
color=color,
label=f"class {target_class}",
alpha=0.5,
marker=marker,
)
ax2.scatter(
x=X_train_std_transformed[y_train == target_class, 0],
y=X_train_std_transformed[y_train == target_class, 1],
color=color,
label=f"class {target_class}",
alpha=0.5,
marker=marker,
)
ax1.set_title("Unscaled training dataset after PCA")
ax2.set_title("Standardized training dataset after PCA")
for ax in (ax1, ax2):
ax.set_xlabel("1st principal component")
ax.set_ylabel("2nd principal component")
ax.legend(loc="upper right")
ax.grid()
_ = plt.tight_layout()
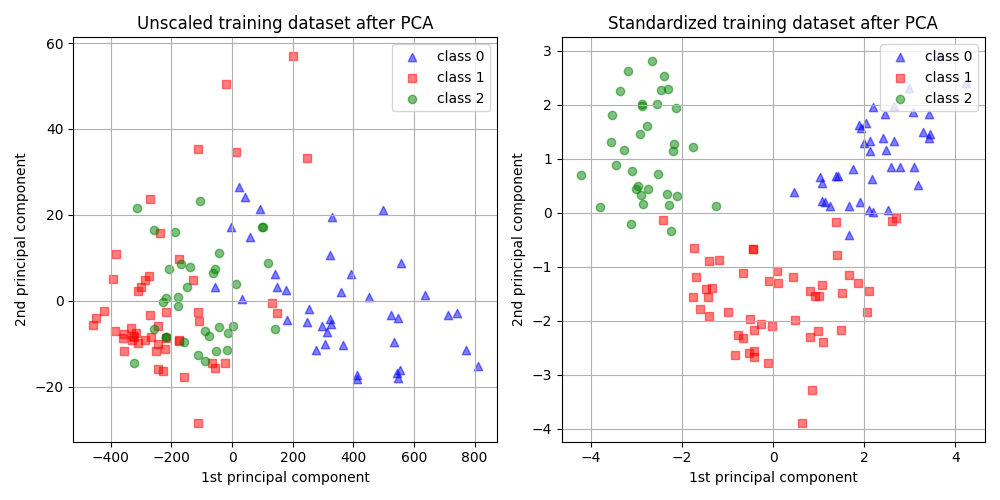
从上图中我们可以观察到,在降维之前对特征进行缩放会使得各个成分具有相同的数量级。在这种情况下,它还提高了类别的可分性。事实上,在下一节中我们确认了更好的可分性对整体模型性能有良好的影响。
对模型性能的重缩放效果#
首先,我们展示了数据的缩放或非缩放如何影响
LogisticRegressionCV 的最优正则化:
import numpy as np
from sklearn.linear_model import LogisticRegressionCV
from sklearn.pipeline import make_pipeline
Cs = np.logspace(-5, 5, 20)
unscaled_clf = make_pipeline(pca, LogisticRegressionCV(Cs=Cs))
unscaled_clf.fit(X_train, y_train)
scaled_clf = make_pipeline(scaler, pca, LogisticRegressionCV(Cs=Cs))
scaled_clf.fit(X_train, y_train)
print(f"Optimal C for the unscaled PCA: {unscaled_clf[-1].C_[0]:.4f}\n")
print(f"Optimal C for the standardized data with PCA: {scaled_clf[-1].C_[0]:.2f}")
Optimal C for the unscaled PCA: 0.0004
Optimal C for the standardized data with PCA: 20.69
对于在应用PCA之前未进行缩放的数据,正则化的需求更高( C 值较低)。我们现在评估缩放对最优模型的准确性和平均对数损失的影响:
from sklearn.metrics import accuracy_score, log_loss
y_pred = unscaled_clf.predict(X_test)
y_pred_scaled = scaled_clf.predict(X_test)
y_proba = unscaled_clf.predict_proba(X_test)
y_proba_scaled = scaled_clf.predict_proba(X_test)
print("Test accuracy for the unscaled PCA")
print(f"{accuracy_score(y_test, y_pred):.2%}\n")
print("Test accuracy for the standardized data with PCA")
print(f"{accuracy_score(y_test, y_pred_scaled):.2%}\n")
print("Log-loss for the unscaled PCA")
print(f"{log_loss(y_test, y_proba):.3}\n")
print("Log-loss for the standardized data with PCA")
print(f"{log_loss(y_test, y_proba_scaled):.3}")
Test accuracy for the unscaled PCA
35.19%
Test accuracy for the standardized data with PCA
96.30%
Log-loss for the unscaled PCA
0.957
Log-loss for the standardized data with PCA
0.0825
在数据进行 PCA 之前进行缩放时,预测准确性有明显差异,缩放后的版本远远优于未缩放的版本。这与前一节中的图表直观感受相一致,在使用 PCA 之前进行缩放时,各成分变得线性可分。
请注意,在这种情况下,使用缩放特征的模型比使用未缩放特征的模型表现更好,因为所有变量都被期望是有预测性的,我们宁愿避免其中一些变量被相对忽略。
如果较低尺度的变量没有预测性,缩放特征后可能会导致性能下降:噪声特征在缩放后会对预测贡献更多,因此缩放会增加过拟合。
最后,我们观察到,通过缩放步骤可以实现更低的对数损失。
Total running time of the script: (0 minutes 0.767 seconds)
Related examples